FFPE单细胞数据处理案例与优化建议
时长: 3 分钟
字数: 659 字
更新: 2025-09-02
阅读: 0 次
案例背景
- 样本类型:胃癌FFPE(甲醛固定石蜡包埋)组织
- 问题描述:原始单细胞表达矩阵直接整合聚类效果不理想,cluster分群不明显,细胞类型注释困难,存在较多背景RNA干扰。
数据处理与优化流程
1. 预处理与质量控制
- 对每个样本的表达矩阵进行初步质控。
- 采用 SoupX 去除背景RNA污染。
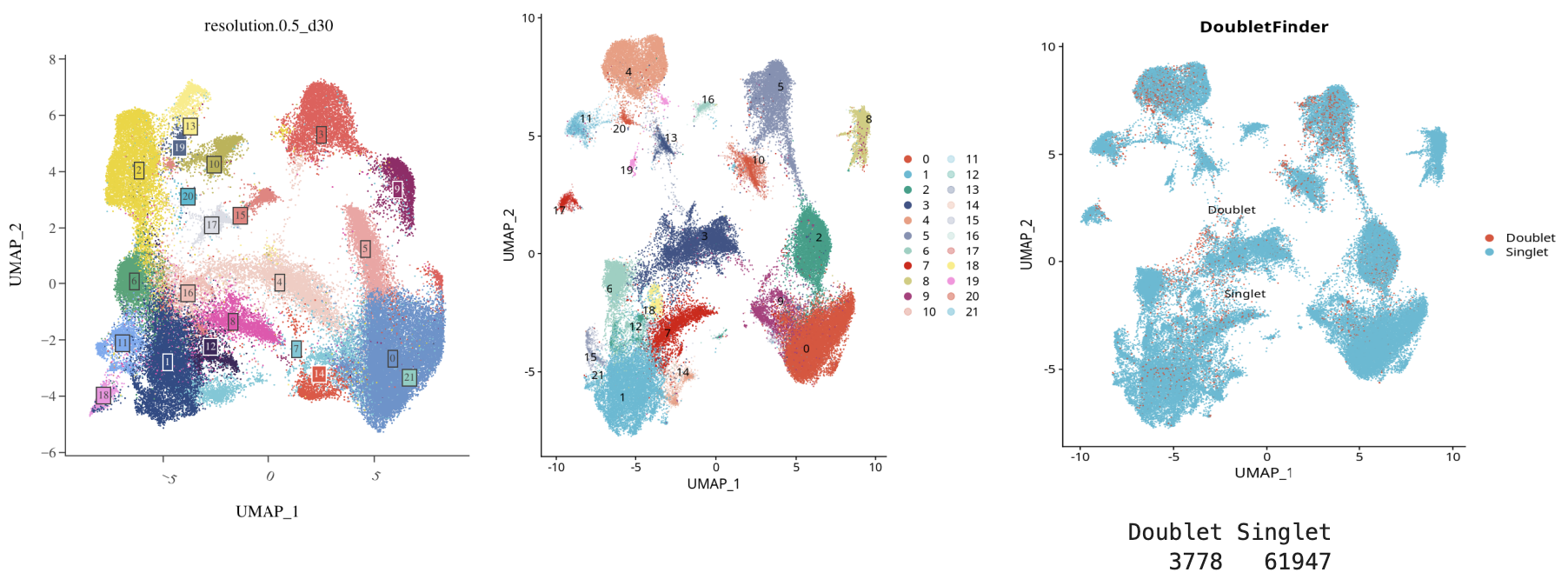
- 使用 DoubletFinder 过滤双细胞(doublets),过滤阈值:
- UMI数:500-75,000
- 基因数:200-7,500
- 线粒体基因比例:<10%
TIP
FFPE样本由于RNA降解和背景污染严重,建议务必进行背景去除和双细胞过滤。
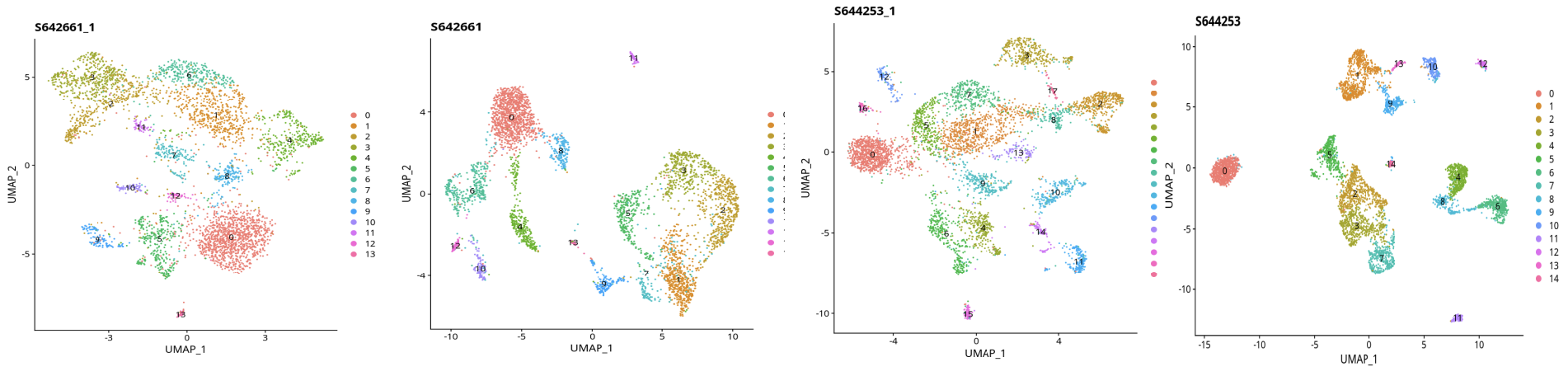
2. 聚类与注释效果对比
- 处理前:聚类分群不明显,细胞类型难以准确注释。
- 处理后:
- SoupX去背景后,cluster边界清晰,分群改善明显。
- DoubletFinder进一步去除双细胞,提升聚类纯度。
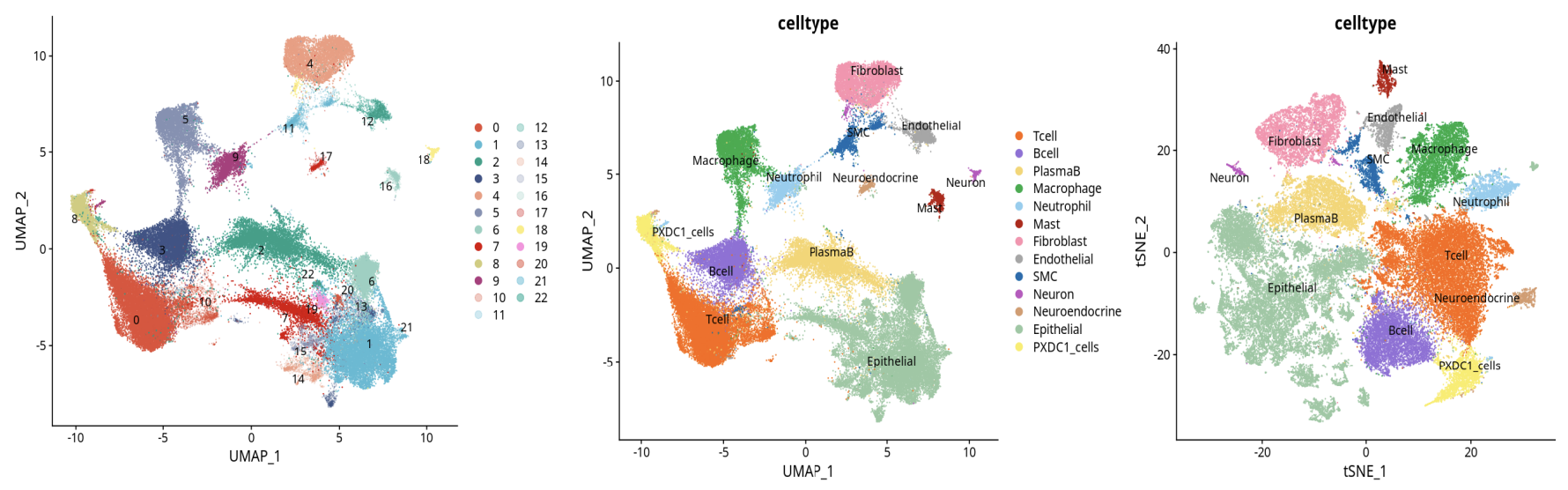
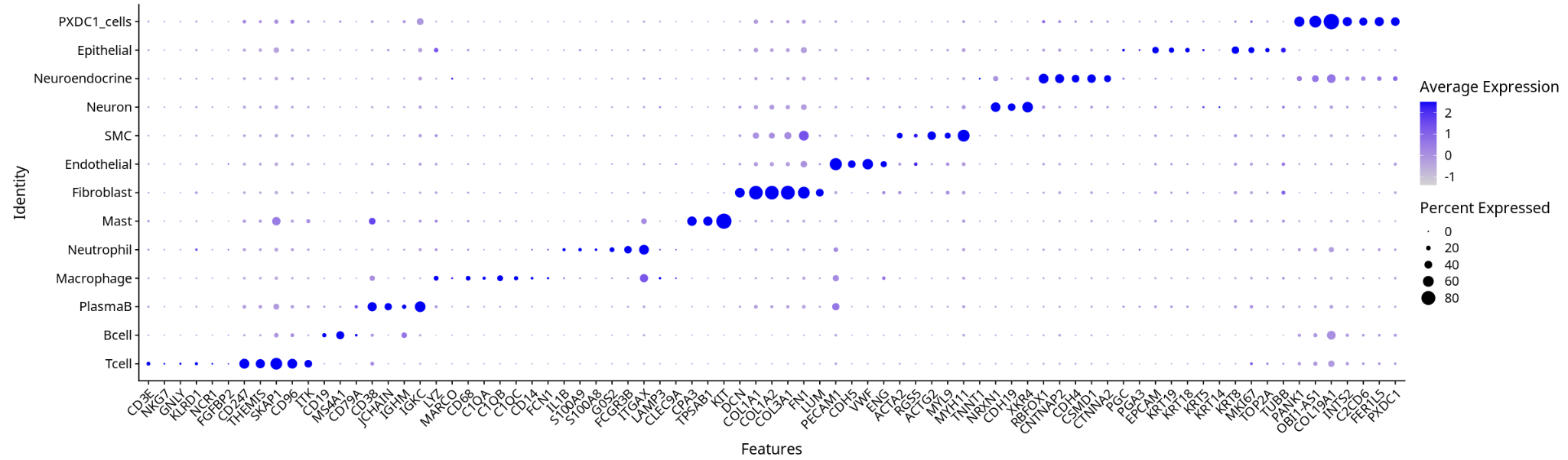
- 细胞类型注释和marker基因表达分布更符合生物学预期。
图注:左为原始聚类,右为SoupX处理后聚类
图注:左为原始聚类,中为SoupX处理,右为DoubletFinder去除双细胞后,双细胞比例约6%
3. 细胞类型注释与marker基因表达
4. 经验总结与优化建议
IMPORTANT
FFPE样本常见问题包括RNA降解严重、总量低、片段短、细胞核完整性差、有核率低(20-30%)、背景RNA多,导致分群弥散。
- 数据处理建议:
- 必须进行背景RNA去除(如SoupX)
- 过滤双细胞(如DoubletFinder)
- 严格质控(UMI、基因数、线粒体比例)
- 特征选择高变基因,优化聚类参数(如PCA数、分辨率)
- 批次整合建议用Harmony等方法优化参数
- 聚类后结合marker基因手动调整亚群,去除混合群体
NOTE
聚类和注释效果的提升依赖于多环节优化,建议结合样本特性灵活调整参数。
结论
本案例展示了针对FFPE及其它RNA表达文库样本,聚类分群不理想时的优化处理流程。通过背景去除、双细胞过滤和参数优化,可显著提升聚类和注释效果。该流程可作为类似问题的参考和建议,帮助用户提升FFPE及复杂样本的单细胞分析质量。