外源 GFP 未检出排查:GTF 注释修复与定量示例
时长: 3 分钟
字数: 528 字
更新: 2026-01-21
阅读: 0 次
问题描述
在分析数据时,参考基因组中已构建外源插入基因 GFP,但在云平台分析结果中找不到 GFP 基因的表达。
排查过程
- 初步确认:
- 表达矩阵中存在 GFP 基因,但 RDS 文件中未包含 GFP 基因,导致云平台分析无对应基因。
- 样本验证:
- 以某样品为例,BAM 文件可比对到 GFP 基因,且比对结果均为 150M。
- 但
XS标签均为XS:Z:Unassigned_NoFeatures。 - 说明 GFP 基因可被比对,但在 featureCounts 定量时未被识别。
shell
zcat features.tsv.gz | grep -n GFP
zcat matrix.mtx.gz | awk -v gene_row=索引 '$1 == gene_row { count++ } END { print count }'
samtools view 某样品_SortedByCoordinate_withTag.bam | grep "GFP"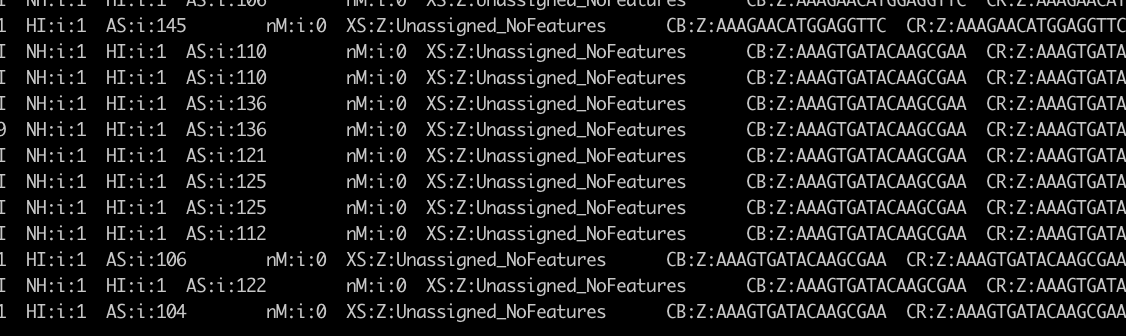
- 多样本复查:
- 其他样本同样存在定量未识别 GFP 基因的问题。
原因分析
IMPORTANT
GFP基因在featureCounts定量未被识别,主要原因是GTF文件格式不规范:外源基因GFP的注释缺少gene和transcript行,仅有exon信息,导致featureCounts无法正确定量。
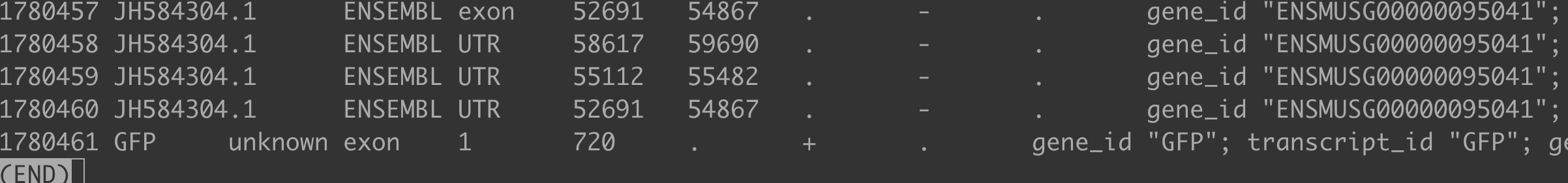
解决办法
TIP
需重新构建包含 GFP 基因的 GTF 注释文件,确保包含 gene、transcript 和 exon 三类注释行。
标准 GFP 基因 GTF 格式示例:
text
# 添加gene行
echo -e 'GFP\tunknown\tgene\t1\t720\t.\t+\t.\tgene_id "GFP"; gene_name "GFP"; gene_biotype "protein_coding";' > GFP.gtf
# 添加transcript行 (使用 >> 追加)
echo -e 'GFP\tunknown\ttranscript\t1\t720\t.\t+\t.\tgene_id "GFP"; transcript_id "GFP"; gene_name "GFP"; gene_biotype "protein_coding";' >> GFP.gtf
# 添加exon行 (使用 >> 追加)
echo -e 'GFP\tunknown\texon\t1\t720\t.\t+\t.\tgene_id "GFP"; transcript_id "GFP"; gene_name "GFP"; gene_biotype "protein_coding"; exon_number 1; exon_id "GFP"' >> GFP.gtf
NOTE
使用修正后的 GTF 文件重新构建参考基因组并进行定量分析,GFP 基因即可被正常识别和定量。
