寻因产品表面蛋白 Antibody 和 CRISPR 质控分析说明
时长: 4 分钟
字数: 883 字
更新: 2025-09-02
阅读: 0 次
本流程适用于 DD 表面蛋白与 CRISPR 数据的质控与初步分析。
WARNING
目前 SeekSoulTools 暂不直接支持这两类文库的原生分析。需先将数据转为 10x 格式后,使用 Cell Ranger 完成分析。请参考 10x 官方教程获取更详细的说明与结果解读。
一、表面蛋白 Antibody
1. 与转录组、VDJ 文库联合分析
- fastp 数据质控(可选)
- 数据转化:
- 先转化 5' 数据,形成
map.txt(SeekOne barcode 与 10x barcode 的对应关系)。 - 再转化表面蛋白与 VDJ 数据,参考已转化成功的 5' 数据
map.txt中的 barcode 对应关系。 - 转化工具详见
Barcode-Converter.zip,压缩包中包含详细使用说明。
- 先转化 5' 数据,形成
- Cell Ranger multi 分析
- 执行脚本:
bash
cellranger multi \
--id= sample \
--csv=multi-config-template-7.1-antigen.csv \
--localcores 8 \
--localmem 64- 说明:
multi-config-template-7.1-antigen.csv:配置文件。- feature 文件:捕获的表面蛋白 feature 文件,由客户提供,格式如下:
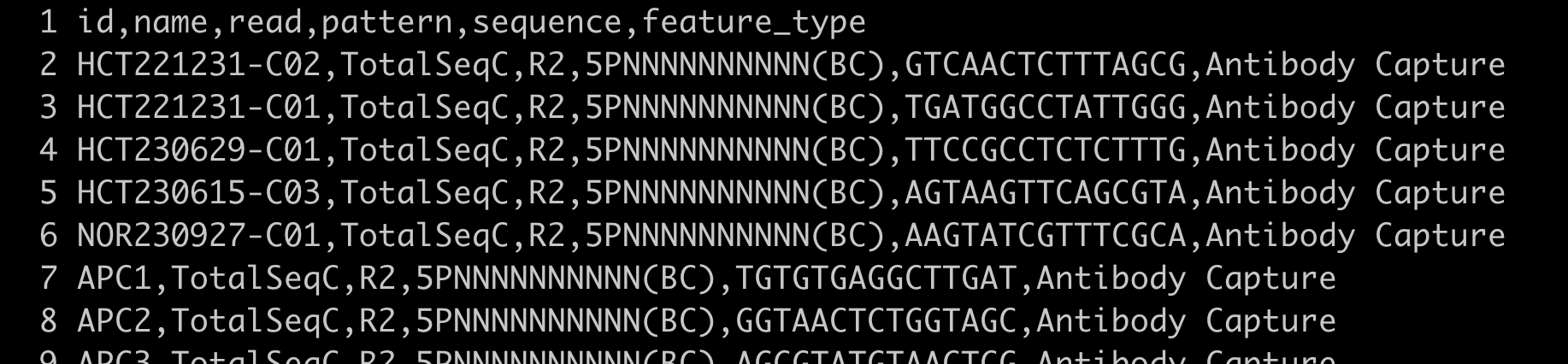
2. 表面蛋白文库单独 count 分析
- fastp 数据质控(可选)
- 将表面蛋白数据直接转化为 10x 格式。
- Cell Ranger count 分析
- 执行脚本:
bash
cellranger count \
--id= Sample \
--libraries=library.csv \
--transcriptome=/PROJ2/DATA/database/refdata-gex-GRCh38-2020-A \
--feature-ref=change_Reference.csv \
--localcores 8 \
--localmem 64- 说明:
library.csv:配置文件。- feature 文件:捕获的表面蛋白 feature 文件,由客户提供,格式与联合分析一致。
二、CRISPR
1. 与转录组文库联合分析
- fastp 数据质控(可选)
- 数据转化:
- 先转化 5' 数据,形成
map.txt(SeekOne barcode 与 10x barcode 的对应关系)。 - 再转化 CRISPR 数据,参考已转化成功的 5' 数据
map.txt中的 barcode 对应关系。 - 转化工具详见
Barcode-Converter.zip,压缩包中包含详细使用说明。
- 先转化 5' 数据,形成
- Cell Ranger 分析参数:
bash
cellranger count \
--id=G1_1 \
--libraries=G1_1.library.csv \
--transcriptome=feature.ref.final_change_final_rev_new_pattern.csv \
--localcores=8 \
--localmem=64说明:
G1_1.library.csv:配置文件,可同时包含转录组、VDJ、CRISPR 数据。feature-ref:捕获的 gene feature 序列,参考 10x 官网准备(格式如下)。pattern:捕获的 feature 识别序列,位于 feature 序列的上游。feature_type:客户提供的基因捕获序列的反向互补序列。

text
library.csv 文件示例:
fastqs,sample,library_type
data/5data/,G1_1_5,Gene Expression
data/crispr_data/,G1_1_crispr,CRISPR Guide Capture2. 与转录组、VDJ 文库联合分析
- fastp 数据质控(可选)
- 数据转化:
- 先转化 5' 数据,形成
map.txt(SeekOne barcode 与 10x barcode 的对应关系)。 - 再转化 CRISPR 与 VDJ 数据,参考已转化成功的 5' 数据
map.txt中的 barcode 对应关系。 - 转化工具详见
Barcode-Converter.zip,压缩包中包含详细使用说明。
- 先转化 5' 数据,形成
- Cell Ranger multi 分析:
bash
cellranger multi \
--id=G1_5 \
--csv=multi-config.csv \
--localcores 8text
multi-config.csv 配置文件格式如下:
[gene-expression]
reference,/database/refdata-gex-GRCh38-2020-A
include-introns,true
[feature]
reference,crispr_Reference.csv
[vdj]
reference,database/refdata-cellranger-vdj-GRCh38-alts-ensembl-5.0.0
[libraries]
fastq_id,fastqs,lanes,feature_types,subsample_rate
G1_5_5,CRISPR/demo/data/,,Gene Expression,1,
G1_5_crispr,CRISPR/demo/data/,,CRISPR Guide Capture,1,
G1_5_BCR,CRISPR/demo/data/,,VDJ-B,1,text
crispr_Reference.csv CRISPR feature 配置文件格式如下:
TIP
转化与分析前,请核对 map.txt 映射关系与 feature 参考文件是否匹配;转化完成后可先抽样进行小规模验证再跑全量数据。
