SeekArc × 10x 跨平台单细胞转录组整合与批次效应评估 (Monocle2 / hdWGCNA)
摘要
本文档展示了 SeekArc 平台与 10x Genomics 平台单细胞转录组数据的整合分析效果,并且通过 Monocle2 和 hdWGCNA 两种主流下游分析流程验证了数据整合的可行性和分析结果的可靠性。结果表明,SeekArc 平台数据与 10x 平台数据具有高度一致性,能够有效支持跨平台单细胞转录组研究的深入开展。
研究背景
在单细胞转录组研究中,不同平台间的数据整合是扩大样本规模、提高统计功效的重要手段。SeekArc 作为新一代单细胞 ATAC 一胞双组转录组测序平台,其与主流平台 10x Genomics 的数据兼容性及整合效果需要系统验证。
数据整合策略
数据预处理
- 原始数据矩阵:包含多个样本的单细胞转录组表达数据。
- 标准化处理:采用 Seurat 标准流程进行数据标准化和质控。
- 批次信息标注:明确标记不同平台和样本来源。
整合方法
采用 Seurat 的整合流程,通过识别锚点 (anchors) 实现跨平台数据的有效对齐。
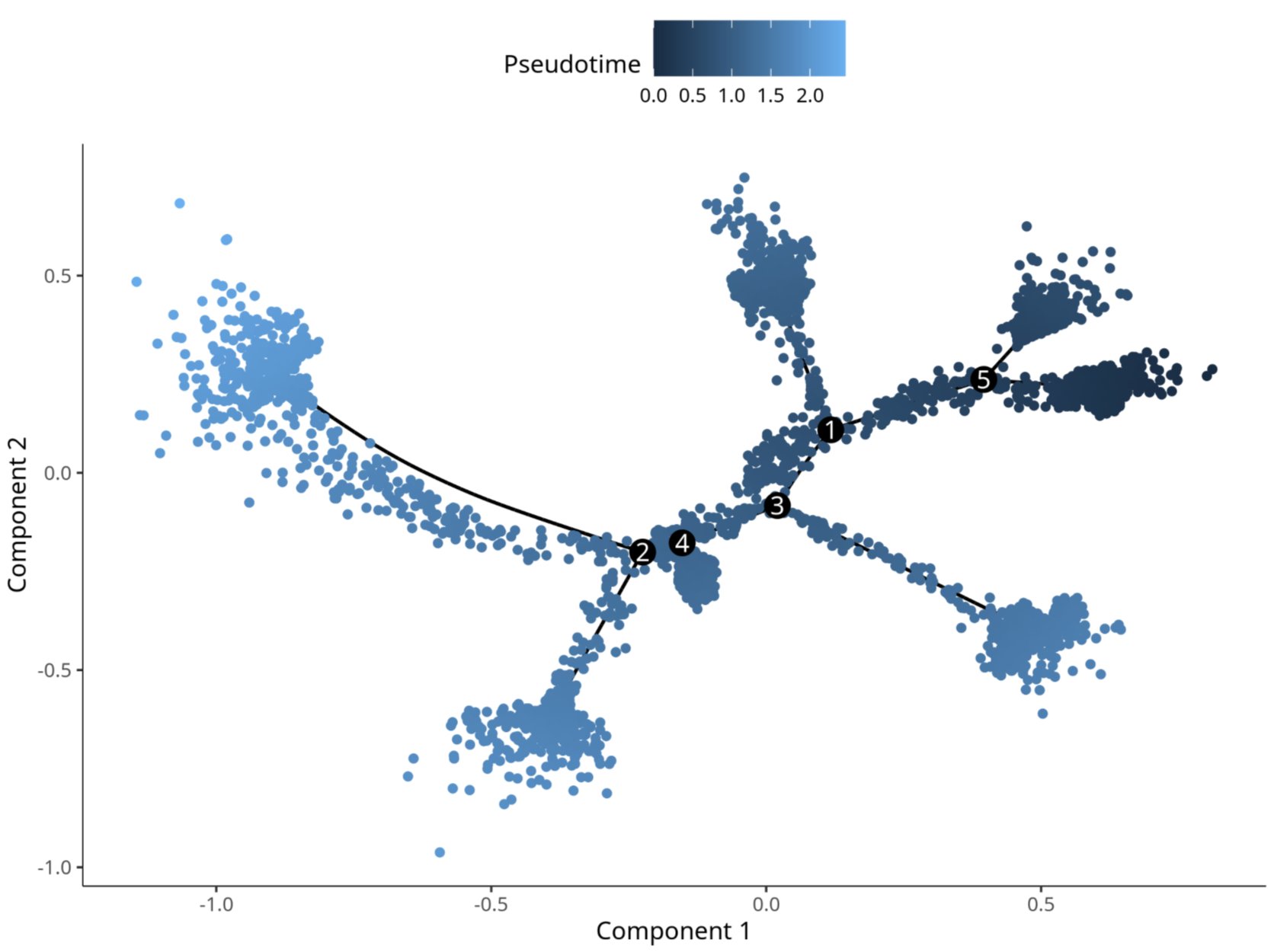
Monocle2 轨迹分析
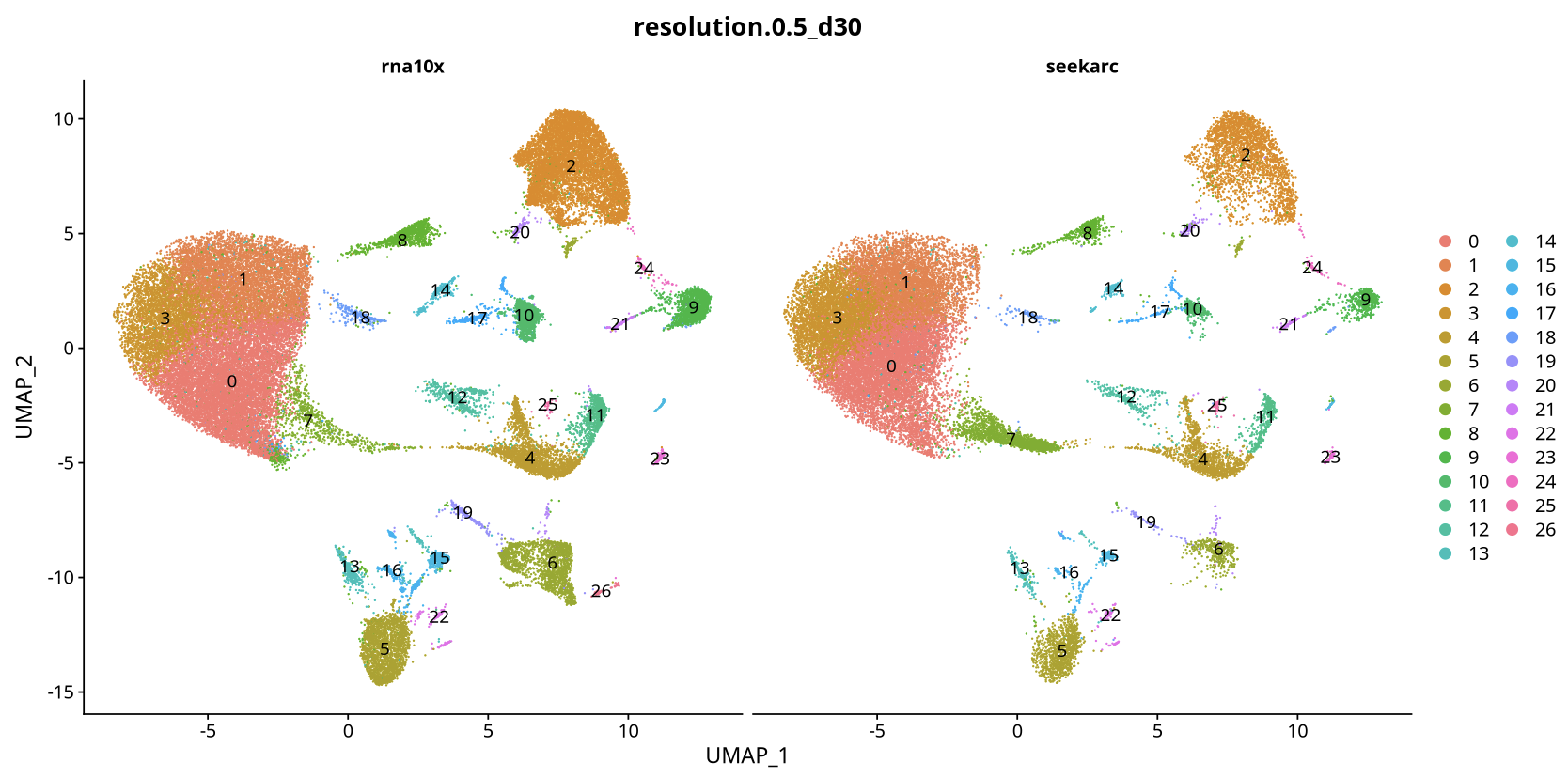
数据矩阵选择对比
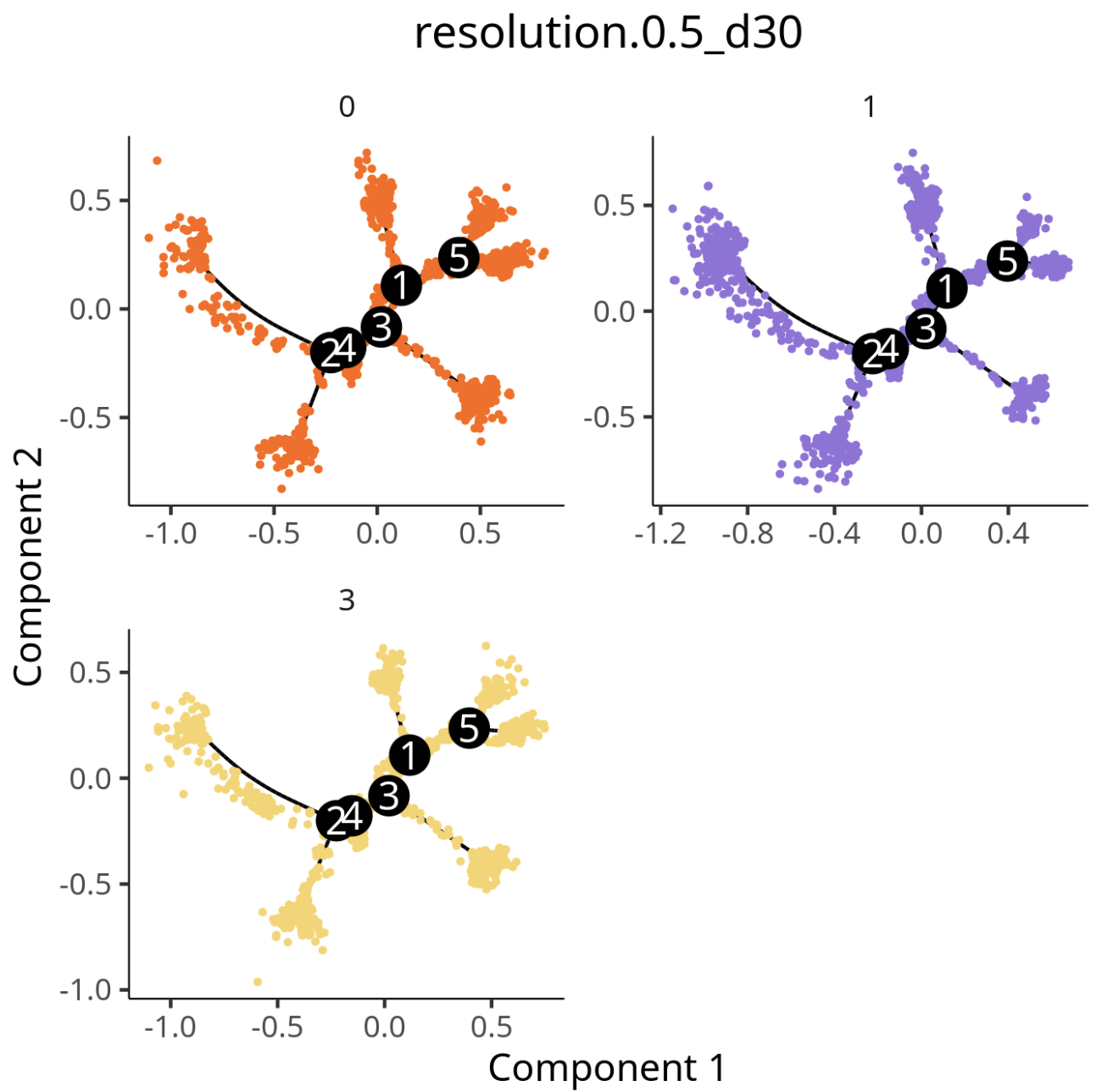
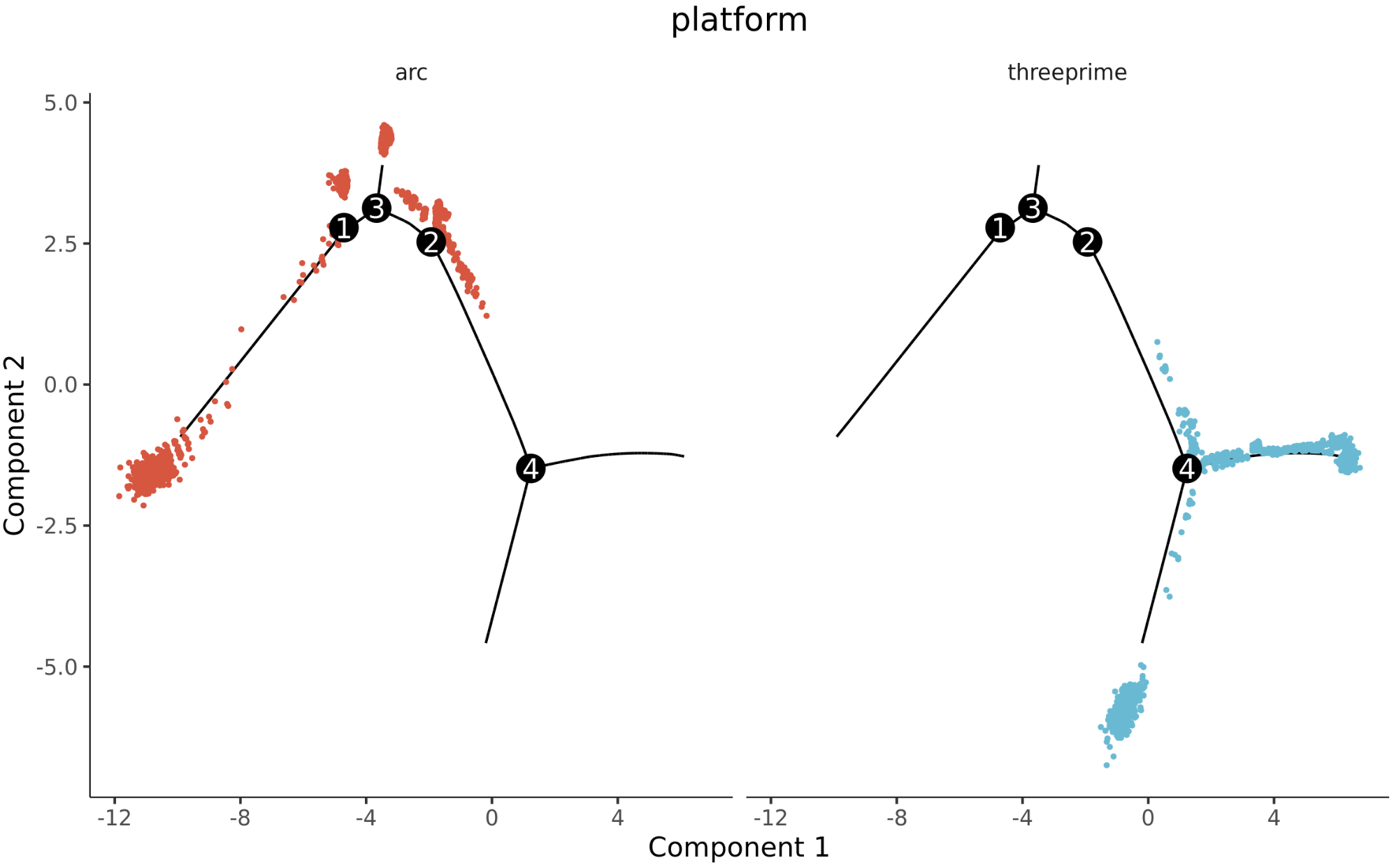
data 矩阵分析结果
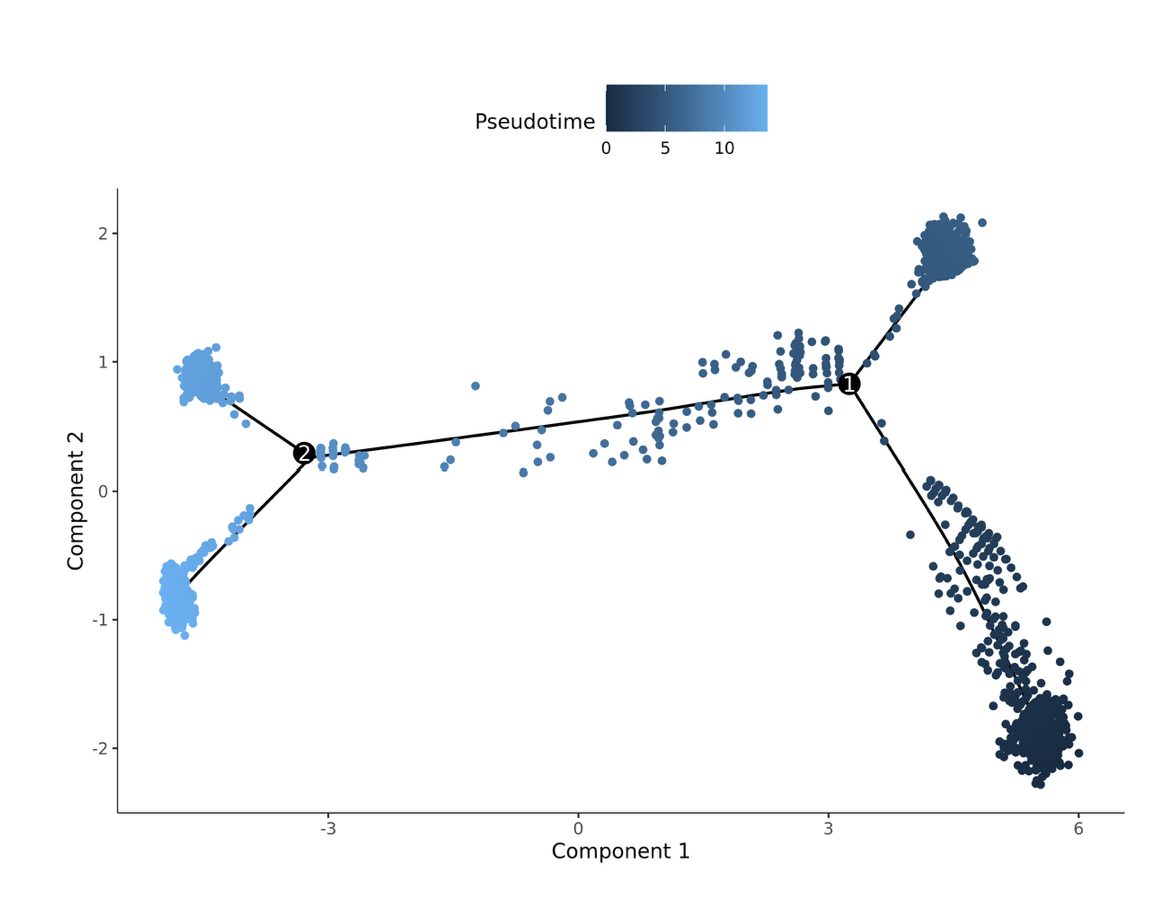
Pseudotime 的细胞发育轨迹图,Pseudotime 是 Monocle2 基于细胞表达信息计算的概率,表示时间的先后。每个点是一个细胞,每个细胞由深到浅,表示时间的顺序。
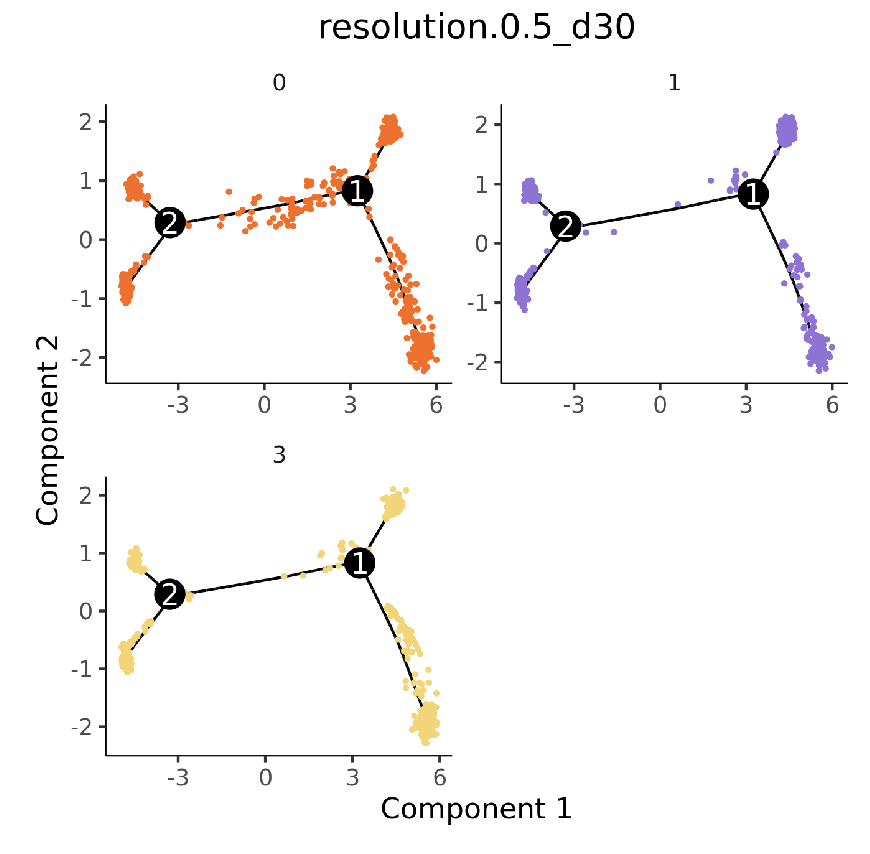
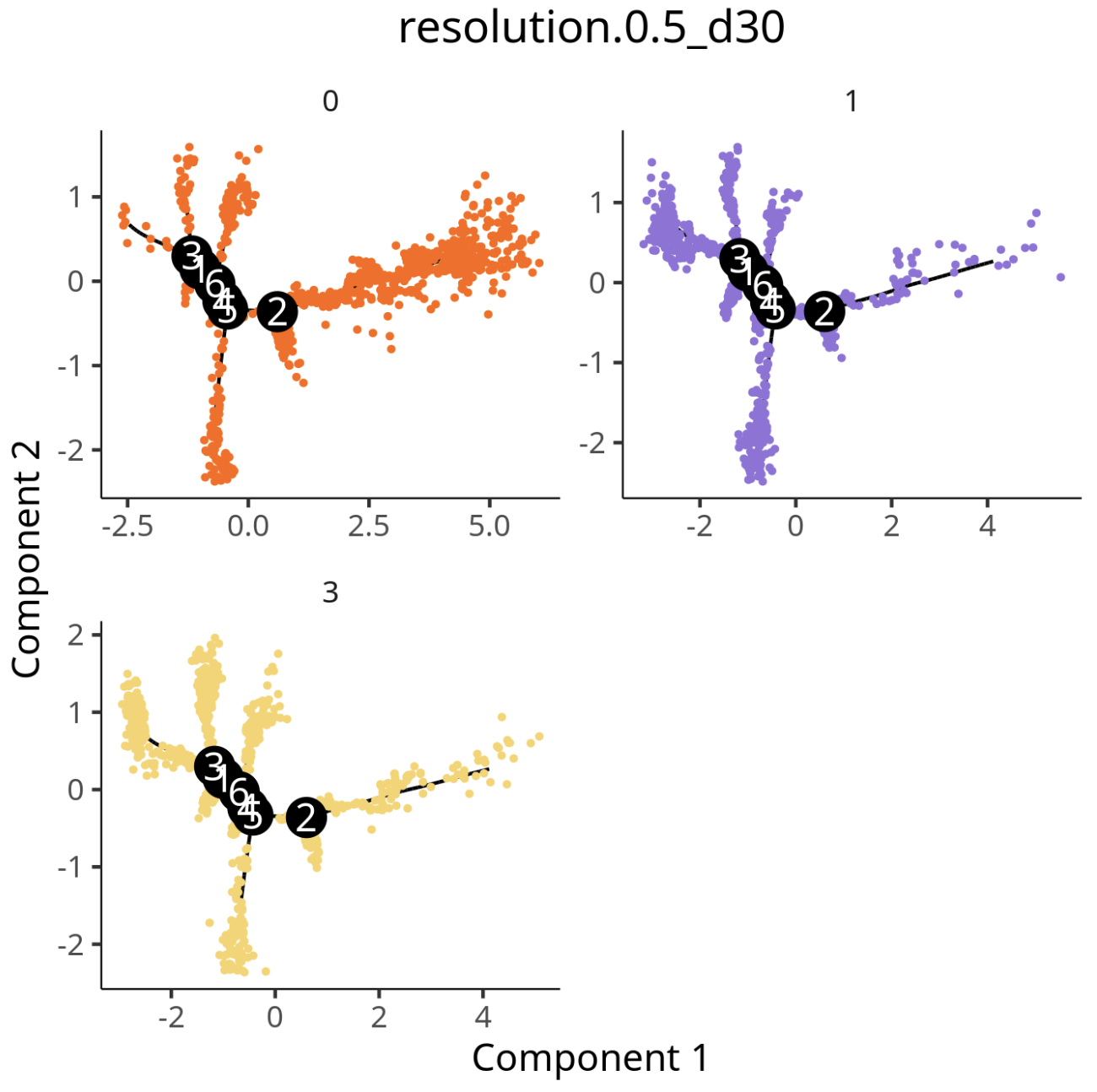
细胞群轨迹发育图,每个点代表一个细胞,不同颜色代表不同细胞群。
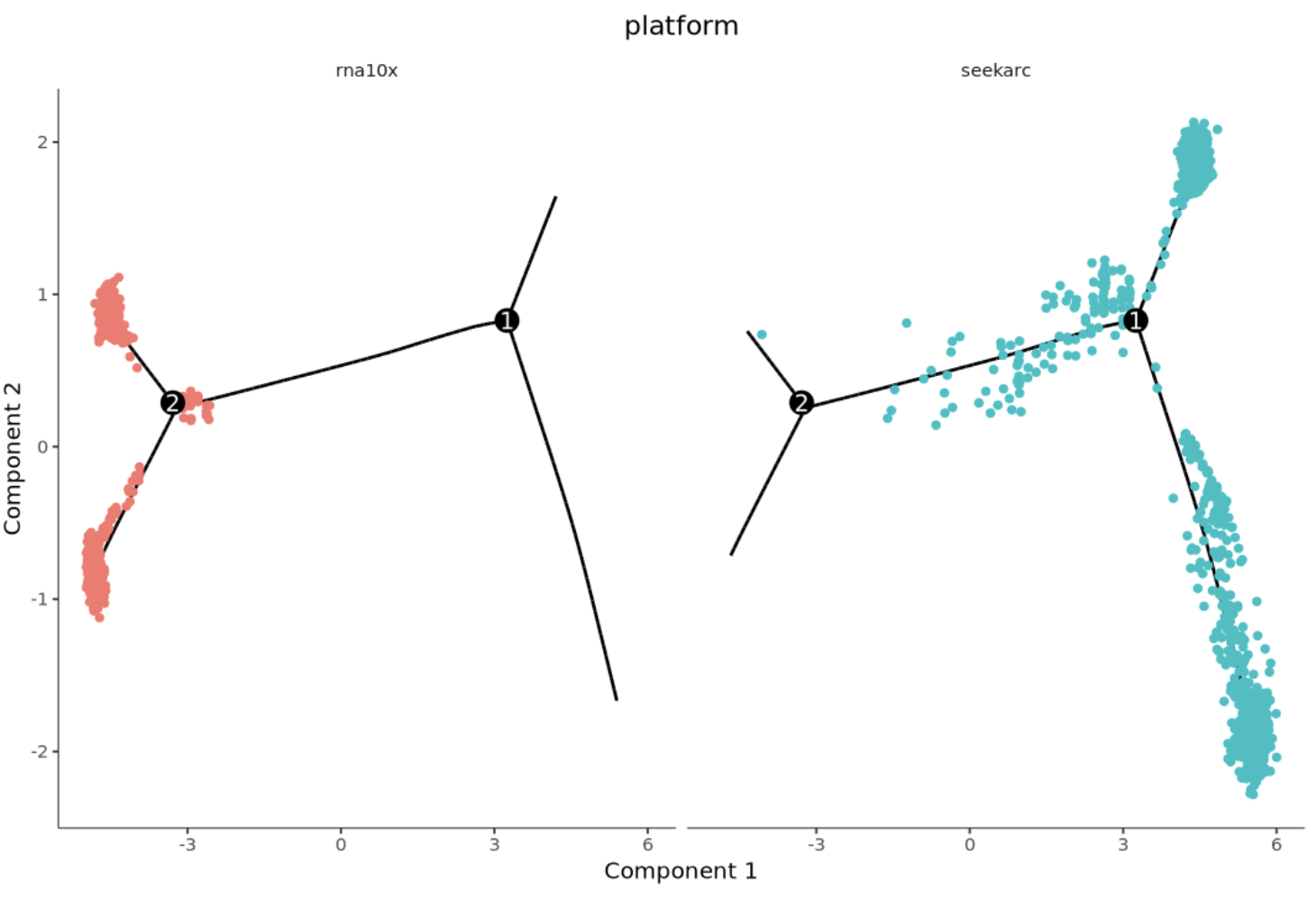
不同平台的细胞轨迹图,每个点代表一个细胞,不同颜色代表不同平台。
counts 矩阵分析结果
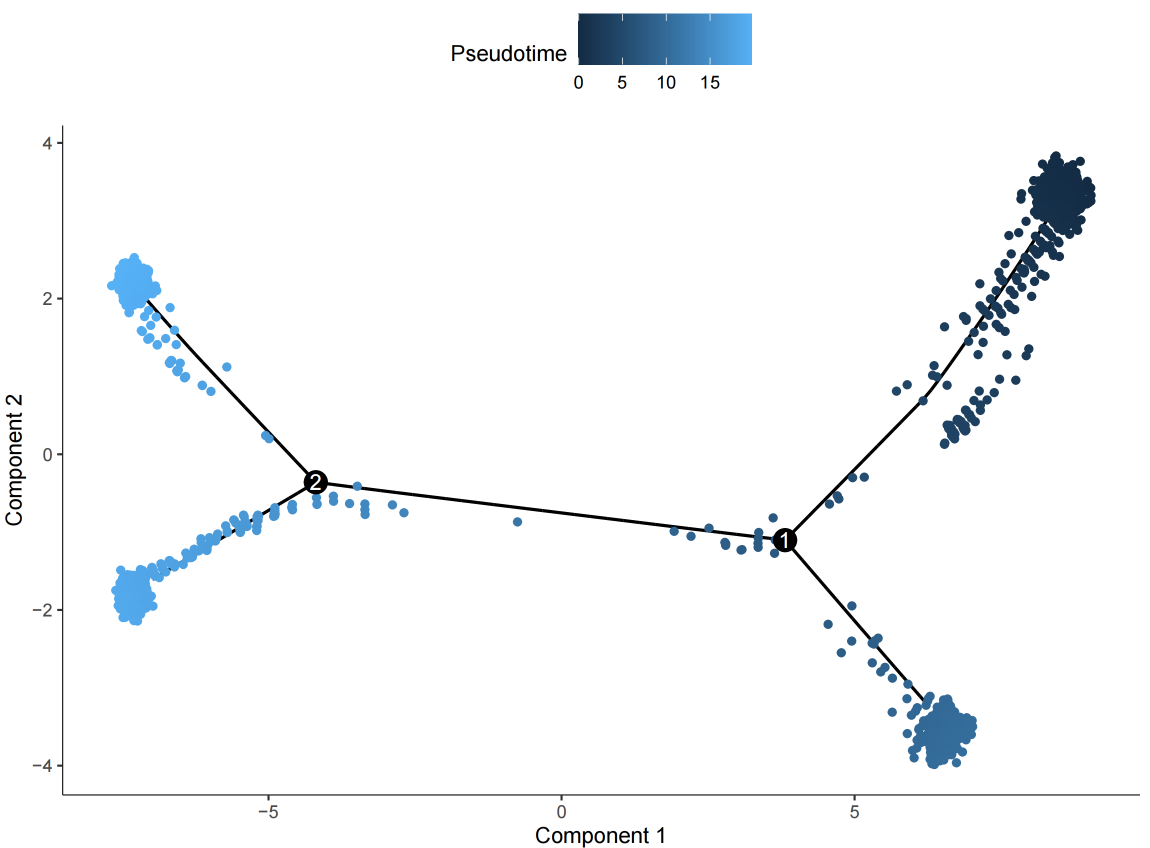
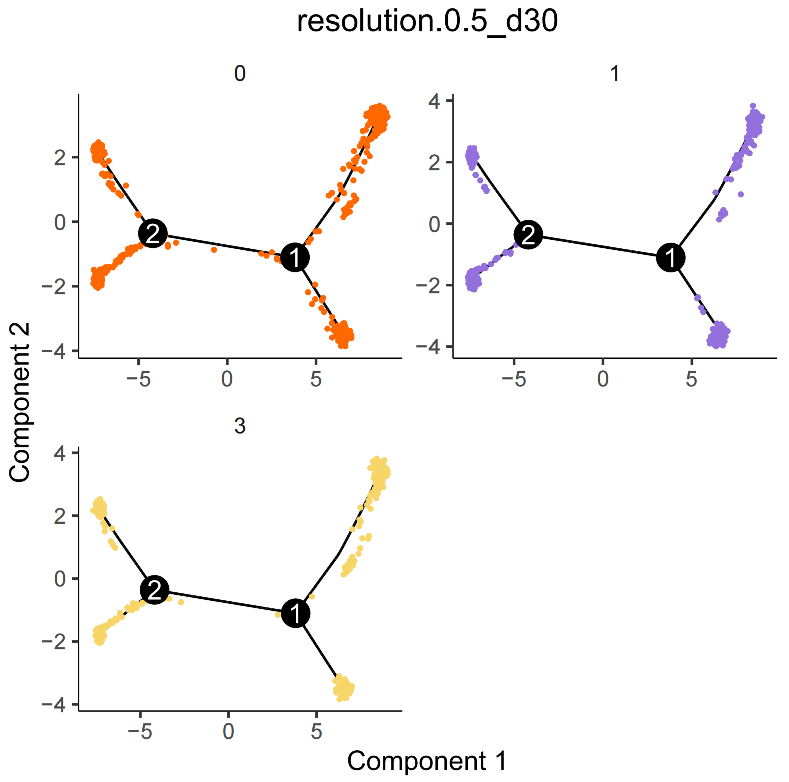
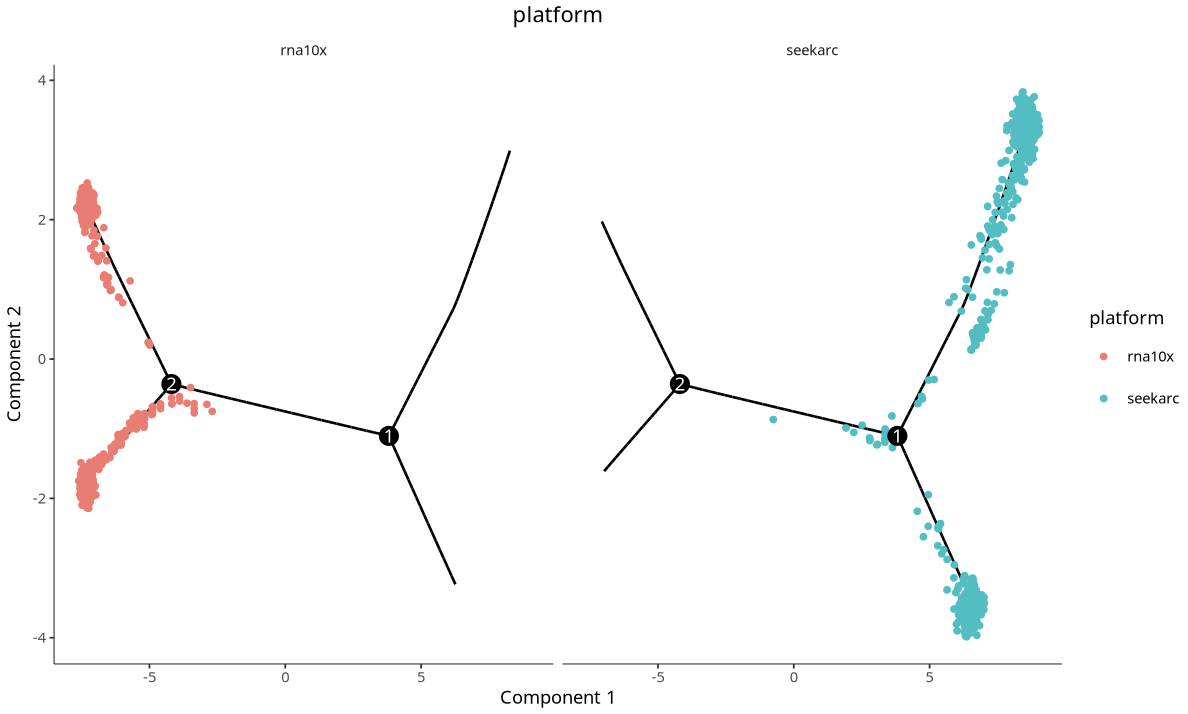
批次效应校正策略
策略 A:使用 integrated 矩阵
策略 B:添加批次校正参数
# 在reduceDimension中添加批次校正
residualModelFormulaStr = "~Sample"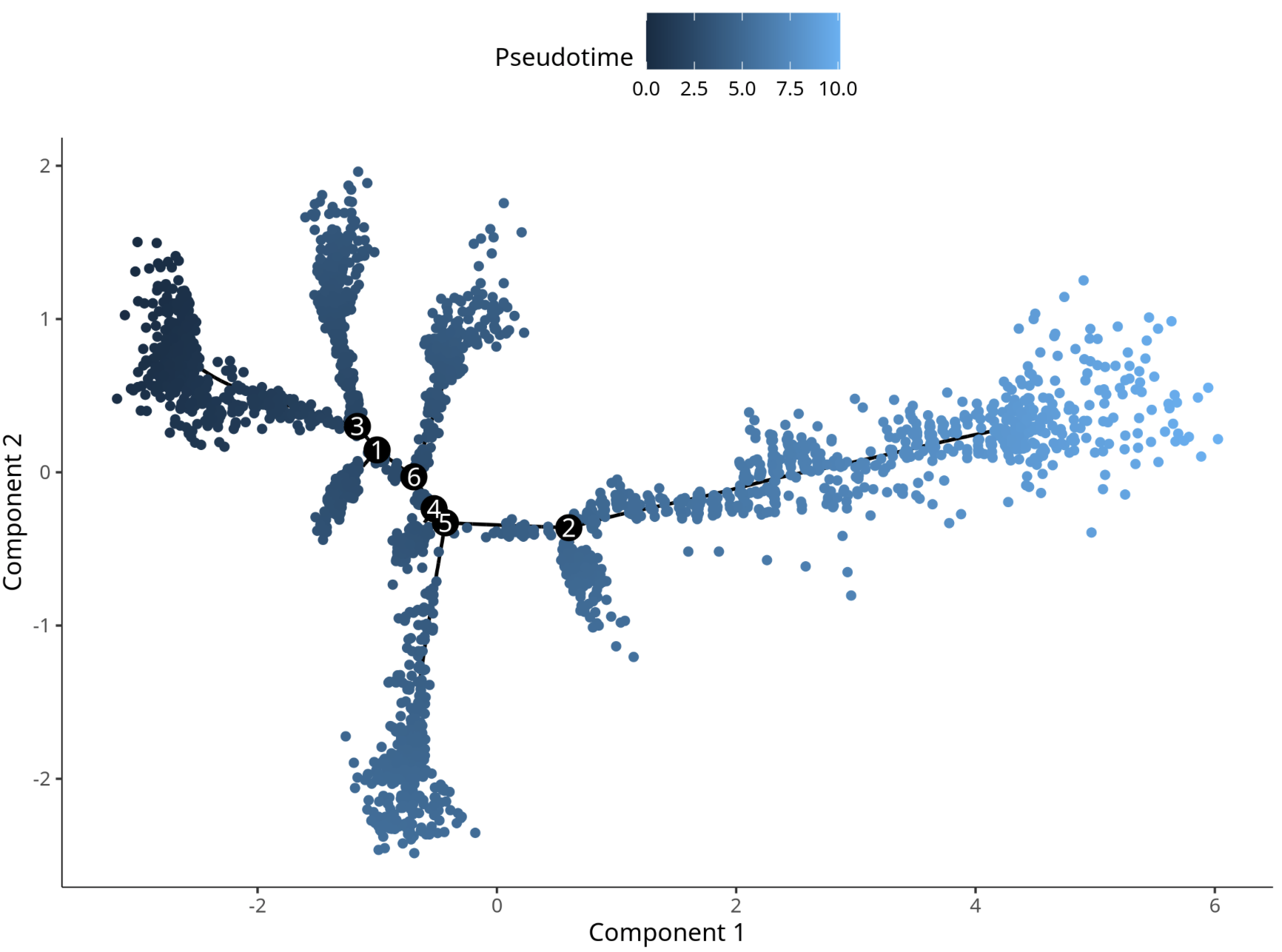
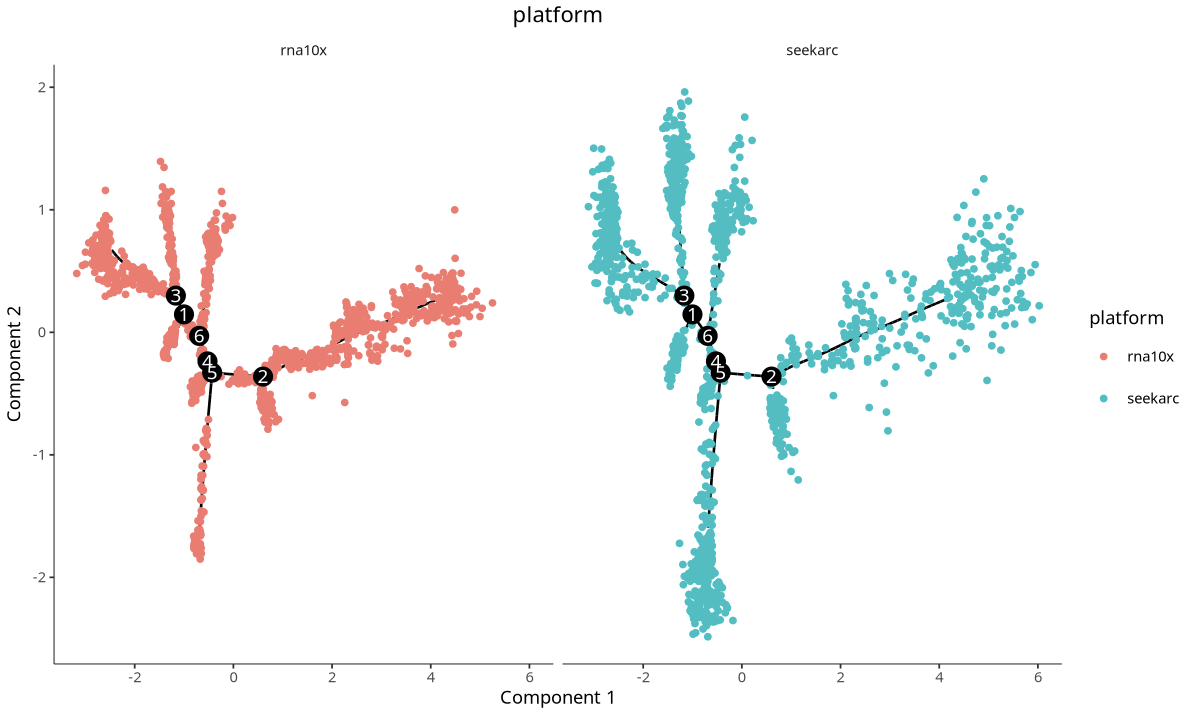
小鼠脑多平台数据验证
标准分析结果:
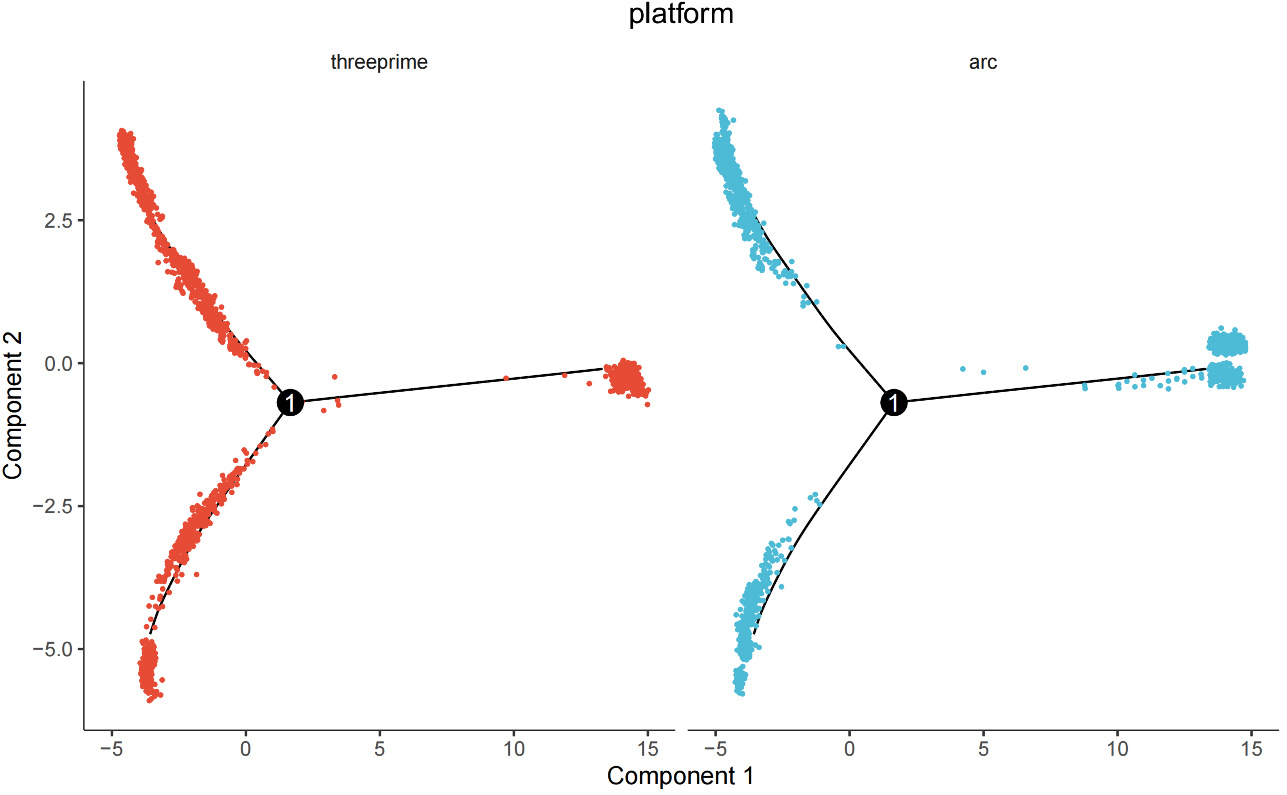
增加 residualModelFormulaStr 参数后:
总结
- 矩阵选择:
counts与data用于 Monocle2 得到的轨迹基本一致。 - 批次校正:在
reduceDimension中加入residualModelFormulaStr = "~Sample"可有效减弱样本/平台驱动的分离,突出生物学信号(参见相关链接);当批次效应显著时,可选择integrated矩阵(参见相关文献 1,文献 2)。 - 跨平台验证:小鼠脑多平台数据验证显示整合与轨迹一致性良好,流程稳健可复用。
- 实践建议:优先使用
counts矩阵;若存在明显平台差异,采用integrated或加入~Sample残差项以保证轨迹连续与生物学可解释性。
hdWGCNA 共表达网络分析
标准分析流程
hdWGCNA 标准分析流程中计算模块特征基因时,也有利用 Harmony 去批次:

consensus 分析流程
对于批次效应较强的数据,hdWGCNA 也建议可以用 consensus 流程:
可视化中的"批次效应"现象
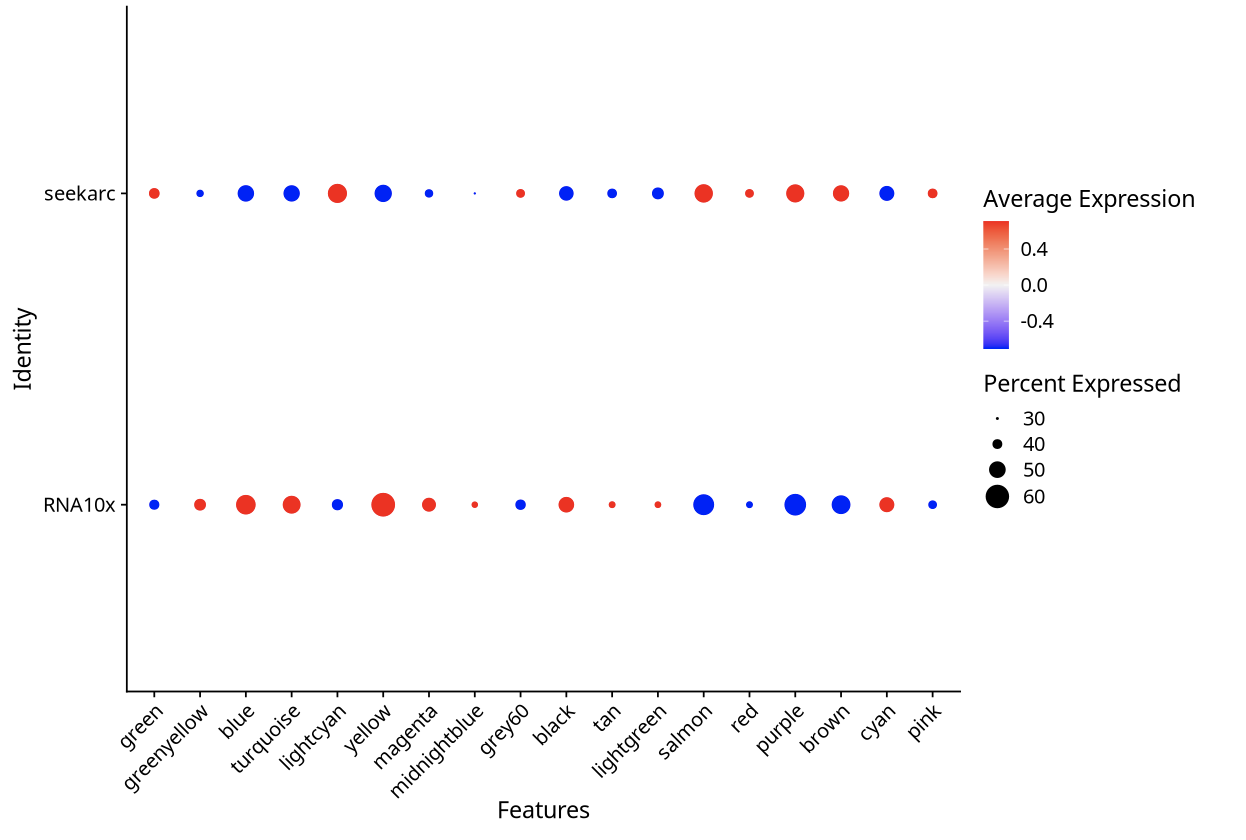
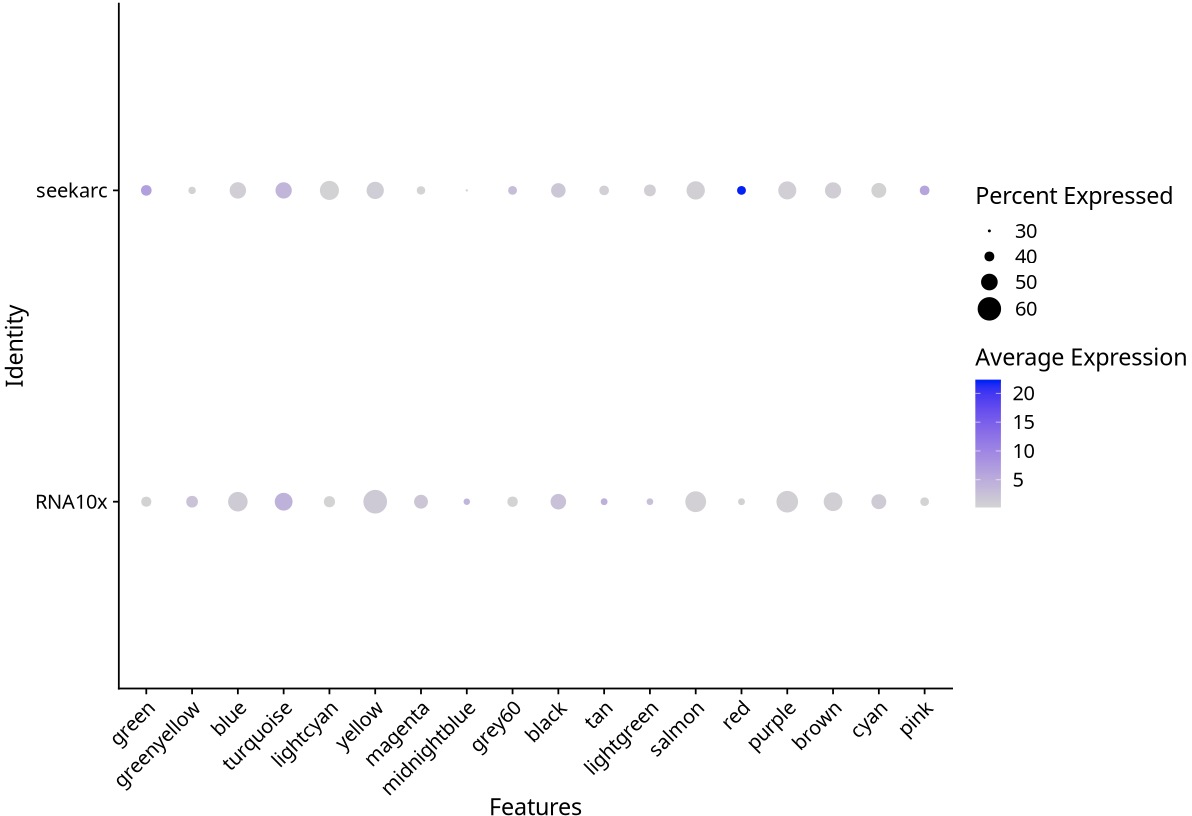
在跨平台单细胞转录组数据分析中,我们经常观察到 DotPlot 可视化结果呈现出明显的"批次效应"特征,即不同平台间的基因表达模式存在显著差异。然而,这种视觉上的差异并不完全反映真实的生物学差异,而是 Seurat 可视化参数设置导致的放大效应。
Seurat::DotPlot 函数中的 scale 参数控制着基因表达值的标准化方式:
- scale=TRUE(默认设置): 对每个基因的平均表达值进行 Z-score 标准化,放大平台间的相对差异。
- scale=FALSE:使用原始平均表达值,保持数据的绝对差异水平。
scale=TRUE(放大差异):
scale=FALSE (保持原始差异):
Seurat 系统在绘图分组少于 5 个时会提示:

总结
- 标准流程稳健:hdWGCNA 标准流程结合 Harmony 可在多数情形下充分削弱批次/平台效应,模块识别稳定。
- 强批次策略:当跨平台差异较强时,采用
consensus流程可提炼跨平台保守模块,提升结果可重复性与可移植性 (参见相关链接)。 - 矩阵选择:网络构建阶段优先
RNA或SCT表达层 (参见相关链接);如何批次效应很强可以考虑以integrated矩阵构建共表达网络。
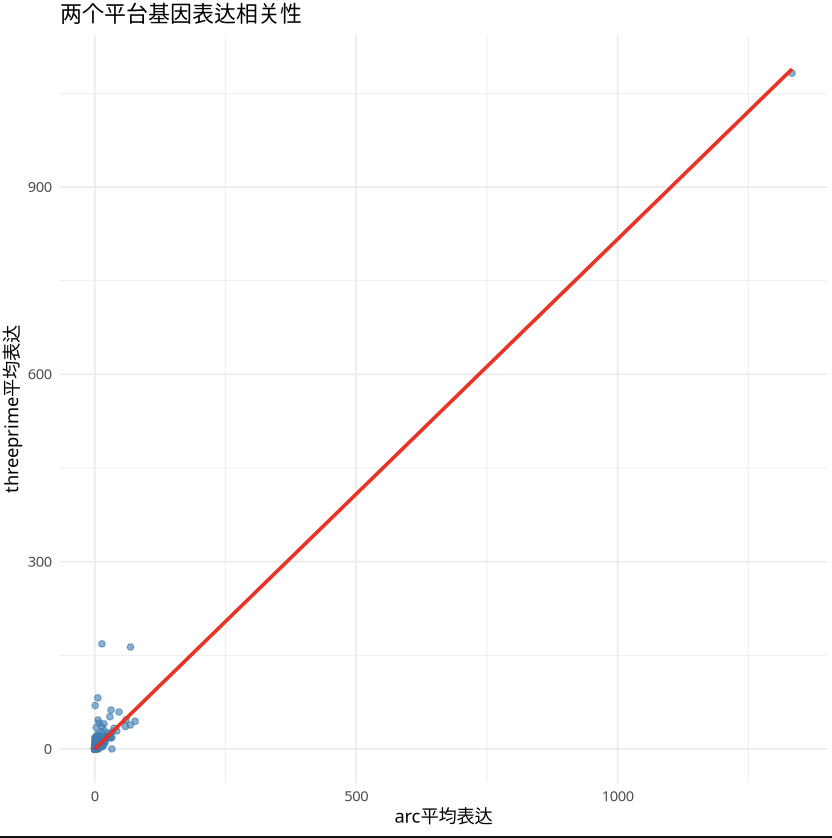
平台间数据基因表达相关性
对同一组织与生物学背景的 SeekArc 与 10x 单细胞转录组数据,SeekArc 与 10x 在基因表达层面具有高度一致性 (Pearson r≈0.97)。
- 基因平均表达相关性 (全基因集平均表达):
- 皮尔逊相关系数为:0.9743382060132
- 图示如下:
结论
SeekArc 平台与 10x Genomics 平台的数据整合分析结果表明:
- 数据兼容性优异:两平台数据具有高度一致性 (相关系数 0.974)。
- 分析流程完整:支持从数据预处理到高级分析的完整流程。
- 批次效应可控:提供多种批次校正策略,确保分析结果可靠。
- 应用前景广阔:为跨平台单细胞转录组研究提供了可靠的技术支撑。
SeekArc 平台作为新一代单细胞 ATAC 一胞双组的转录组测序技术,在数据质量和分析兼容性方面表现优异,能够有效支持多平台数据整合分析,为单细胞转录组研究的深入开展提供强有力的技术保障。
