ATAC+RNA 多组学:细胞注释 (RNA 表达结合 ATAC 可及性)
文档概述
对于一胞多组学数据 (同一细胞同时检测 RNA 和 ATAC),注释工作需要同时考虑基因表达 (RNA) 和染色质可及性 (ATAC) 两种信息源,通过双证据验证提升注释的准确性和鲁棒性。
核心思想:双证据融合
RNA 证据 (转录输出):通过 marker 基因的表达水平识别细胞类型,反映细胞当前的“功能状态”。
ATAC 证据 (调控潜能):通过 marker 基因区域的染色质开放状态识别细胞类型,反映细胞的“调控状态”。
TIP
- 在实际细胞注释过程中,某些细胞类型 (如静息状态的中性粒细胞),RNA 表达量可能很低,单独 RNA 注释容易误判;但染色质结构完整,ATAC 信息可以作为补充证据。
- 有时某些细胞亚群在转录水平相似但调控机制不同,通过同时观察 RNA 和 ATAC,能够识别出单一组学难以区分的细分类别。
双证据注释策略
注释流程
第一步:查看聚类结果 在 WNN UMAP 上可视化聚类结果
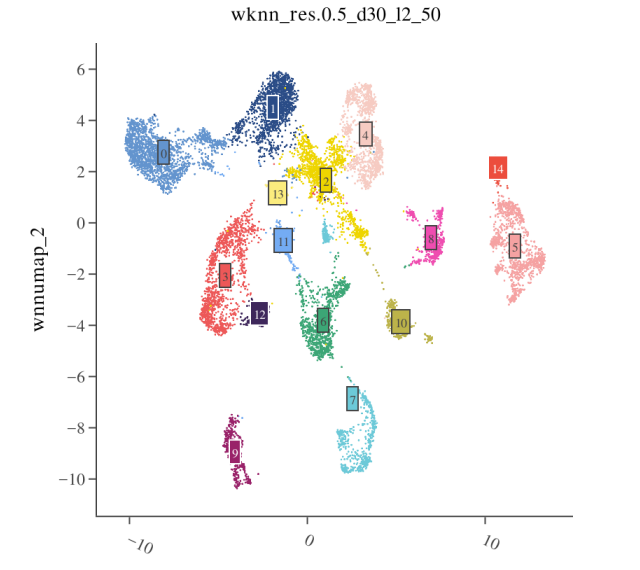
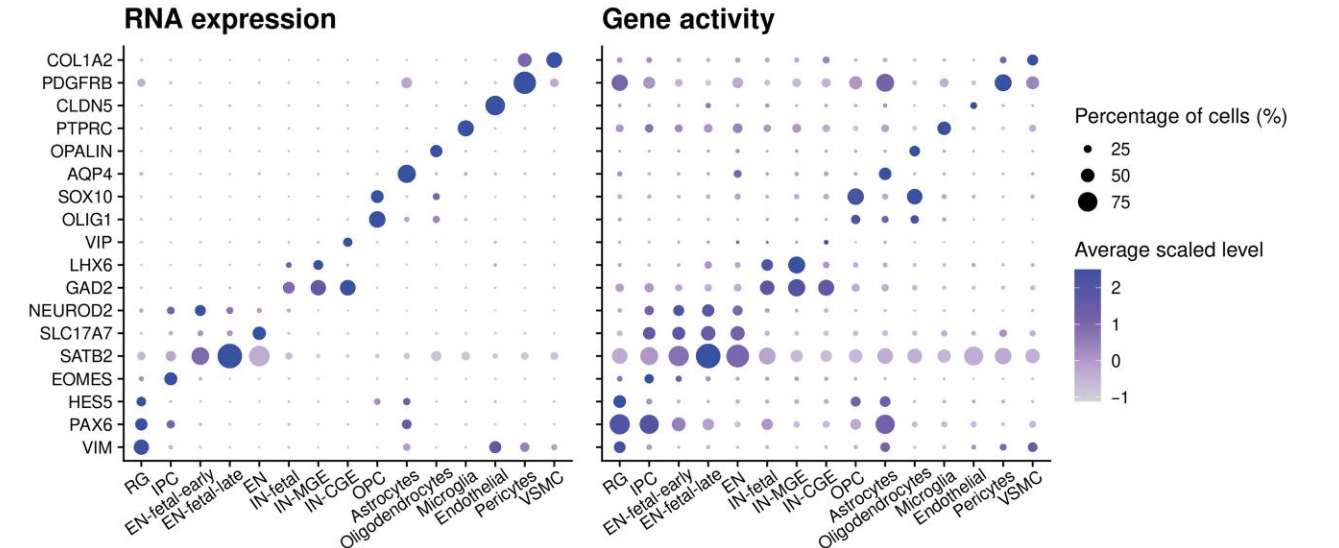
- 第二步:检查 RNA marker 基因 通常在注释之前会先查阅文献,确定同组织细胞类型及其经典 marker 基因列表,用 FeaturePlot() 或 DotPlot() 等函数查看 marker 基因在不同细胞群上的表达情况。
# 假设 data 为你的 Seurat 对象,markers 为你关心的 Marker 基因列表
markers <- c("CD3D", "MS4A1", "NKG7", "LYZ", "GNLY", "PPBP") # 示例 Marker,可根据实际研究替换
# 可视化各 Marker 基因在各聚类的表达分布
DotPlot(
object = data,
features = markers,
dot.scale = 8,
group.by = "seurat_clusters" # 此参数填写 WNN 联合降维聚类的结果
)- 第三步:检查 ATAC 基因活性 通过 GeneActivity() 函数计算基因的可及性分数,用 FeaturePlot() 或 DotPlot() 等函数查看 marker 基因在不同细胞群上的可及性情况。
# 计算基因活力
gene.activities <- GeneActivity(data)
data[["GeneActivity"]] <- CreateAssayObject(counts = gene.activities)
# 切换到 GeneActivity Assay
DefaultAssay(data) <- "GeneActivity"
# DotPlot 可视化
DotPlot(
object = data,
features = markers,
dot.scale = 8,
group.by = "wnn_clusters" # 此参数填写 WNN 联合降维聚类的结果
)
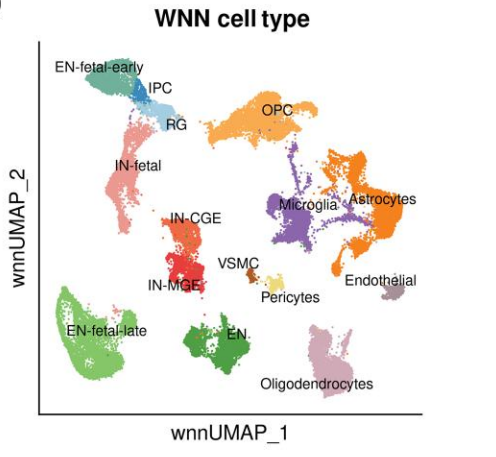
第四步:综合判断并注释
- 如果 RNA 和 ATAC 证据一致,可以确信该聚类为某种细胞类型。
- 如果两者不一致,需要进一步检查:
- 数据质量问题(某一模态信息不可靠)。
- 生物学真实情况(如细胞处于状态转换期)。
- 聚类分辨率不合适(需要调整分辨率重新聚类)。
常见问题
Q1:为什么某些 Marker 基因在 RNA 端高表达,但在 ATAC 端活性较低?
A:这是正常现象,可能的原因包括:
- 转录后调控:某些基因的 RNA 表达可能受转录后调控(如 RNA 稳定性)影响,而不完全依赖染色质开放状态。
- 时间延迟:染色质开放状态的变化通常先于基因表达的变化。如果细胞处于状态转换期,可能出现 ATAC 先开放但 RNA 还未高表达的情况(或反之)。
- 技术因素:ATAC-seq 的信号可能受 peak calling、片段长度分布等因素影响,导致某些基因的活性计算不够准确。
- 生物学复杂性:某些基因的调控可能涉及远端增强子,基因活性计算只考虑了基因体和启动子区域,可能遗漏了重要信息。
建议:优先关注在 RNA 和 ATAC 端都表现出高信号的 Marker 基因,这些基因的双证据支撑最强,注释最可靠。对于只在单一模态高表达的基因,需要结合其他证据(如已知的 Marker 列表、参考数据集等)综合判断。
Q2:基因活性(GeneActivity)的计算是否准确?它与 RNA 表达的关系是什么?
A:
基因活性的准确性:
- 基因活性是基于 peak 片段数计算的,受测序深度、peak calling 质量等因素影响,可能存在一定误差。
- 基因活性主要反映基因调控区域的染色质开放状态,而非直接的转录活性。
基因活性与 RNA 表达的关系:
- 正相关但非完全一致:通常情况下,基因活性高的基因,RNA 表达也较高,但两者并非完全对应。
- 调控的时间差:染色质开放通常先于转录发生,因此某些情况下可能出现 ATAC 高活性但 RNA 低表达的情况(基因即将被激活)。
- 其他调控机制:RNA 表达还受转录后调控、RNA 稳定性等因素影响,不完全依赖染色质状态。
建议:将基因活性作为 RNA 表达的补充证据,而非替代证据。当两者一致时,注释更可靠;当两者不一致时,需要结合其他信息综合判断。
Q3:注释结果中出现了"Unknown" 或无法确定的聚类,应该怎么办?
A:
检查是否是技术问题:
- 查看该聚类的质控指标,确认是否为低质量细胞或 doublet。
- 检查该聚类在 UMAP 空间中的位置,是否与已知细胞类型重叠或相邻。
扩大 Marker 基因列表:
- 查找该聚类的 Top Marker 基因,检查是否有组织特异性或稀有细胞类型的 Marker。
- 使用数据库 (如 CellMarker、PanglaoDB) 查找可能的细胞类型。
暂时标记为"未确定":
- 如果确实无法确定,可以暂时标记为"Unknown"或"Unassigned",在后续分析中重点关注这些细胞,看是否有新的发现。
Q4:如何验证注释结果的准确性?
A:
双证据一致性:检查每个注释细胞类型的 Marker 基因是否在 RNA 和 ATAC 端都高表达。
UMAP 空间分布:相同细胞类型的聚类应该在 UMAP 空间中聚集或形成连续的区域。
Marker 基因特异性:使用 DotPlot 或 VlnPlot 检查 Marker 基因是否在目标细胞类型中特异性高表达。
