3. 分析流程与内容解读
免疫组库分析通常包含以下核心步骤,每一步都揭示了免疫应答的不同层面信息:
3.1 细胞质控
细胞质控是确保单细胞免疫组库分析准确可靠的基石。此步骤旨在通过一系列严格的质量评估指标,过滤掉低质量的细胞和测序数据,为后续的生物信息学分析提供高质量的输入。为了保证分析结果的可靠性,我们关注以下核心质控指标:
- 文库质量:合格的 TCR/BCR 文库在 Agilent 4200 TapeStation 检测时,应在 250-1500bp 范围内呈现特异性尖峰,且文库浓度不低于 1ng/μL。
- 测序深度:建议每个细胞的平均测序 Reads 数(Mean Read Pairs per Cell)不低于 5,000,以确保能够捕获到足够的信息进行准确的 Contig 拼接。
- 数据质量:
- Estimated Number of Cells:评估捕获到的细胞数量。
- Valid Barcodes:带有有效细胞 Barcode 的 Reads 比例,通常应 > 80%。
- Q30 Bases in Barcode/UMI:Barcode 和 UMI 序列中,测序质量值大于等于 Q30 的碱基比例,是衡量测序准确性的关键指标,通常要求 > 90%。
- Reads Mapped to Any V(D)J Gene:比对到 V(D)J 参考基因的 Reads 比例,反映了 V(D)J 富集的特异性和效率。
- Number of Cells With Productive V-J Spanning Pair:鉴定出具有完整且有功能的 V-J 配对链的细胞数量,这是后续克隆型分析的基础。
3.2 免疫组库拼接与克隆型定义
此步骤的目标是将原始测序数据转化为具有生物学意义的免疫细胞克隆,核心流程包括TCR/BCR结构识别、序列组装注释与最终的克隆型定义。
3.2.1 TCR/BCR 结构简介
TCR 和 BCR 的结构决定了其识别抗原的特异性。其可变区由 V、D、J 基因片段重排形成,其中 CDR3(互补决定区3) 是变异最大的区域,直接决定了抗原结合的特异性。
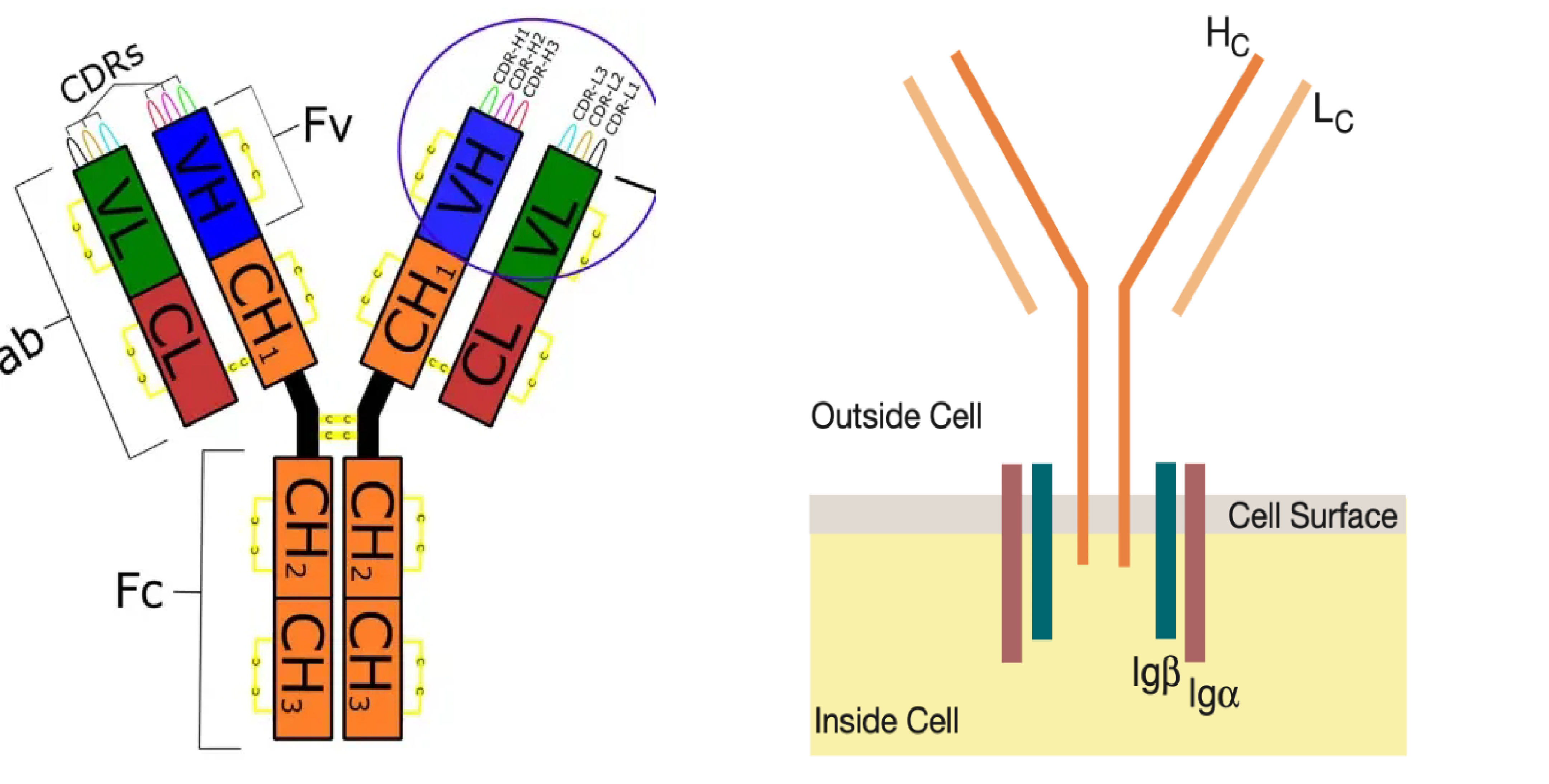 BCR 由两条重链(H)和两条轻链(L)组成,其抗原结合能力由 V 区决定。
BCR 由两条重链(H)和两条轻链(L)组成,其抗原结合能力由 V 区决定。
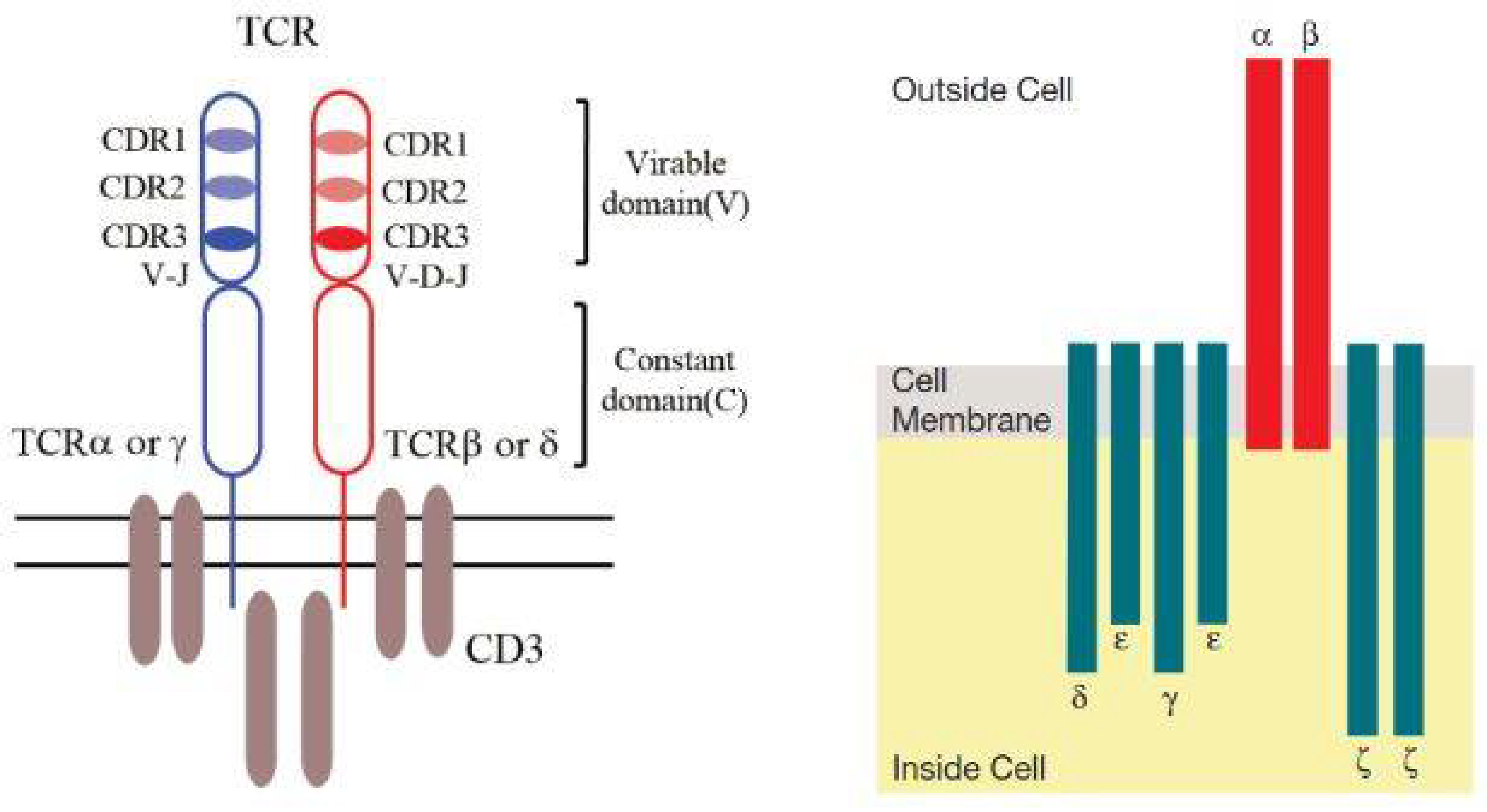 TCR 主要为 αβ 链或 γδ 链组成的异二聚体,通过可变区识别并结合由 MHC 分子递呈的抗原肽。
TCR 主要为 αβ 链或 γδ 链组成的异二聚体,通过可变区识别并结合由 MHC 分子递呈的抗原肽。
3.2.2 序列组装与 VDJ 注释
SeekSoul® Tools 采用以下流程进行序列组装与注释:
- 序列提取与降采样:利用 TRUST4 工具从原始数据中提取可能包含 TCR/BCR 序列的 Reads。为平衡计算效率,对于 Reads 数超过 80,000 的细胞,会进行降采样处理。
- UMI 矫正与序列组装:在组装前,使用
umitools对 UMI 进行矫正,以消除测序和 PCR 扩增引入的错误。随后,基于矫正后的 UMI 和 Reads,TRUST4 对每个细胞的 V(D)J 序列进行组装,生成 Contigs。 - VDJ 基因注释:利用 TRUST4 的
annotator模块对组装好的 Contigs 进行注释,确定 V/D/J/C 基因的使用情况以及 CDR3 的精确位置和序列。- Primary Chain 选择:在一个细胞内,如果某条链(如 TRB)有多条注释结果,软件会选择 UMI 数量最高 的那条 Contig 作为该链的
primary_chain。
- Primary Chain 选择:在一个细胞内,如果某条链(如 TRB)有多条注释结果,软件会选择 UMI 数量最高 的那条 Contig 作为该链的
3.2.3 Clonotype 定义
Clonotype(克隆型)是一组具有相同或高度相似免疫受体的细胞。SeekSoul® Tools 通过以下精细的步骤来定义克隆型:
- 高质量 Contig 筛选:首先,对细胞进行筛选,要求其支持 Contig 的 UMI 总数不少于 3。
- 基于 CDR3 序列相似性聚类:使用 dandelion 工具,基于 CDR3 氨基酸序列 的相似性来定义克隆。相似性阈值根据受体类型有所不同:
- BCR 设置为 85%
- TCR 设置为 100%
- 单链克隆型的优化处理:为优化单链克隆型(如仅含TRB链),软件会自动将其与具有相同VJ基因及CDR3序列的双链克隆型进行匹配与合并,以确保克隆型定义的准确性。
TIP
Contig 拼接的质量和 Clonotype 定义的准确性直接决定了后续所有分析的可靠性。在分析报告中,应重点关注高质量(high_confidence, full_length, productive)Contig 的数量和最终鉴定出的克隆型数量。
3.3 细胞映射
此步骤的目标是将VDJ库(克隆型身份)与GEX库(转录组功能)在单细胞水平上进行精确匹配,从而将细胞的克隆型特征与其功能状态关联起来。
3.3.1 映射原理
由于 VDJ 库和 5' GEX 库共享同一套细胞 Barcode 系统,我们可以精确地将每个细胞的 TCR/BCR 序列信息与其转录组表达谱关联起来。这使得我们能够在特定的细胞亚群(如耗竭性 T 细胞、记忆 B 细胞)中研究其免疫受体的特征。
3.3.2 映射结果解读
通过 UMAP 等降维可视化方法,可以直观地展示 VDJ 信息在不同细胞亚群中的分布情况。
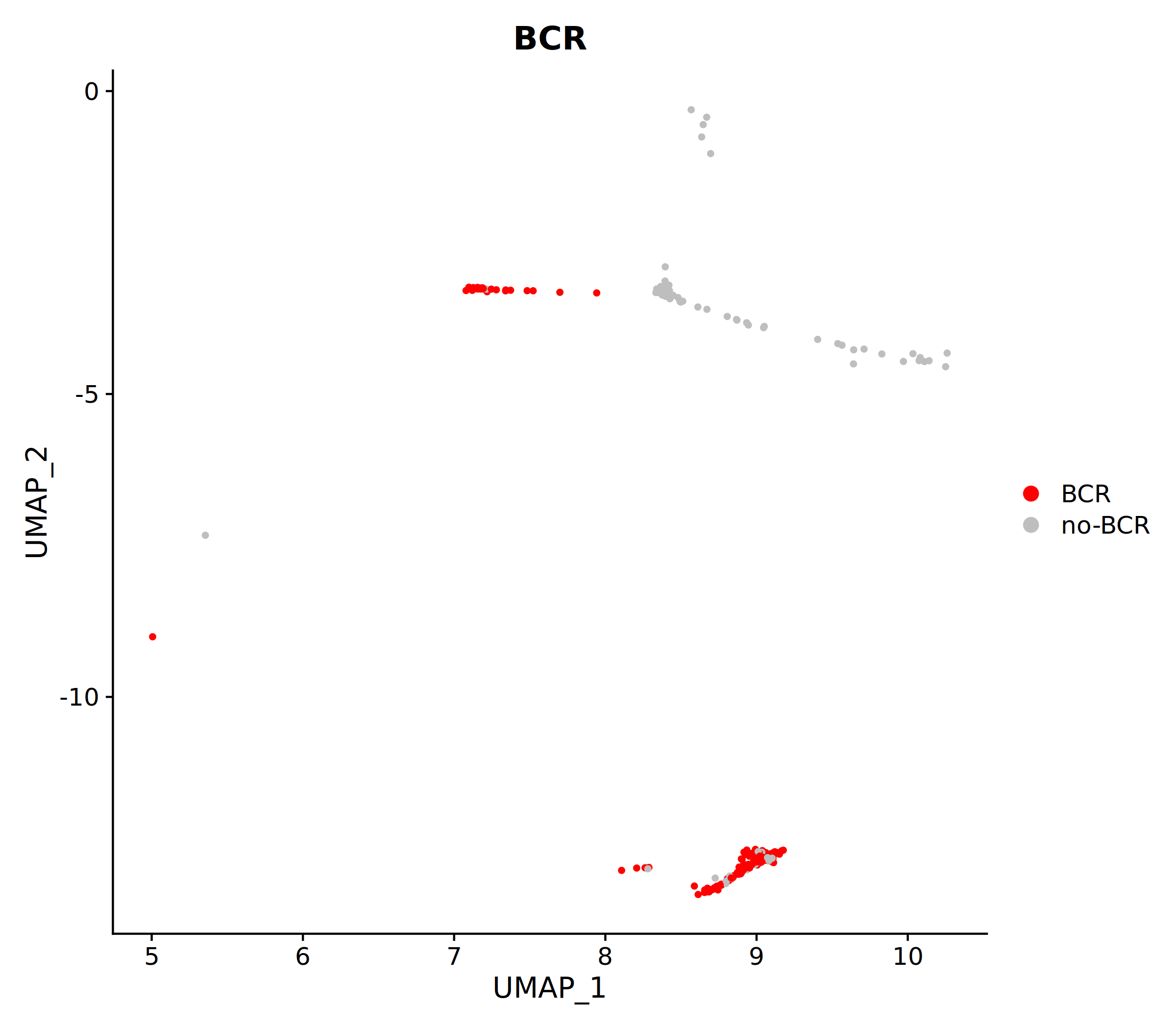 上图展示了 BCR 检出细胞(红色)在 UMAP 聚类图中的分布。通过观察红色细胞主要富集在哪些细胞群中,可以分析不同 B 细胞亚群的 BCR 检出率及其生物学意义。
上图展示了 BCR 检出细胞(红色)在 UMAP 聚类图中的分布。通过观察红色细胞主要富集在哪些细胞群中,可以分析不同 B 细胞亚群的 BCR 检出率及其生物学意义。
- "TCR/BCR" 细胞:在 VDJ 库中成功检测到免疫受体序列的细胞。
- "no-TCR/BCR" 细胞:未检测到免疫受体序列的细胞。可能的原因包括:
- 该细胞本身不表达 TCR/BCR(如巨噬细胞、成纤维细胞)。
- 免疫受体基因表达水平极低,低于检测阈值。
- 细胞裂解不充分或 V(D)J 富集效率不佳。
NOTE
细胞映射的检出率是衡量实验质量的重要指标之一。较低的检出率可能意味着需要优化实验条件或提高测序深度。通过分析特定细胞类型的检出率,可以为后续的功能研究提供重要线索。
3.4 V/J 基因特征分析
V/J 基因的使用频率和配对模式是免疫组库的关键特征,反映了免疫系统对抗原的响应偏好和克隆选择。通过分析这些特征,可以深入了解免疫应答的广度与特异性。
3.4.1 V/J 基因使用频率 (Gene Usage)
该分析统计 V 基因和 J 基因在免疫组库中的使用频率。在免疫应答过程中,某些 V/J 基因可能因为能有效识别特定抗原而被优先选择和扩增,导致其在组库中的占比显著高于其他基因。
- 分析方法:分别对整体和每组中每个细胞类型的 V 基因和 J 基因丰度分布情况进行统计。
- 结果解读:通常使用柱状图展示各个 V/J 基因的相对丰度。图中 X 轴代表不同的 V 或 J 基因,Y 轴代表其在总克隆型中的百分比(frequency)。优势表达的基因(即较高的柱子)可能与样本所处的特定免疫状态相关。
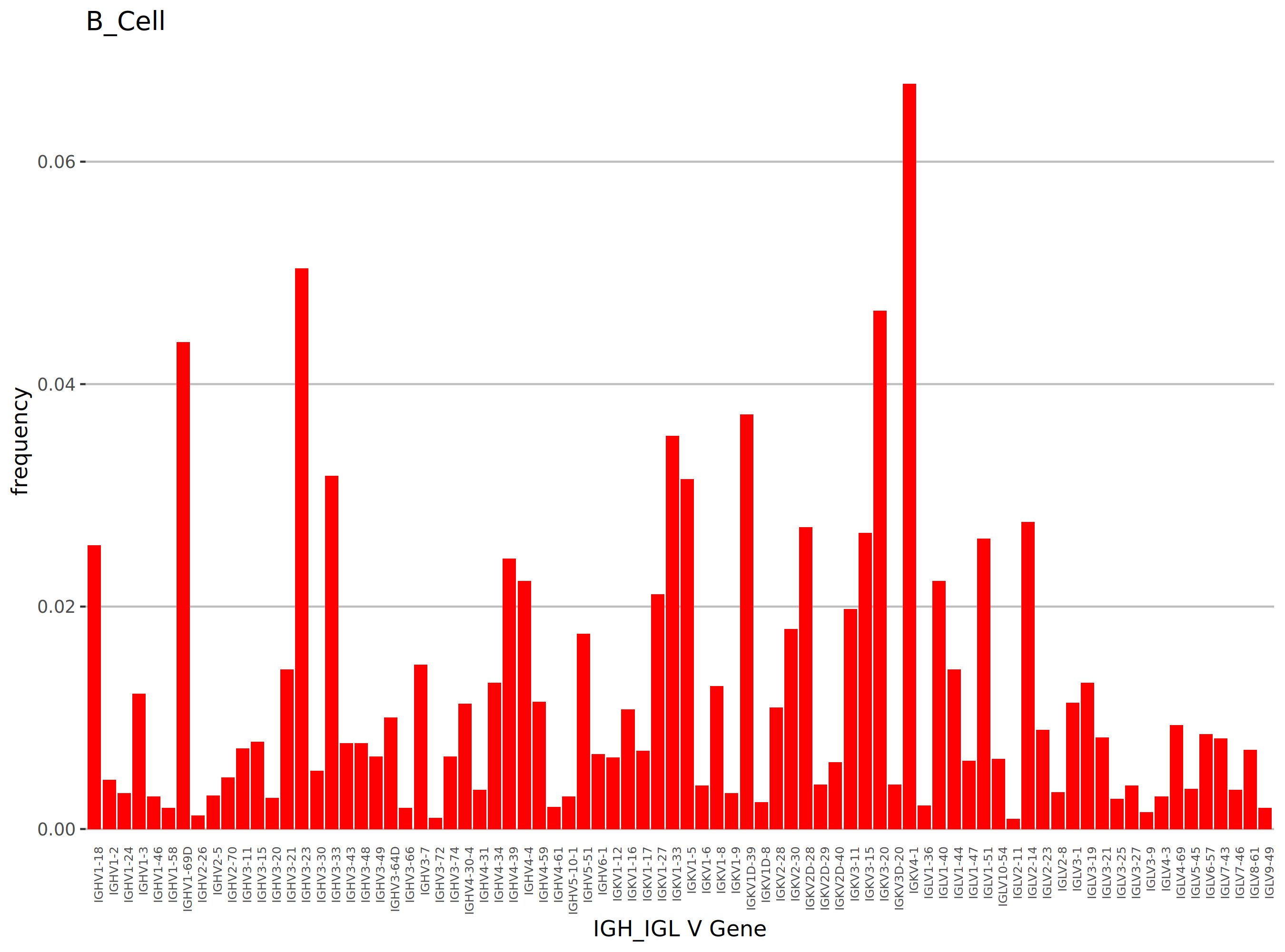 以BCR为例,TCR分析同理。上图展示了 B 细胞中不同 V 基因的使用频率分布,可直观识别丰度最高的优势基因。横坐标为 IGH_IGL V 基因名称,纵坐标为频率。
以BCR为例,TCR分析同理。上图展示了 B 细胞中不同 V 基因的使用频率分布,可直观识别丰度最高的优势基因。横坐标为 IGH_IGL V 基因名称,纵坐标为频率。
TIP
解读要点:
- 通过比较不同细胞类型或不同样本间的 V/J 基因使用模式,可以识别特定的免疫应答特征。
- 显著富集的 V/J 基因可能提示存在针对特定抗原的克隆扩增。
- 结合后续的差异分析热图,可以更全面地评估细胞类型间的 V/J 基因使用差异。
3.4.2 V/J 基因差异分析
为了更系统地比较不同细胞类型间的 V/J 基因使用差异,可以对整体和每组中细胞类型间的 V/J 基因频率进行差异分析。
- 结果解读:热图中每一行代表一个 V 或 J 基因,每一列代表一个细胞类型。颜色编码表示基因使用频率的标准化得分(Z-Score),橙色/红色表示该基因在该细胞类型中使用频率较高(上调),蓝色表示使用频率较低(下调)。通过层次聚类,可以识别具有相似 V/J 基因使用模式的细胞类型群组。
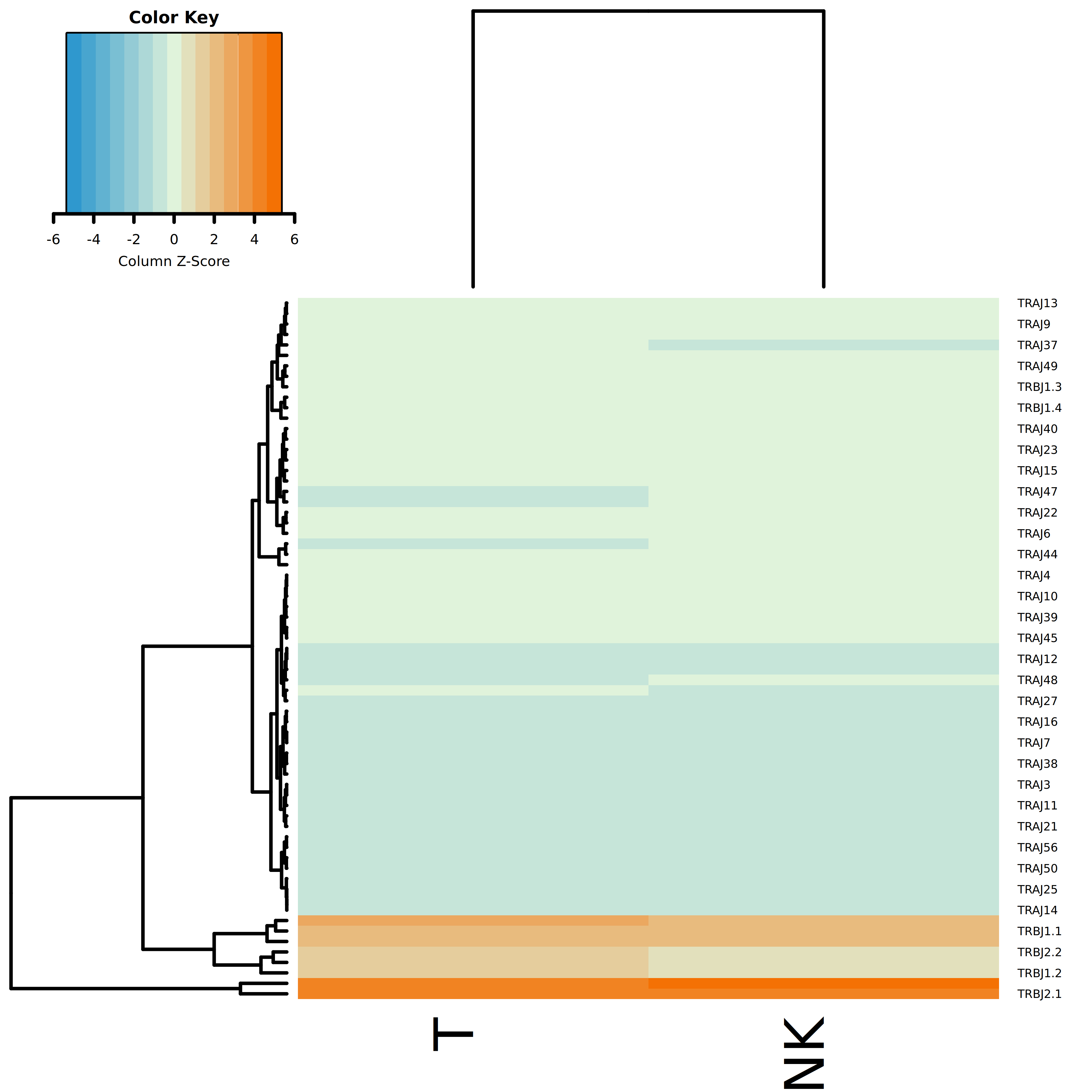 上图展示了不同细胞类型间 J 基因使用频率的差异分析热图。通过颜色深浅和聚类树可以识别细胞类型特异性的基因使用模式。
上图展示了不同细胞类型间 J 基因使用频率的差异分析热图。通过颜色深浅和聚类树可以识别细胞类型特异性的基因使用模式。
3.4.3 V-J 基因配对分析
除了单个基因的使用频率,V 和 J 基因的配对组合也包含重要的生物学信息。某些特定的 V-J 基因组合可能更倾向于被选择,形成优势配对,这同样暗示了其在抗原识别中的重要作用。
- 分析方法:在不同免疫组库中统计 V-J 基因对的配对频率,可以反映 CDR3 或免疫组库的变化特征。通过比较不同免疫时期高丰度的 V-J 基因对,可以识别特异性表达的免疫基因组合。
- 结果解读:V-J 配对信息常通过和弦图(Chord Diagram)进行可视化。和弦图能够直观展示不同 V 基因(外环的一侧)和 J 基因(外环的另一侧)之间的连接关系,连接条带的宽度与该 V-J 配对的丰度成正比。条带越宽,表示该配对组合在样本中的出现频率越高。
 上图通过 V-J 基因配对和弦图,清晰地展示了 T 细胞中优势 V-J 配对组合及其相对丰度。外环显示不同的 V 和 J 基因,连线宽度代表配对频率。
上图通过 V-J 基因配对和弦图,清晰地展示了 T 细胞中优势 V-J 配对组合及其相对丰度。外环显示不同的 V 和 J 基因,连线宽度代表配对频率。
3.5 CDR3 特征分析
CDR3 区是 TCR/BCR 可变区的核心,直接决定了抗原结合的特异性。该区域由 V、D、J 基因片段共同编码,并通过随机的核苷酸插入和删除形成极高的多样性。因此,对 CDR3 的特征分析是免疫组库研究的重中之重。
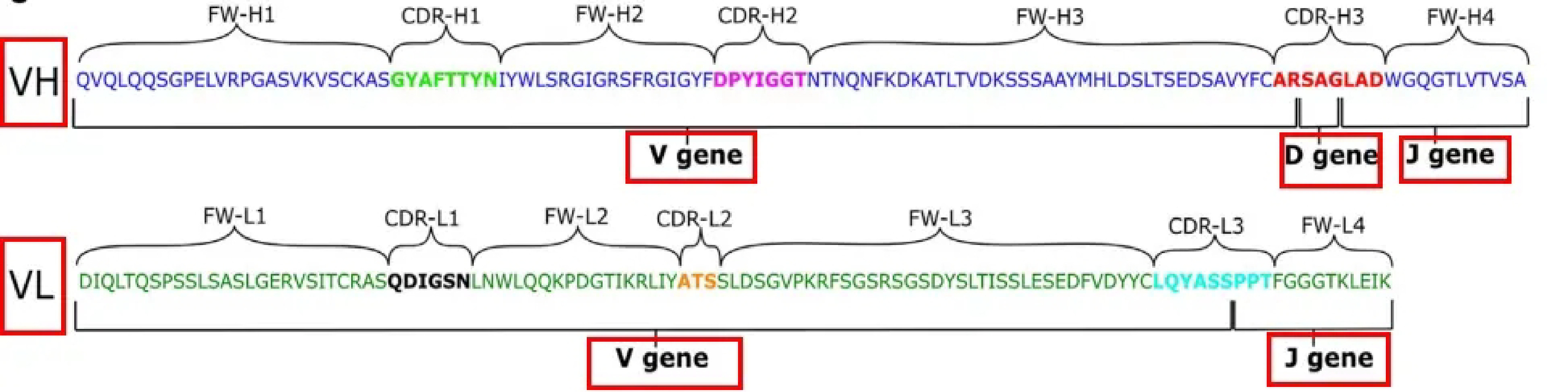 BCR 和 TCR 的可变区包含三个互补决定区(CDR1/CDR2/CDR3),其中 CDR3 的变异最大,直接决定了抗原结合的特异性。
BCR 和 TCR 的可变区包含三个互补决定区(CDR1/CDR2/CDR3),其中 CDR3 的变异最大,直接决定了抗原结合的特异性。
3.5.1 CDR3 序列长度与 V 基因组合分析
分析 CDR3 与 V 基因的组合特征,可以进一步揭示克隆选择的规律。通过"谱系图"(Spectratype Plot)可以同时展示 CDR3 长度分布与 V 基因使用频率的关系。
- 分析方法:对整体和每组中不同 CDR3 序列长度下 V 基因的分布情况进行统计。
- 结果解读:在谱系图中,X 轴为 CDR3 的碱基序列长度(bp),Y 轴为频率(frequency)。每个柱子代表特定长度的 CDR3,柱内的不同颜色分段代表构成该长度 CDR3 的不同 V 基因来源及其比例。图中对丰度前12的 V 基因用不同颜色填充,其余为灰色(Other)。这有助于识别是否存在特定长度的 CDR3 与特定 V 基因的优势组合。
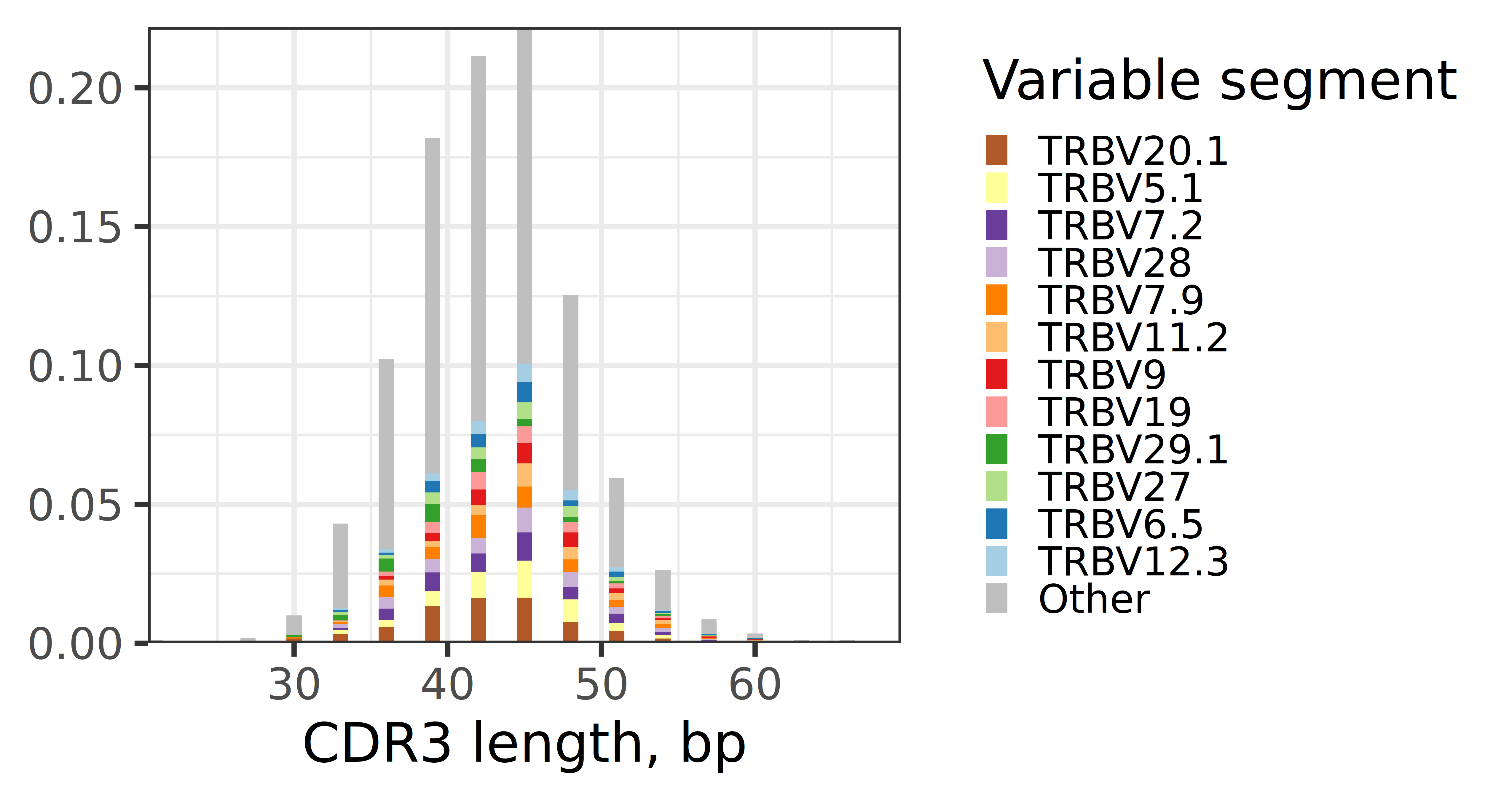 上图展示了 T 细胞中不同 CDR3 长度下,各类 V 基因的分布情况。横坐标为 CDR3 碱基序列长度,纵坐标为频率,不同颜色代表不同的 V 基因(TRBV)。通过此图可以分析特定 V 基因是否倾向于形成特定长度的 CDR3。
上图展示了 T 细胞中不同 CDR3 长度下,各类 V 基因的分布情况。横坐标为 CDR3 碱基序列长度,纵坐标为频率,不同颜色代表不同的 V 基因(TRBV)。通过此图可以分析特定 V 基因是否倾向于形成特定长度的 CDR3。
NOTE
生物学意义:
- CDR3 长度与 V 基因的组合模式反映了免疫受体的结构特征和多样性。
- 某些 V 基因可能倾向于产生特定长度范围的 CDR3,这与其编码区的结构特点有关。
- 异常的 CDR3 长度分布或 V 基因偏好性可能提示存在克隆扩增或免疫选择。
3.6 CDR3 长度分布统计
CDR3 是 V(D)J 基因编码的核心区域,通常包含 V 基因的一部分、全部 D 基因以及部分 J 基因,因此是 BCR/TCR 上最具代表性、最具辨识度的区域。在绝大多数免疫研究中,会把 CDR3 序列作为定义和识别某一个特定 BCR 或 TCR 的唯一依据,即具有相同 CDR3 序列的细胞为一个克隆型。CDR3 序列的长度并非完全随机,其分布特征可以反映免疫组库的整体状态和克隆扩增程度。
3.6.1 CDR3 长度分布的生物学意义
在正常的免疫系统中,CDR3 长度通常呈现一种准高斯(bell-shaped)分布,反映了免疫组库的多样性和随机性。
- 正常分布:多样化的免疫组库通常表现为覆盖多种长度、形状对称的钟形曲线,提示免疫系统具有广泛的抗原识别能力。
- 分布偏移或寡克隆优势:当机体针对特定抗原(如病毒感染或肿瘤)产生强烈的免疫应答时,少数高效识别抗原的 T/B 细胞会大量扩增。这会导致其对应的 CDR3 长度在分布图上形成一个或几个突出的尖峰,使整体分布偏离标准形态。因此,CDR3 长度分布是评估克隆扩增程度的直观指标。
3.6.2 CDR3 长度分布分析方法
分别对整体和每组中每个细胞类型的 CDR3 序列长度分布情况进行统计。
- 柱状图展示:横坐标为 CDR3 序列长度,纵坐标为细胞数(counts),可以直观显示每个长度的绝对数量。
- 折线图展示:横坐标为 CDR3 序列长度(CDR3 Length),纵坐标为占比(Percent),可以清晰比较不同细胞类型或样本间的 CDR3 长度分布模式。
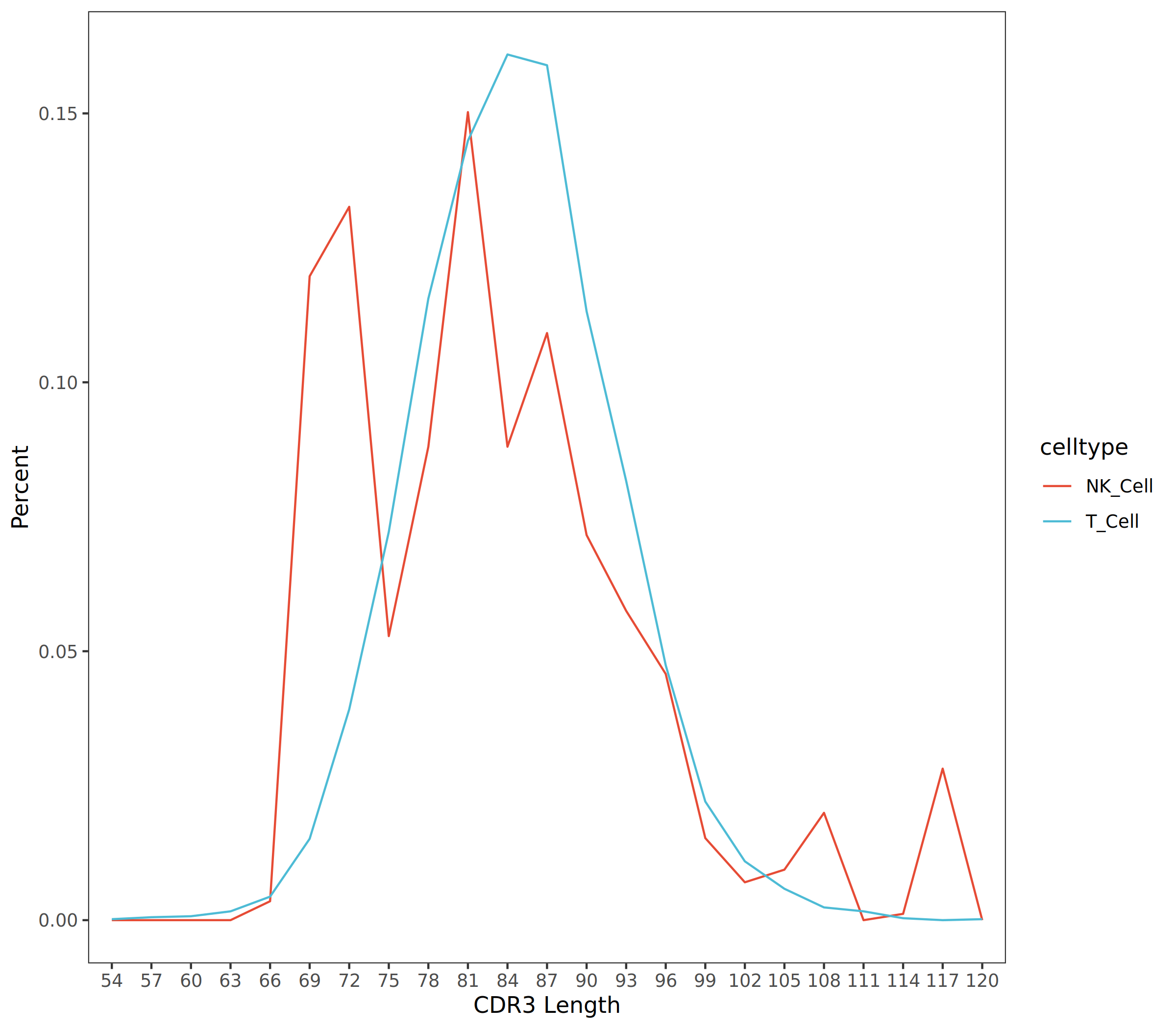 上图展示了不同细胞群(T_Cell 和 NK_Cell)中 CDR3 的长度分布情况。横坐标为 CDR3 长度,纵坐标为百分比。通过比较不同细胞群的分布曲线,可以评估各细胞群的克隆扩增状态和免疫组库多样性。T 细胞和 NK 细胞呈现出不同的 CDR3 长度分布特征,T 细胞的峰值主要集中在81-87bp左右,而 NK 细胞则在72和81bp附近有两个峰值。
上图展示了不同细胞群(T_Cell 和 NK_Cell)中 CDR3 的长度分布情况。横坐标为 CDR3 长度,纵坐标为百分比。通过比较不同细胞群的分布曲线,可以评估各细胞群的克隆扩增状态和免疫组库多样性。T 细胞和 NK 细胞呈现出不同的 CDR3 长度分布特征,T 细胞的峰值主要集中在81-87bp左右,而 NK 细胞则在72和81bp附近有两个峰值。
TIP
结果解读要点:
- 对称的钟形曲线提示正常的免疫组库多样性。
- 显著的尖峰或峰值偏移可能提示存在克隆扩增或免疫选择。
- 不同细胞类型可能具有不同的 CDR3 长度分布特征,这与其功能状态和发育阶段有关。
- 测定特定 CDR3 序列出现的频率可以反映免疫细胞扩增的程度,为后续的克隆型分析提供基础。
3.7 克隆丰度统计
克隆丰度(Clone Abundance)也称为克隆大小(Clone Size),是指具有相同 CDR3 序列的细胞数量。克隆丰度统计是评估免疫组库克隆扩增状态的重要指标,能够直观反映免疫应答的强度和特异性。
3.7.1 克隆丰度的生物学意义
在正常生理状态下,免疫组库中的大多数克隆型仅包含少量细胞(clone size ≤ 2),呈现高度多样化的状态。然而,当机体遭遇特定抗原刺激(如病原体感染、肿瘤抗原或自身抗原)时,能够识别该抗原的 T/B 细胞会发生克隆扩增,导致特定克隆型的丰度显著增加。
- 非克隆性细胞(Non-clonal):clone size < 2 的克隆型,代表未经扩增或扩增程度极低的细胞群,反映免疫组库的基础多样性。
- 克隆性细胞(Clonal):clone size ≥ 2 的克隆型,提示该细胞群可能经历了抗原驱动的克隆扩增,在免疫应答中发挥重要作用。
- 高丰度克隆:clone size 很大的克隆型(如 >100)通常提示强烈的免疫应答或克隆优势,可能与病理状态(如慢性感染、自身免疫病或肿瘤)相关。
3.7.2 克隆丰度分析方法
分别对整体和每组中每一个细胞群的克隆型丰度分布情况进行统计。横坐标为每一个克隆型所包含的细胞数(即克隆丰度或克隆大小),纵坐标为具有相同 clone size 的克隆数目。通过折线图展示不同细胞群的克隆丰度分布模式。
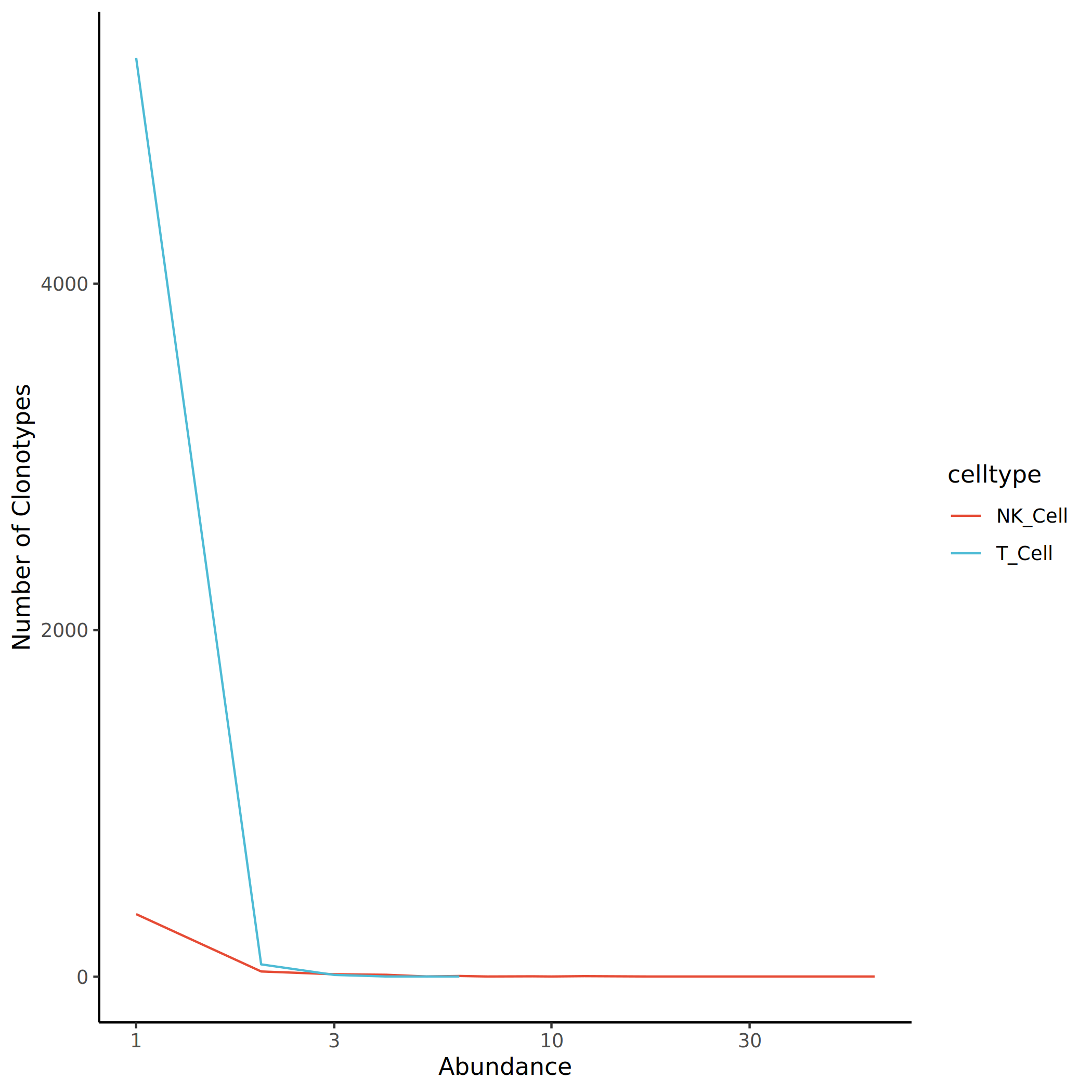 上图展示了不同细胞群的克隆型丰度分布情况。横坐标为克隆大小(Clone Size),纵坐标为克隆型数目(Number of Clonotypes)。从图中可以看出,大部分克隆型的 clone size 较小(< 10),而少数克隆型表现出较高的丰度,提示存在克隆扩增现象。
上图展示了不同细胞群的克隆型丰度分布情况。横坐标为克隆大小(Clone Size),纵坐标为克隆型数目(Number of Clonotypes)。从图中可以看出,大部分克隆型的 clone size 较小(< 10),而少数克隆型表现出较高的丰度,提示存在克隆扩增现象。
3.7.3 Log2 克隆丰度分布
为了更清晰地区分非克隆性(non-clonal)和克隆性(clonal)细胞群,可以对克隆丰度进行 log2 转换。横坐标表示每一个克隆型的 log2 clone size,纵坐标为该 clone size 下的克隆型数目。在图中,辅助线 x=1 左侧的克隆型 clone size < 2(non-clonal),右侧的克隆型 clone size ≥ 2(clonal)。
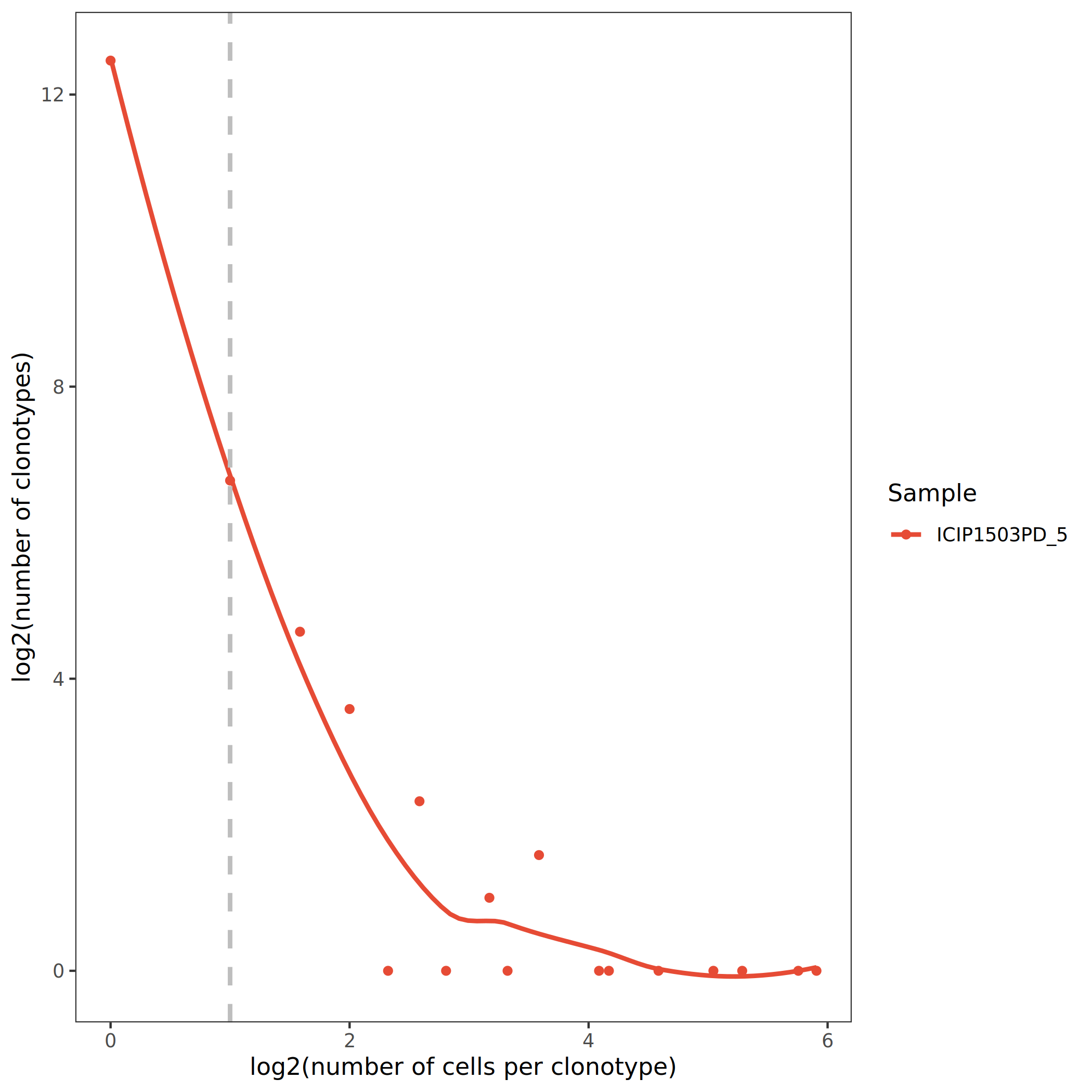 上图以 log2 尺度展示克隆丰度分布。辅助虚线 x=1 将克隆型分为非克隆性(左侧)和克隆性(右侧)两部分。可以观察到,样本中存在一定比例的克隆扩增细胞(x>1),这些克隆可能参与了特异性免疫应答。
上图以 log2 尺度展示克隆丰度分布。辅助虚线 x=1 将克隆型分为非克隆性(左侧)和克隆性(右侧)两部分。可以观察到,样本中存在一定比例的克隆扩增细胞(x>1),这些克隆可能参与了特异性免疫应答。
TIP
结果解读要点:
- 克隆丰度分布呈现右偏(right-skewed)特征,即大量低丰度克隆和少量高丰度克隆共存,符合正常免疫组库的特征。
- Clone size ≥ 2 的克隆比例可以反映克隆扩增的程度。比例越高,提示免疫应答越活跃。
- 高丰度克隆的出现可能与特定抗原刺激、慢性炎症或肿瘤微环境相关,需要结合临床背景综合分析。
- 不同细胞亚群的克隆丰度分布差异可以揭示其在免疫应答中的不同角色。
3.8 克隆型占比分析
克隆型占比分析通过评估高丰度克隆型在免疫组库中的比例,来量化克隆扩增的程度和免疫应答的集中性。该分析有助于识别优势克隆群体,并评估免疫组库的克隆稳态(Clonal Homeostasis)。
3.8.1 Top 克隆型占比统计
对每个样本(或指定分组)中 clone size 最大的 top10、top100、top1000、top3000 和 top10000 克隆型进行占比统计分析。通过比较不同细胞群或样本间的 top 克隆型占比,可以反映克隆的扩增或收缩情况。
- Top10 克隆占比高:提示免疫组库呈现寡克隆优势(oligoclonal dominance),少数克隆型主导了免疫应答,常见于急性感染、肿瘤浸润淋巴细胞(TIL)或自身免疫性疾病。
- Top 克隆占比低:提示免疫组库保持高度多样性,克隆分布较为均匀,符合健康个体或初始免疫状态的特征。
 上图展示了不同样本中 top 克隆型的占比情况。横坐标为样本名称,纵坐标为占比(Proportion)。不同颜色的堆叠柱状图分别代表 top10、top100、top1000 等克隆型的累计占比。通过比较可以发现,某些样本的 top10 克隆型占比较高,提示存在显著的克隆扩增;而其他样本则保持相对均匀的克隆分布。
上图展示了不同样本中 top 克隆型的占比情况。横坐标为样本名称,纵坐标为占比(Proportion)。不同颜色的堆叠柱状图分别代表 top10、top100、top1000 等克隆型的累计占比。通过比较可以发现,某些样本的 top10 克隆型占比较高,提示存在显著的克隆扩增;而其他样本则保持相对均匀的克隆分布。
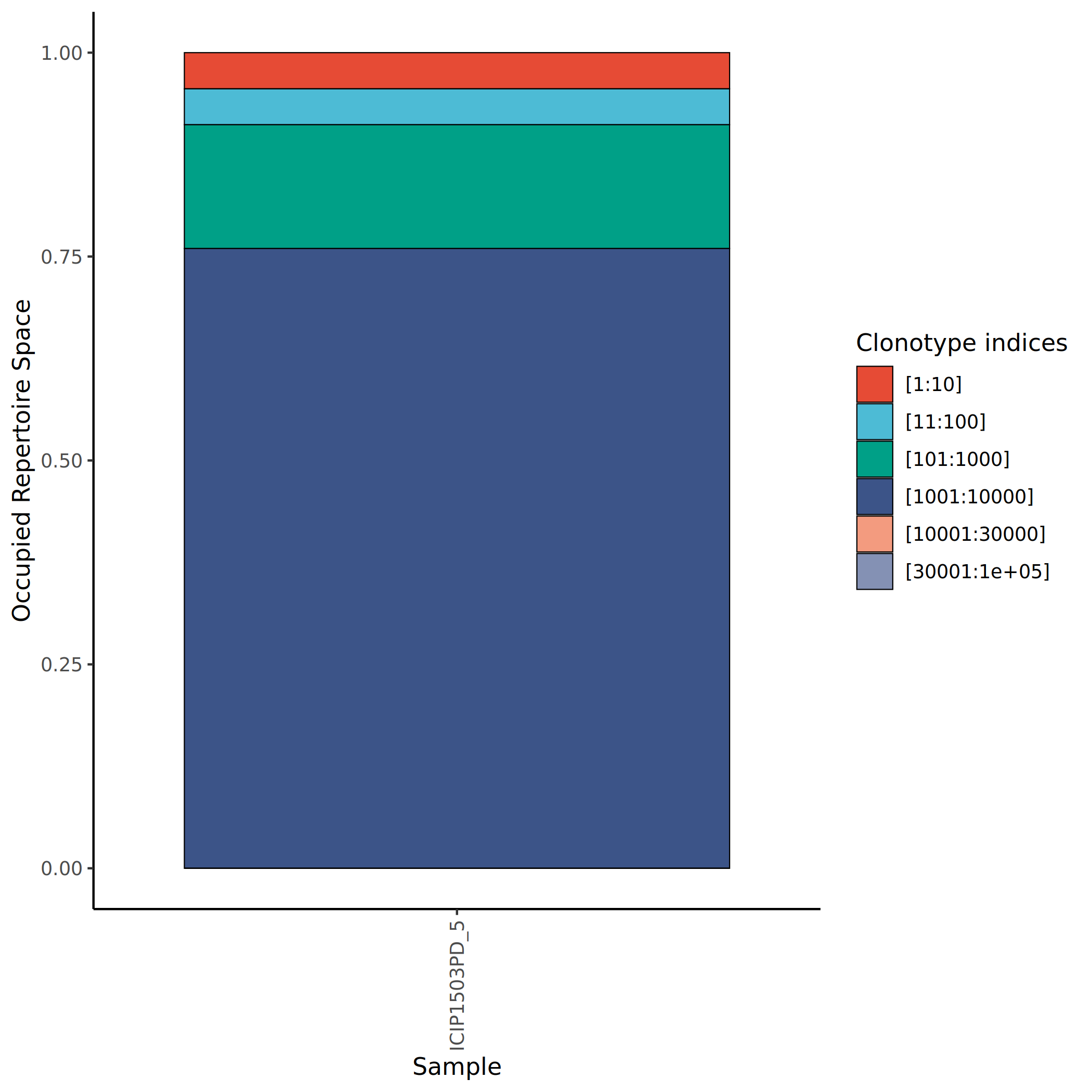
3.8.2 克隆稳态分析(Clonal Homeostasis)
根据每个克隆在样本中的占比,将克隆型分为五个等级,以评估免疫组库的克隆稳态:
- Rare(罕见克隆):占比极低的克隆,通常代表免疫组库的多样性基础。
- Small(小克隆):占比较低的克隆,可能处于初始扩增阶段或维持基础免疫监视功能。
- Medium(中等克隆):占比中等的克隆,可能参与了一定程度的免疫应答。
- Large(大克隆):占比较高的克隆,提示经历了显著的克隆扩增。
- Hyperexpanded(超扩增克隆):占比极高的克隆,通常提示强烈的抗原特异性免疫应答。
克隆占比的计算方法为:某个克隆的 clone size 除以样本中所有克隆的 clone size 总和。
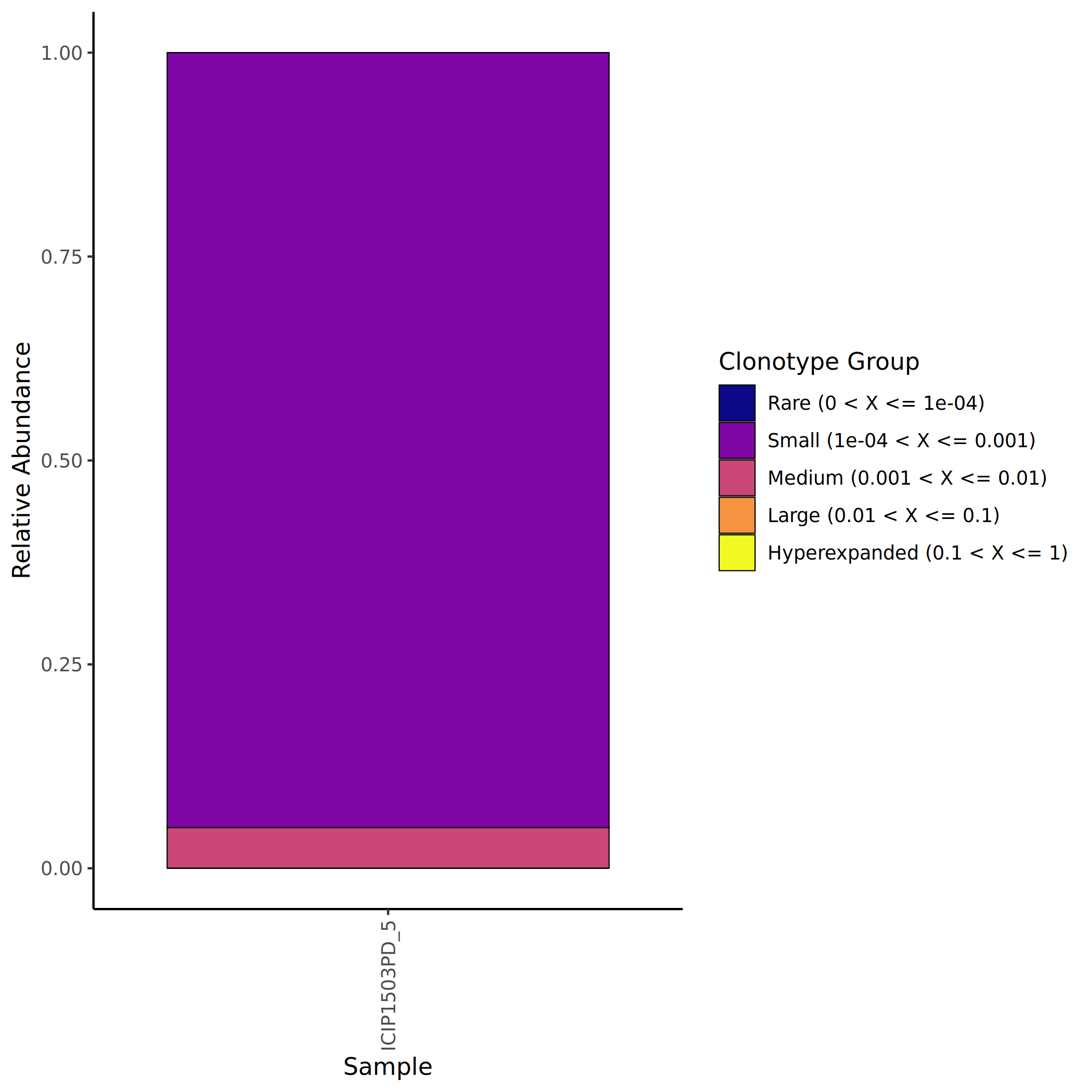 上图展示了不同样本中各克隆级别的占比分布。横坐标为样本名称,纵坐标为占比(Proportion)。不同颜色代表不同的克隆级别(Rare、Small、Medium、Large、Hyperexpanded)。健康样本通常以 Rare 和 Small 克隆为主,而在免疫激活状态下,Large 和 Hyperexpanded 克隆的比例会显著增加。
上图展示了不同样本中各克隆级别的占比分布。横坐标为样本名称,纵坐标为占比(Proportion)。不同颜色代表不同的克隆级别(Rare、Small、Medium、Large、Hyperexpanded)。健康样本通常以 Rare 和 Small 克隆为主,而在免疫激活状态下,Large 和 Hyperexpanded 克隆的比例会显著增加。
3.8.3 克隆型空间映射
将克隆型按照占比分组后映射到转录组 UMAP 图中,可以可视化不同克隆级别在细胞群中的空间分布,帮助识别克隆扩增的细胞亚群和组织定位。
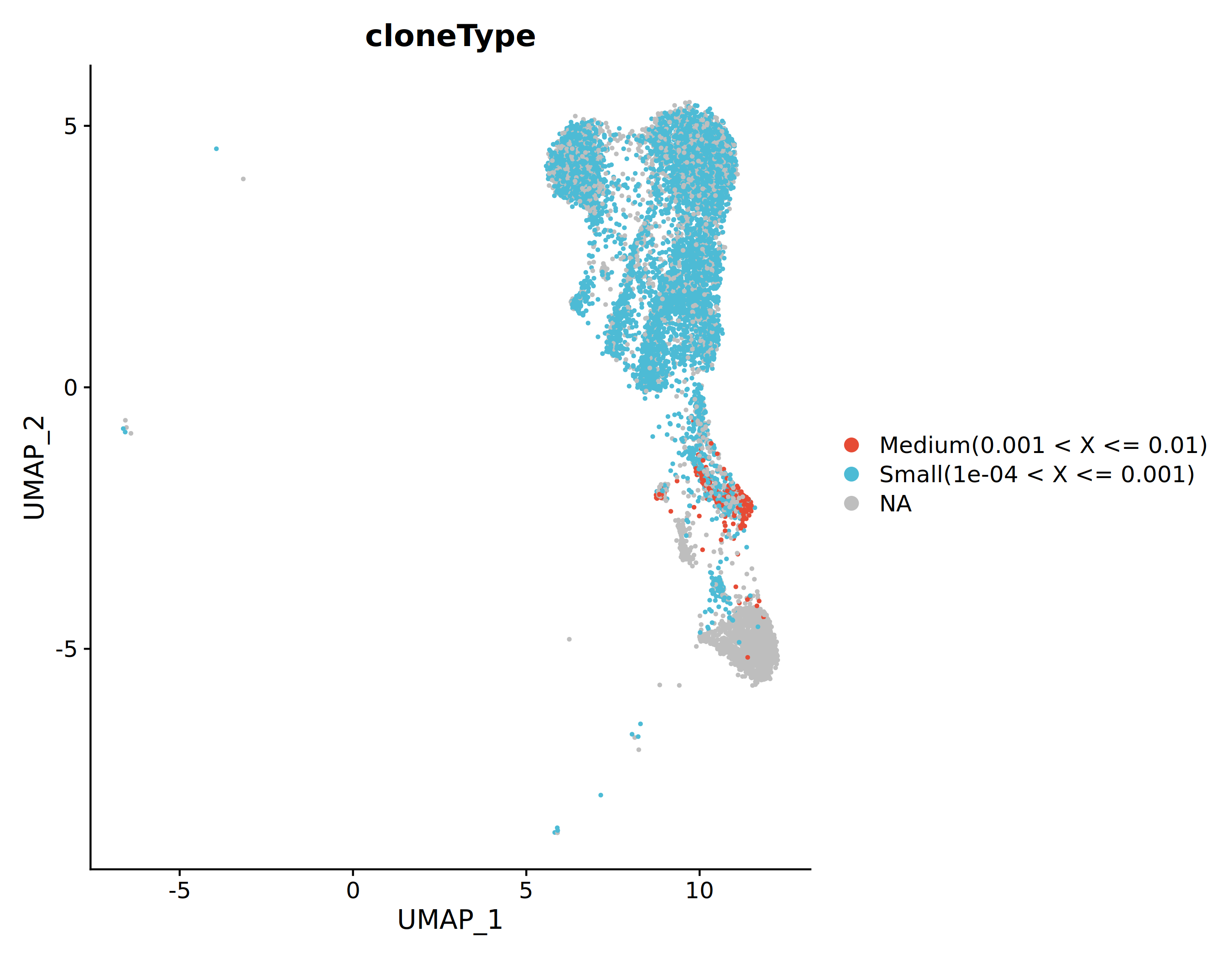 上图将不同克隆级别的细胞映射到 UMAP 空间中。不同颜色代表不同的克隆级别。Hyperexpanded 和 Large 克隆往往聚集在特定的细胞亚群区域,提示这些亚群可能经历了特异性的克隆扩增。
上图将不同克隆级别的细胞映射到 UMAP 空间中。不同颜色代表不同的克隆级别。Hyperexpanded 和 Large 克隆往往聚集在特定的细胞亚群区域,提示这些亚群可能经历了特异性的克隆扩增。
此外,还可以将占比最高的前 5 个克隆单独映射到 UMAP 图中,以更精确地定位优势克隆的分布位置。
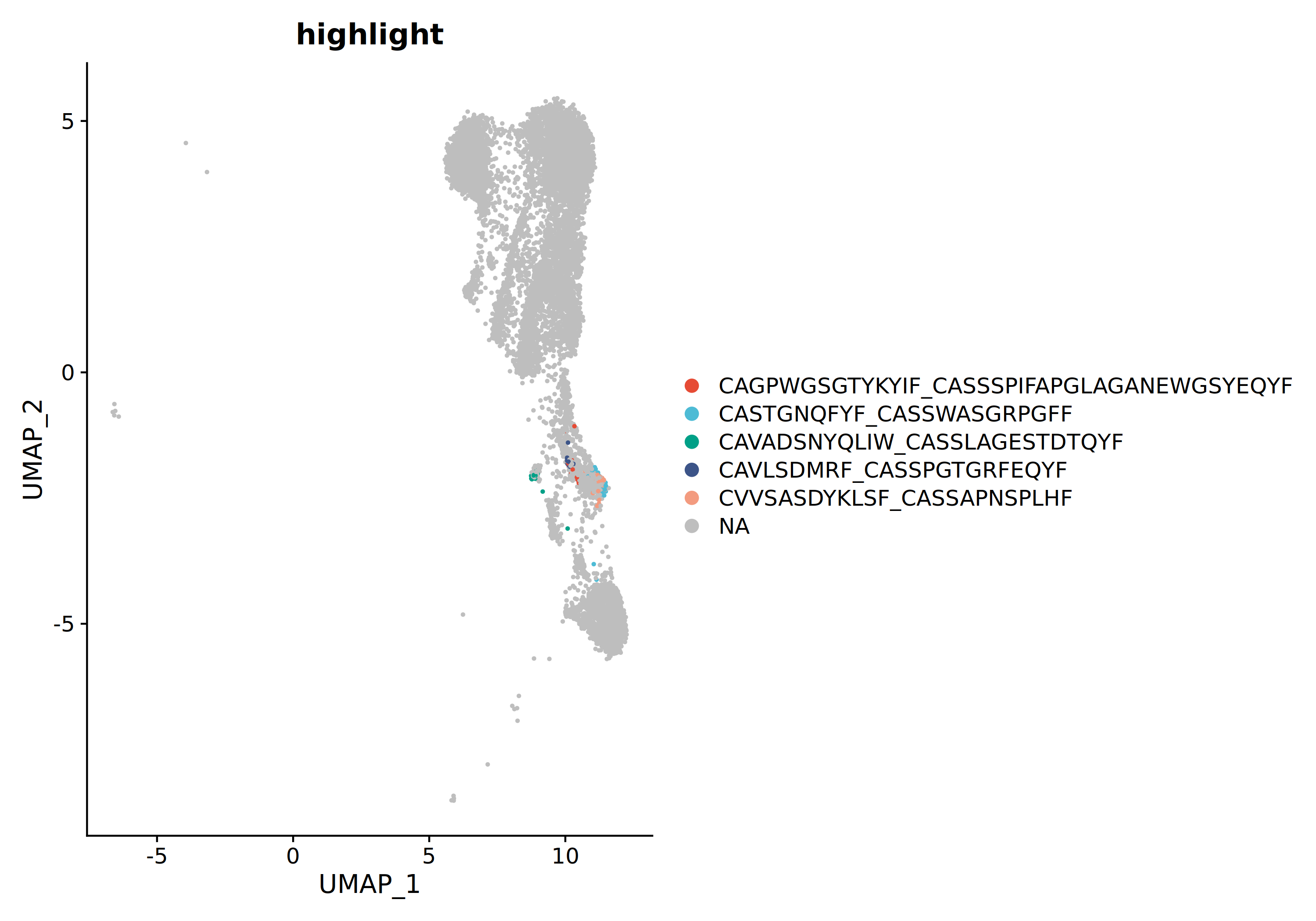 上图展示了占比最高的前 5 个克隆型在 UMAP 空间中的分布。每个克隆型用不同颜色标识。通过观察可以发现,优势克隆可能集中在特定的细胞簇中,反映了克隆特异性的功能状态和组织归巢特性。
上图展示了占比最高的前 5 个克隆型在 UMAP 空间中的分布。每个克隆型用不同颜色标识。通过观察可以发现,优势克隆可能集中在特定的细胞簇中,反映了克隆特异性的功能状态和组织归巢特性。
TIP
结果解读要点:
- Top 克隆型占比可以量化免疫组库的集中度。高占比提示寡克隆优势,低占比提示多样性保持。
- 克隆稳态分析有助于评估免疫系统的激活状态。Hyperexpanded 克隆的增加通常与特异性免疫应答相关。
- 克隆型的空间分布可以揭示克隆扩增与细胞亚群、组织微环境的关联,为理解免疫应答的时空动态提供线索。
- 比较不同样本或治疗前后的克隆占比变化,可以评估治疗效果或疾病进展。
3.9 克隆型桑基图分析
克隆型桑基图(Sankey Diagram)也称为冲积图(Alluvial Plot),是一种流程图类型的可视化工具,通过连接不同层级的节点来展示数据的流动和分布模式。在免疫组库分析中,桑基图可以直观地展示克隆型在不同样本、细胞群之间的分布关系和动态变化,帮助识别克隆型的迁移、扩增或收缩模式。
3.9.1 克隆型桑基图的生物学意义
在单细胞免疫组库分析中,同一克隆型可能分布在不同的细胞亚群或不同的样本/分组中。通过桑基图可以追踪特定克隆型的分布轨迹,从而:
- 揭示克隆型的细胞群归属:了解某个克隆型主要分布在哪些细胞亚群中,反映其功能特性和分化状态。
- 识别共享克隆:发现在多个细胞群或样本间共享的克隆型,提示这些克隆可能具有广泛的抗原反应性或迁移能力。
- 追踪克隆动态变化:在时间序列或治疗前后的样本中,追踪克隆型的扩增或收缩趋势,评估免疫应答的动态演变。
- 发现优势克隆的分布模式:高丰度克隆型在不同细胞群的分布可以提示其在免疫应答中的核心作用。
3.9.2 样本-细胞群-克隆型桑基图
将样本(或分组)、细胞群与克隆型三个层级联系起来,可以全面展示每个样本中各个细胞群的分布情况,以及每个细胞群中各种克隆型的构成信息。图中的流带宽度代表相应克隆型的丰度(细胞数量)。
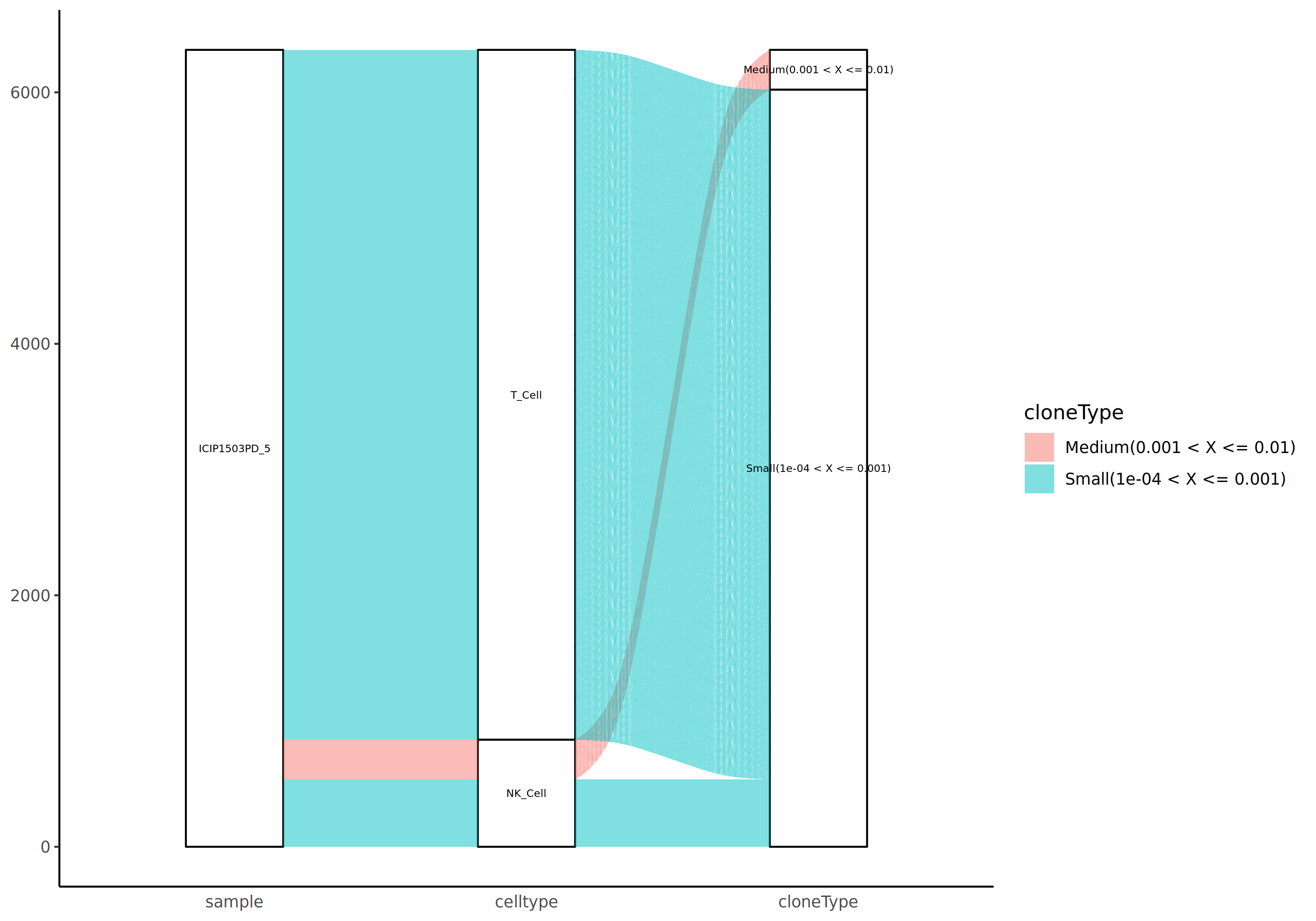 上图展示了从样本到细胞群再到克隆型的三层级桑基图。左侧为样本或分组,中间为细胞亚群,右侧为克隆型分类。流带的宽度表示细胞数量,颜色区分不同的类别。通过该图可以观察到:(1)每个样本中不同细胞群的比例;(2)每个细胞群中克隆型的分布;(3)某些克隆型可能跨越多个细胞群分布,提示其可能具有多功能性或处于不同的分化阶段。
上图展示了从样本到细胞群再到克隆型的三层级桑基图。左侧为样本或分组,中间为细胞亚群,右侧为克隆型分类。流带的宽度表示细胞数量,颜色区分不同的类别。通过该图可以观察到:(1)每个样本中不同细胞群的比例;(2)每个细胞群中克隆型的分布;(3)某些克隆型可能跨越多个细胞群分布,提示其可能具有多功能性或处于不同的分化阶段。
3.9.3 Top5 克隆型桑基图比较分析
为了更精细地研究优势克隆型在不同细胞群间的占比和流动情况,可以提取每个细胞群中占比最高的前 5 个克隆型,通过桑基图直观展示这些优势克隆型在细胞群间的变化趋势和分布模式。
- 克隆特异性分布:某些克隆型仅在特定细胞群中富集,流带集中于单一细胞群,提示其功能特异性。
- 克隆跨群流动:某些克隆型的流带横跨多个细胞群,表明该克隆在不同细胞亚群中均有分布,可能代表具有多重功能或处于分化过渡状态的细胞。
- 克隆丰度变化:流带宽度反映克隆型在各细胞群中的丰度差异,可以识别克隆型的扩增或收缩趋势。
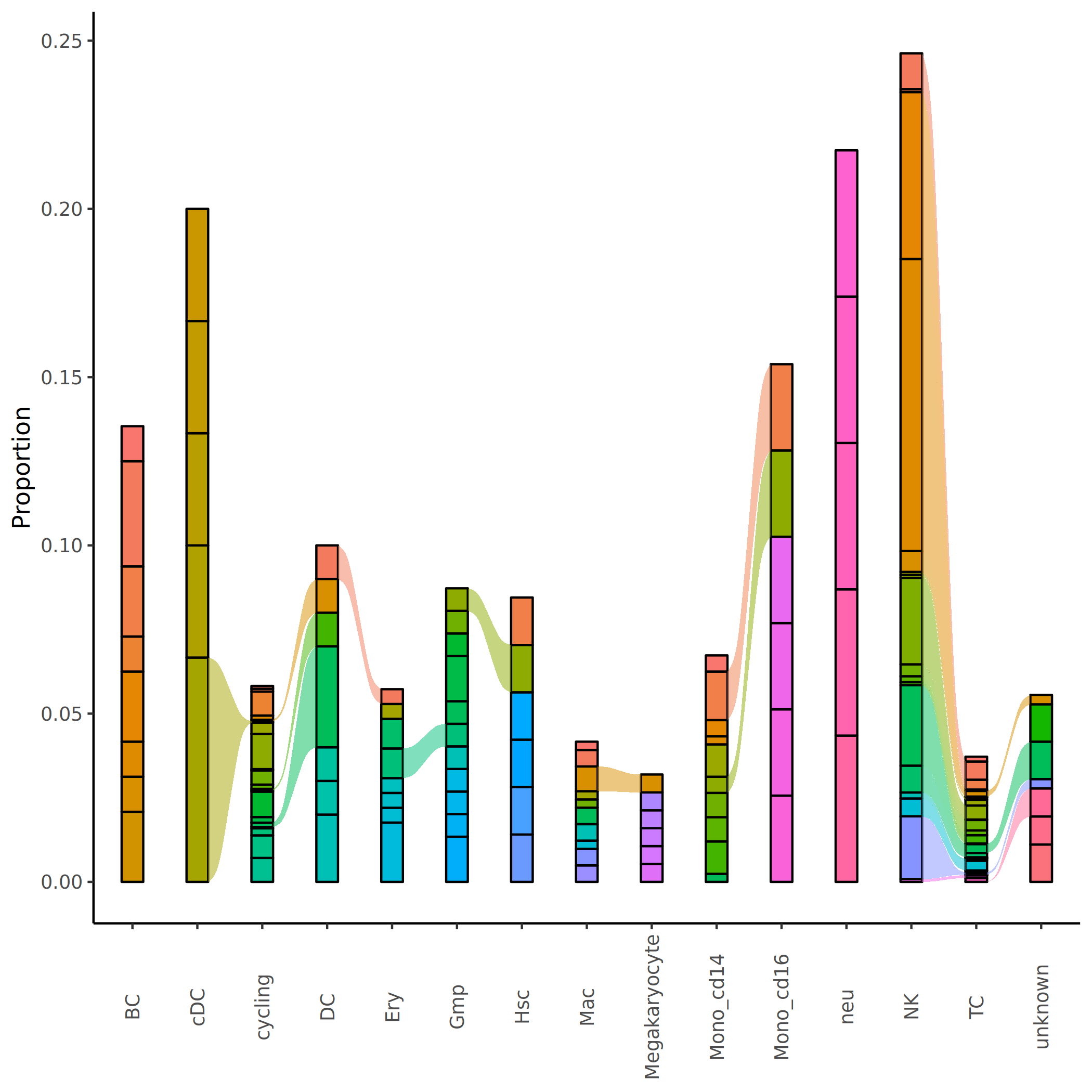 上图以桑基图形式展示了各细胞群中 Top5 克隆型的分布和流动情况。每个细胞群的 Top5 克隆型用不同颜色的流带表示,流带的宽度反映该克隆型在该细胞群中的丰度。通过该图可以直观地观察:(1)每个细胞群的 Top5 克隆型组成;(2)某些高丰度克隆型在多个细胞群中的流动和占比变化(相同颜色流带);(3)不同细胞群的克隆优势特征和特异性,以及克隆型在细胞群间的迁移模式。
上图以桑基图形式展示了各细胞群中 Top5 克隆型的分布和流动情况。每个细胞群的 Top5 克隆型用不同颜色的流带表示,流带的宽度反映该克隆型在该细胞群中的丰度。通过该图可以直观地观察:(1)每个细胞群的 Top5 克隆型组成;(2)某些高丰度克隆型在多个细胞群中的流动和占比变化(相同颜色流带);(3)不同细胞群的克隆优势特征和特异性,以及克隆型在细胞群间的迁移模式。
3.9.4 纵向追踪与动态分析
若研究设计中包含患者信息或时间序列数据,桑基图可以进一步用于分析同一患者在不同时间点或不同组织部位中克隆型的动态变化。这种纵向追踪分析在以下场景中具有重要价值:
- 治疗应答评估:通过比较治疗前后克隆型的组成和丰度变化,可以直观地评估治疗效果。有效治疗通常会导致特定病理性克隆的消退或收缩。
- 疾病进展监测:追踪特定克隆型在不同时间点的扩增、收缩或消失,有助于评估疾病状态的变化和预测疾病转归。
- 组织归巢与迁移研究:对比外周血和组织浸润细胞中的克隆型组成,可以揭示 T/B 细胞的迁移轨迹和组织定位模式,帮助理解免疫细胞的空间分布特征。
TIP
结果解读要点:
- 桑基图的流带宽度代表克隆丰度,宽度越大表示该克隆包含的细胞数量越多。
- 跨越多个细胞群的克隆型可能具有多功能性或代表处于过渡状态的细胞群。
- 高度集中在单一细胞群的克隆型提示其功能特异性和终末分化状态。
- 通过比较不同样本或时间点的桑基图,可以识别克隆扩增、收缩或细胞群转换事件。
- 优势克隆的分布模式可以提示免疫应答的主导方向和关键细胞亚群。
3.10 克隆型共有与特有分析
克隆型的共有(shared)与特有(unique)分析是评估不同细胞群或样本间克隆型组成相似性和差异性的重要方法。该分析有助于识别细胞群特异性的克隆型,以及在多个细胞群或样本间共享的克隆型,从而揭示免疫应答的广度和特异性。
3.10.1 生物学意义
- 特有克隆(Unique Clones):仅在单一细胞群或样本中出现的克隆型,代表该细胞群的特异性免疫反应或独特的抗原识别能力。特有克隆的比例反映了细胞群的免疫学独立性。
- 共有克隆(Shared Clones):在两个或多个细胞群/样本间共同存在的克隆型,提示这些克隆可能:
- 识别共同的抗原表位
- 在细胞群间发生迁移或分化转换
- 代表记忆性或长期存在的免疫反应
- 克隆重叠度(Clone Overlap):衡量不同细胞群或样本间克隆型组成的相似程度,可以评估免疫组库的聚集性或离散性。
3.10.2 UpSet 图展示共有克隆
当需要比较3个以上细胞群或样本间的克隆型交集时,传统的韦恩图(Venn Diagram)会变得复杂难读。UpSet 图是一种更清晰的可视化方法,特别适合多集合的交集分析。
UpSet 图的组成部分:
- 左侧水平柱状图:展示每个细胞群的总克隆型数量(Set Size),反映各细胞群的克隆多样性。
- 底部矩阵图:用点和连线表示细胞群的组合。单个点表示特有克隆,多个点通过连线表示共有克隆。
- 上方垂直柱状图:显示每种组合(特有或共有)中的克隆型数量(Intersection Size),柱子高度代表克隆数量。
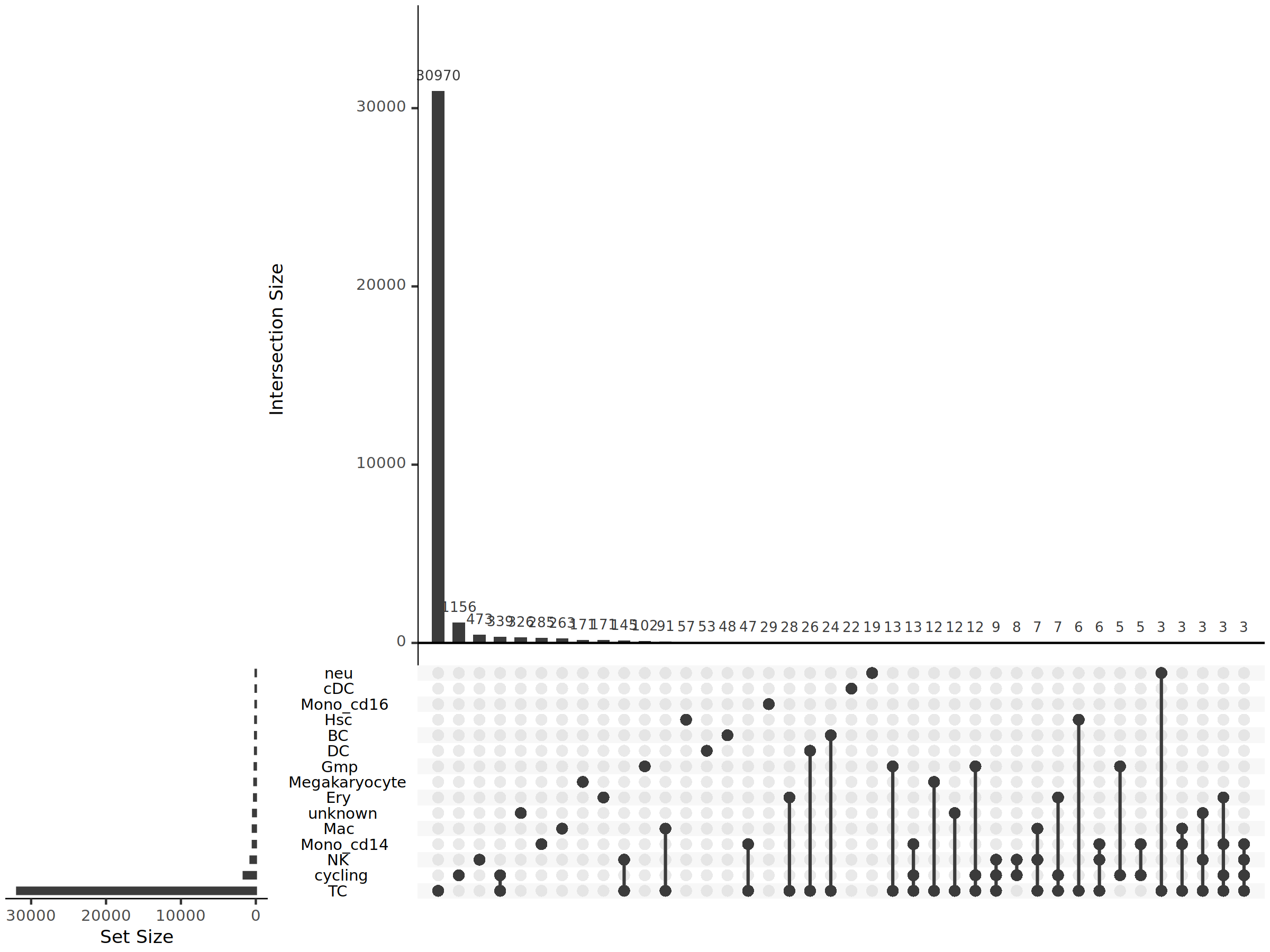 上图为 UpSet 图,用于展示不同细胞群间克隆型的共有与特有关系。左侧水平柱表示各细胞群的总克隆型数;底部矩阵中,每一列代表一种细胞群组合,单个点表示该细胞群特有的克隆,多个点通过竖线连接表示这些细胞群共享的克隆;上方垂直柱的高度表示每种组合中的克隆型数量。通过该图可以一目了然地观察:(1)哪些细胞群具有最多的特有克隆;(2)哪些细胞群组合共享最多的克隆;(3)共有克隆的分布模式和细胞群间的关联性。
上图为 UpSet 图,用于展示不同细胞群间克隆型的共有与特有关系。左侧水平柱表示各细胞群的总克隆型数;底部矩阵中,每一列代表一种细胞群组合,单个点表示该细胞群特有的克隆,多个点通过竖线连接表示这些细胞群共享的克隆;上方垂直柱的高度表示每种组合中的克隆型数量。通过该图可以一目了然地观察:(1)哪些细胞群具有最多的特有克隆;(2)哪些细胞群组合共享最多的克隆;(3)共有克隆的分布模式和细胞群间的关联性。
3.10.3 特有克隆型数量统计
统计每个细胞群中全部克隆型的数量,可以评估各细胞群的克隆多样性。横坐标为细胞群名称,纵坐标为该细胞群包含的克隆型总数(包括特有和共有克隆)。
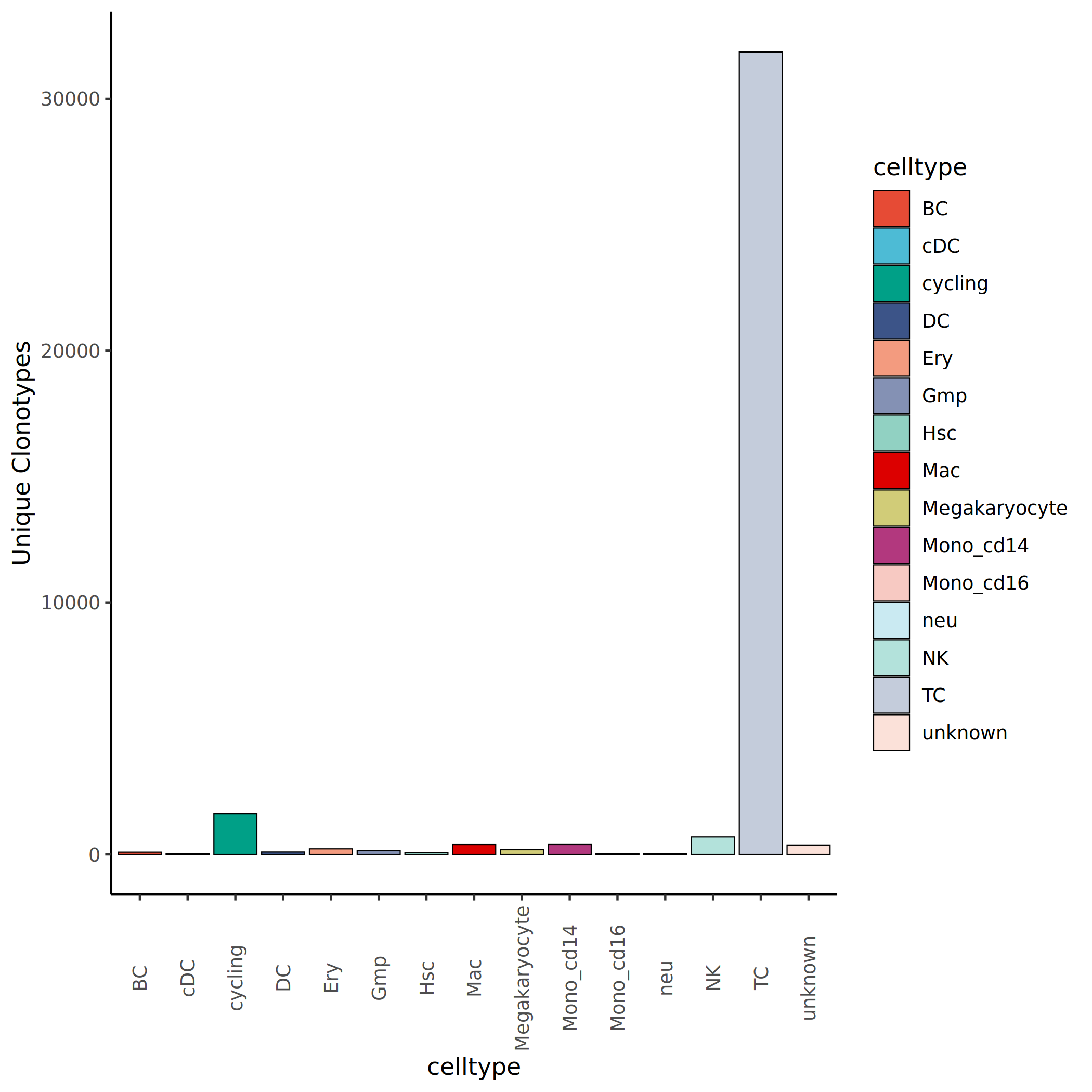 上图展示了各细胞群中克隆型的总数量。横坐标为细胞群名称,纵坐标为克隆型数目。可以看出,不同细胞群的克隆型数量存在差异,这与细胞数量、克隆扩增程度和免疫多样性有关。克隆型数量较多的细胞群通常具有更高的免疫多样性,而克隆型数量较少但克隆丰度高的细胞群则提示存在寡克隆扩增。
上图展示了各细胞群中克隆型的总数量。横坐标为细胞群名称,纵坐标为克隆型数目。可以看出,不同细胞群的克隆型数量存在差异,这与细胞数量、克隆扩增程度和免疫多样性有关。克隆型数量较多的细胞群通常具有更高的免疫多样性,而克隆型数量较少但克隆丰度高的细胞群则提示存在寡克隆扩增。
3.10.4 特有克隆型占比分析
计算每个细胞群中特有克隆型的占比(scaled = contigs/total),即该细胞群特有的克隆型数量占其总克隆型数量的比例。高占比提示该细胞群具有独特的克隆型库,低占比则表明该细胞群与其他细胞群共享较多克隆型。
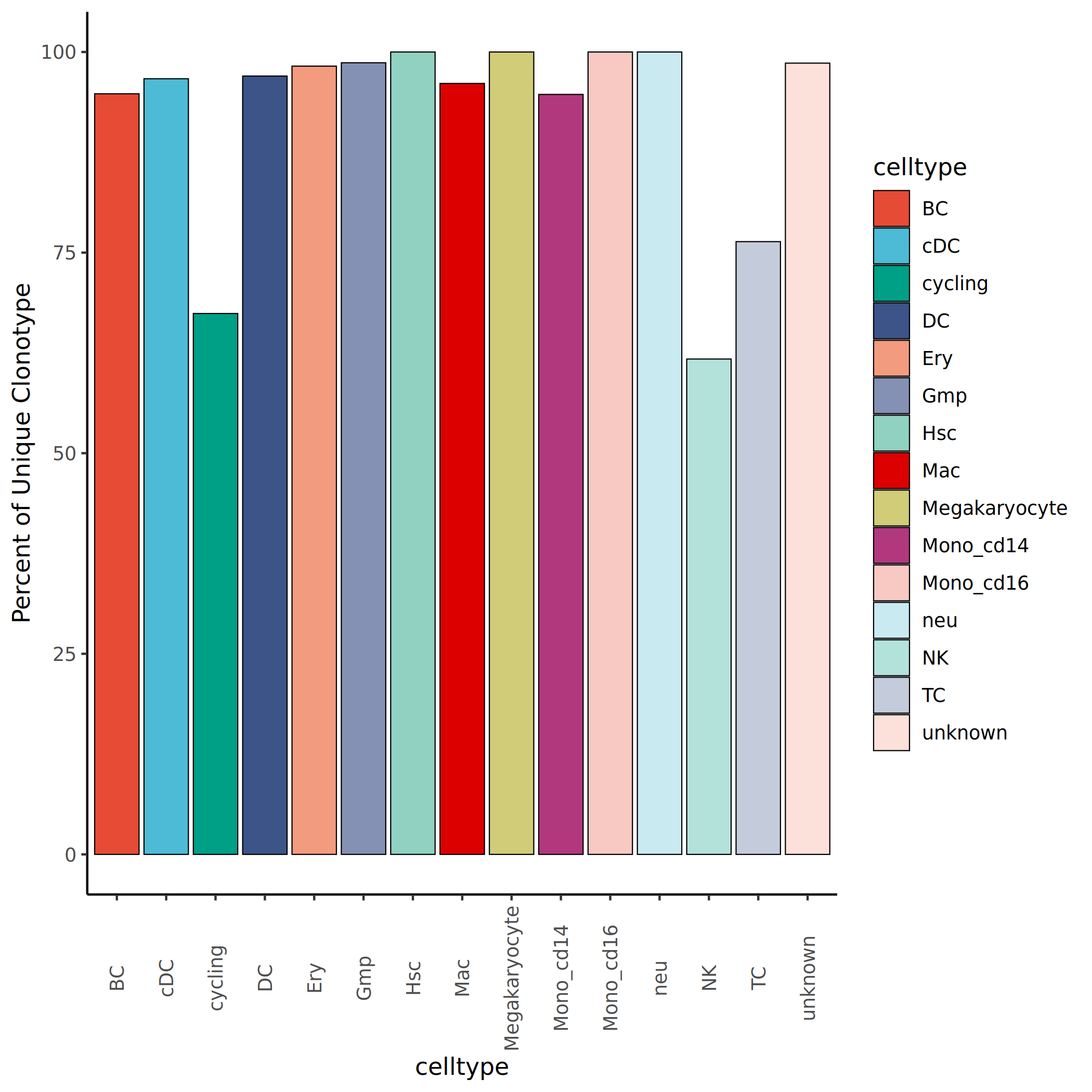 上图展示了各细胞群中特有克隆型的占比。横坐标为细胞群名称,纵坐标为特有克隆型占比(百分比)。较高的特有克隆占比提示该细胞群具有独特的免疫反应性和功能特异性;较低的占比则表明该细胞群与其他细胞群共享较多克隆型,可能存在细胞群间的克隆迁移或共同的抗原靶点。
上图展示了各细胞群中特有克隆型的占比。横坐标为细胞群名称,纵坐标为特有克隆型占比(百分比)。较高的特有克隆占比提示该细胞群具有独特的免疫反应性和功能特异性;较低的占比则表明该细胞群与其他细胞群共享较多克隆型,可能存在细胞群间的克隆迁移或共同的抗原靶点。
3.10.5 克隆型重叠分析(Morisita Overlap Index)
Morisita 重叠指数(Morisita Overlap Index)是一种统计度量方法,用于衡量不同群体间个体分布的相似程度。在免疫组库分析中,该指数可以评估不同细胞群或样本间克隆型组成的重叠程度。
- 指数范围:0 到 1。
- 接近 1:表示两个细胞群的克隆型组成高度相似,克隆型重叠度高。
- 接近 0:表示两个细胞群的克隆型组成差异很大,克隆型重叠度低。
- 生物学解释:
- 高重叠度提示细胞群可能来源于共同的祖细胞、识别相似的抗原、或存在频繁的细胞迁移和交流。
- 低重叠度提示细胞群具有独立的克隆库和不同的抗原特异性,反映功能分化和免疫应答的多样性。
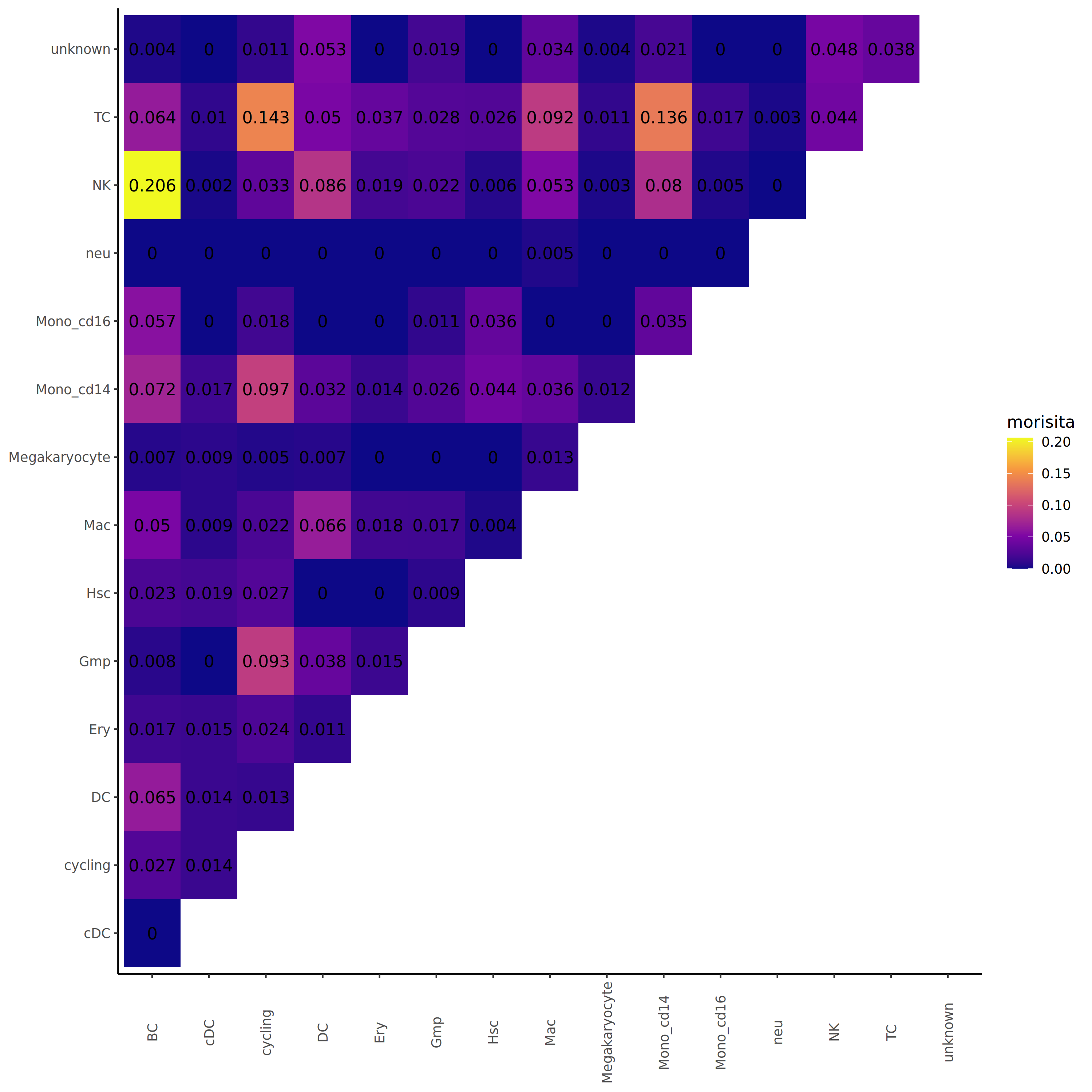 上图以热图形式展示了不同细胞群间的 Morisita 重叠指数。横纵坐标均为细胞群名称,颜色代表重叠指数。颜色越亮(接近黄色)表示两个细胞群间的克隆型重叠度越高,克隆组成越相似;颜色越暗(接近紫色)表示重叠度越低,克隆组成差异越大。通过该图可以识别哪些细胞群具有相似的克隆型库,哪些细胞群则保持独立的免疫特征。
上图以热图形式展示了不同细胞群间的 Morisita 重叠指数。横纵坐标均为细胞群名称,颜色代表重叠指数。颜色越亮(接近黄色)表示两个细胞群间的克隆型重叠度越高,克隆组成越相似;颜色越暗(接近紫色)表示重叠度越低,克隆组成差异越大。通过该图可以识别哪些细胞群具有相似的克隆型库,哪些细胞群则保持独立的免疫特征。
TIP
结果解读要点:
- 特有克隆型占比高的细胞群通常具有独特的功能特性和抗原特异性,反映其在免疫应答中的特殊角色。
- 共有克隆型的存在提示细胞群间可能存在克隆迁移、共同抗原识别或发育谱系关系。
- UpSet 图适合多组比较,能清晰展示复杂的克隆共享关系,优于传统韦恩图。
- Morisita 重叠指数可以量化细胞群间的克隆组成相似性,高重叠度提示功能相关或发育连续性。
- 通过比较健康对照和疾病样本的克隆重叠度,可以识别疾病相关的克隆扩增模式。
- 在纵向研究中,追踪共有克隆的动态变化可以评估治疗效果和疾病转归。
3.11 克隆扩增与克隆收缩分析
克隆扩增(Clonal Expansion)与克隆收缩(Clonal Contraction)是免疫应答过程中T/B细胞克隆型动态变化的重要特征。通过比较不同样本或时间点的克隆型组成,可以识别哪些克隆发生了扩增、收缩或新出现,从而揭示免疫应答的动态演变过程。
3.11.1 生物学意义
T/B细胞克隆型的扩增和收缩在免疫学和疾病研究中具有重要意义:
- 感染应答:当机体遭遇病原体时,特定的T/B细胞克隆型会被激活并大量扩增,以应对感染。这种扩增和收缩的动态过程有助于清除病原体并恢复免疫平衡。
- 疾病诊断与监测:某些疾病(如肿瘤、自身免疫病)的发展可能伴随着特定克隆型的异常扩增或缩减。通过检测和分析克隆型的变化,可以帮助诊断疾病、评估疾病进展以及监测治疗效果。
- 治疗效果评估:在免疫治疗(如CAR-T、免疫检查点抑制剂)过程中,追踪特定克隆型的扩增或收缩可以评估治疗应答和预测预后。
- 免疫记忆形成:克隆扩增后的收缩期会保留部分记忆性细胞,这些长期存在的克隆为再次应答提供基础。
总之,克隆型的扩增和收缩对于免疫应答、免疫记忆、免疫监测和免疫治疗等方面都具有重要意义。对这些过程的研究有助于深入了解免疫系统的功能和疾病的发展机制,为免疫相关疾病的预防、诊断和治疗提供指导和策略。
3.11.2 克隆扩增与收缩散点图
通过比较两个样本(或两个时间点)之间克隆型的频率变化,可以识别三类克隆型:
- Novel clones(新出现克隆):在样本B中新检出但在样本A中未检出的克隆型(Fisher's exact p < 0.05),代表新激活或新募集的免疫细胞克隆。
- Expanded clones(扩增克隆):在样本B中的频率显著高于样本A的克隆型(Fisher's exact p < 0.05),提示该克隆经历了特异性扩增,可能与抗原刺激或免疫激活相关。
- Contracted clones(收缩克隆):在样本B中的频率显著低于样本A的克隆型(Fisher's exact p < 0.05),表明该克隆发生了收缩或凋亡,可能反映免疫应答的消退或细胞迁移。
- Persistent clones(持续存在克隆):在两个样本间频率没有显著差异的克隆型(Fisher's exact p > 0.05),表明该克隆在两个样本中维持相对稳定的丰度,可能代表基线免疫监视或长期存在的记忆性克隆。
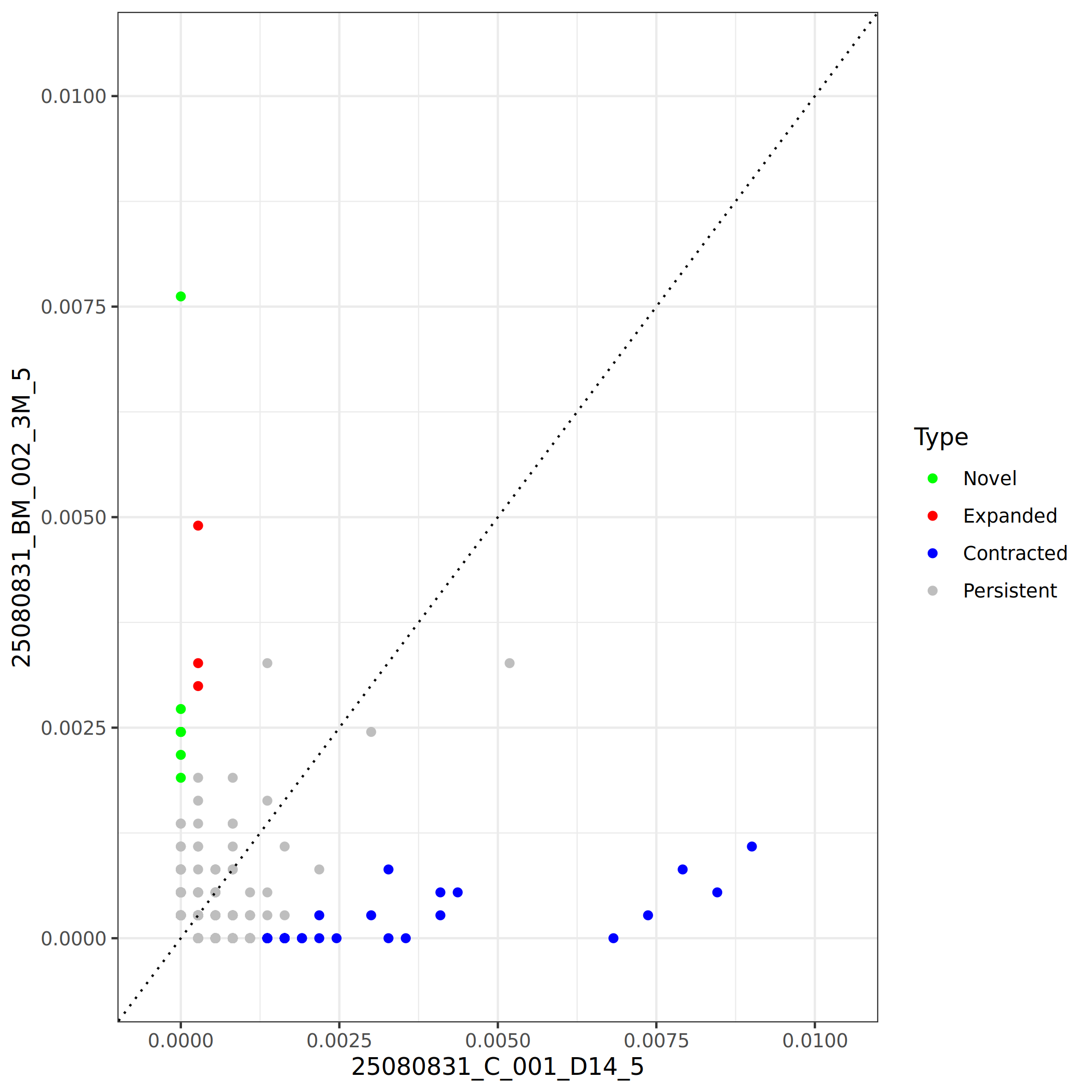 上图展示了两个样本间克隆型的扩增与收缩分析。每个点代表一个克隆型,横坐标为该克隆型在样本A中的频率,纵坐标为其在样本B中的频率。不同颜色代表不同的克隆类型:绿色为Novel clones(新出现克隆)、红色为Expanded clones(扩增克隆)、蓝色为Contracted clones(收缩克隆)、灰色为Persistent clones(持续存在克隆,无显著变化)。对角线表示两个样本中频率相等。偏离对角线越远的点表示变化越显著。通过该图可以直观地识别哪些克隆发生了显著的扩增或收缩,以及新出现的克隆数量和频率。
上图展示了两个样本间克隆型的扩增与收缩分析。每个点代表一个克隆型,横坐标为该克隆型在样本A中的频率,纵坐标为其在样本B中的频率。不同颜色代表不同的克隆类型:绿色为Novel clones(新出现克隆)、红色为Expanded clones(扩增克隆)、蓝色为Contracted clones(收缩克隆)、灰色为Persistent clones(持续存在克隆,无显著变化)。对角线表示两个样本中频率相等。偏离对角线越远的点表示变化越显著。通过该图可以直观地识别哪些克隆发生了显著的扩增或收缩,以及新出现的克隆数量和频率。
3.11.3 应用场景
克隆扩增与收缩分析在以下研究场景中具有重要应用价值:
- 纵向监测:追踪同一患者在不同治疗阶段或疾病进展过程中的克隆动态,评估疾病状态和治疗效果。
- 样本比较:比较不同组织部位(如外周血vs肿瘤组织)的克隆组成差异,揭示细胞归巢和迁移模式。
- 治疗前后对比:识别治疗相关的克隆扩增(如CAR-T细胞扩增)或肿瘤相关克隆的消退,评估治疗应答。
- 疫苗研究:监测疫苗接种后特异性克隆的扩增动态,评估疫苗免疫原性和持久性。
TIP
结果解读要点:
- 大量扩增克隆提示存在强烈的免疫激活或抗原刺激,可能与感染、疫苗接种或免疫治疗相关。
- 新出现的克隆可能来自新募集的免疫细胞或初始T/B细胞的激活。
- 收缩克隆可能反映免疫应答的消退、细胞凋亡或从该组织/样本中迁出。
- 显著扩增的克隆型值得进一步分析其CDR3序列和抗原特异性,可能是疾病特征性克隆。
- 结合临床信息解读克隆动态变化,如治疗应答者通常表现为病理性克隆的收缩和免疫效应克隆的扩增。
3.12 克隆多样性分析
克隆多样性(Clonal Diversity)是评估免疫组库复杂程度和健康状态的重要指标。高多样性的免疫组库通常意味着更广泛的抗原识别能力和更强的免疫应答潜能,而多样性降低可能提示免疫衰老、免疫缺陷或克隆性疾病。
3.12.1 多样性指数简介
免疫组库的多样性分析通常涉及多个多样性指数,每个指数从不同角度描述克隆型的分布特征:
- Shannon指数:综合考虑克隆型的丰富度(数量)和均匀度(分布),值越大表示多样性越高。
- Simpson指数:衡量随机选择两个细胞时它们属于同一克隆型的概率,值越小表示多样性越高。
- Inverse Simpson指数:Simpson指数的倒数,值越大表示多样性越高,更直观地反映多样性水平。
- Chao1指数:估计样本中克隆型的总数(包括未观察到的稀有克隆),用于评估克隆丰富度。
- Gini指数:衡量克隆型分布的不均匀程度,值越大表示克隆越集中于少数优势克隆。
在本分析中,我们使用 Immunarch 软件对样本的多样性指标进行了计算和可视化。
3.12.2 Inverse Simpson指数分析
Inverse Simpson指数(反Simpson指数)是Simpson指数的倒数,其取值范围从1到无穷大。该指数越大,表示克隆多样性越高,样本中包含更多不同的克隆型;反之,指数越小,则表示克隆多样性越低,样本中的克隆型更为单一或存在寡克隆优势。
Inverse Simpson指数的优势在于其对优势克隆型的敏感性较高,能够有效捕捉免疫组库中克隆扩增导致的多样性下降。
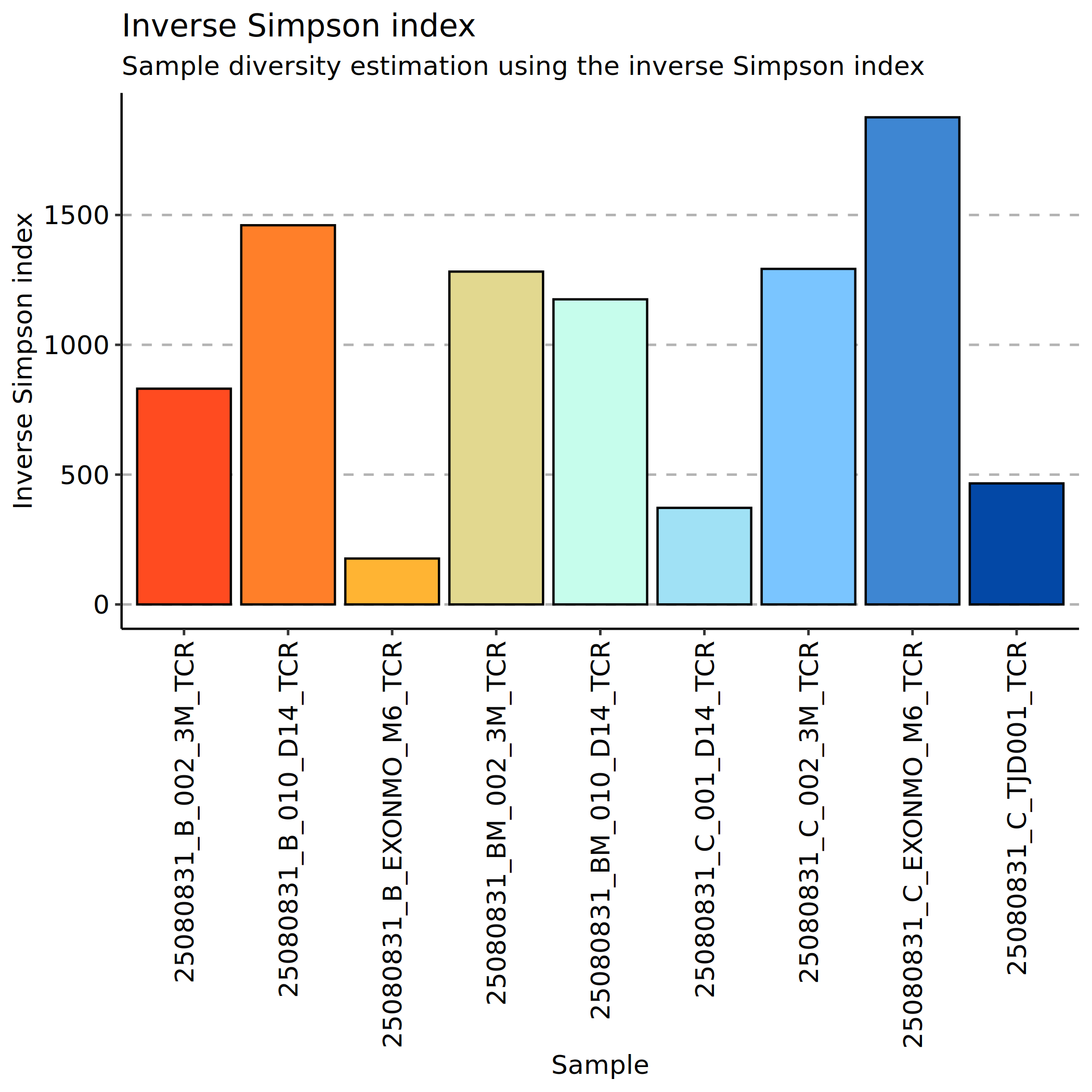 上图展示了各样本的 Inverse Simpson 指数。横坐标为样本名称,纵坐标为 Inverse Simpson 指数值。每个点代表一个样本,柱状图的高度反映该样本的克隆多样性水平。指数值越高表示该样本的克隆多样性越高,反映克隆型种类丰富且分布相对均衡;指数值越低则表示多样性降低,存在明显的寡克隆优势,少数克隆型占据主导地位。通过比较不同样本或组间的多样性差异,可以评估疾病状态、治疗效果或免疫系统功能。
上图展示了各样本的 Inverse Simpson 指数。横坐标为样本名称,纵坐标为 Inverse Simpson 指数值。每个点代表一个样本,柱状图的高度反映该样本的克隆多样性水平。指数值越高表示该样本的克隆多样性越高,反映克隆型种类丰富且分布相对均衡;指数值越低则表示多样性降低,存在明显的寡克隆优势,少数克隆型占据主导地位。通过比较不同样本或组间的多样性差异,可以评估疾病状态、治疗效果或免疫系统功能。
3.12.3 Chao1指数分析
Chao1指数是一种常用的评估样本克隆丰富度的方法。它基于样本中观察到的克隆型数量和它们的出现频率,通过统计学方法估计未观察到的稀有克隆数量,从而推断整个样本的克隆丰富度。
Chao1指数的值越大,表示估计的克隆型总数越多,克隆丰富度越高。该方法的优点是能够考虑到稀有克隆的存在(如仅出现一次或两次的克隆),对于评估免疫组库的潜在多样性具有重要价值。
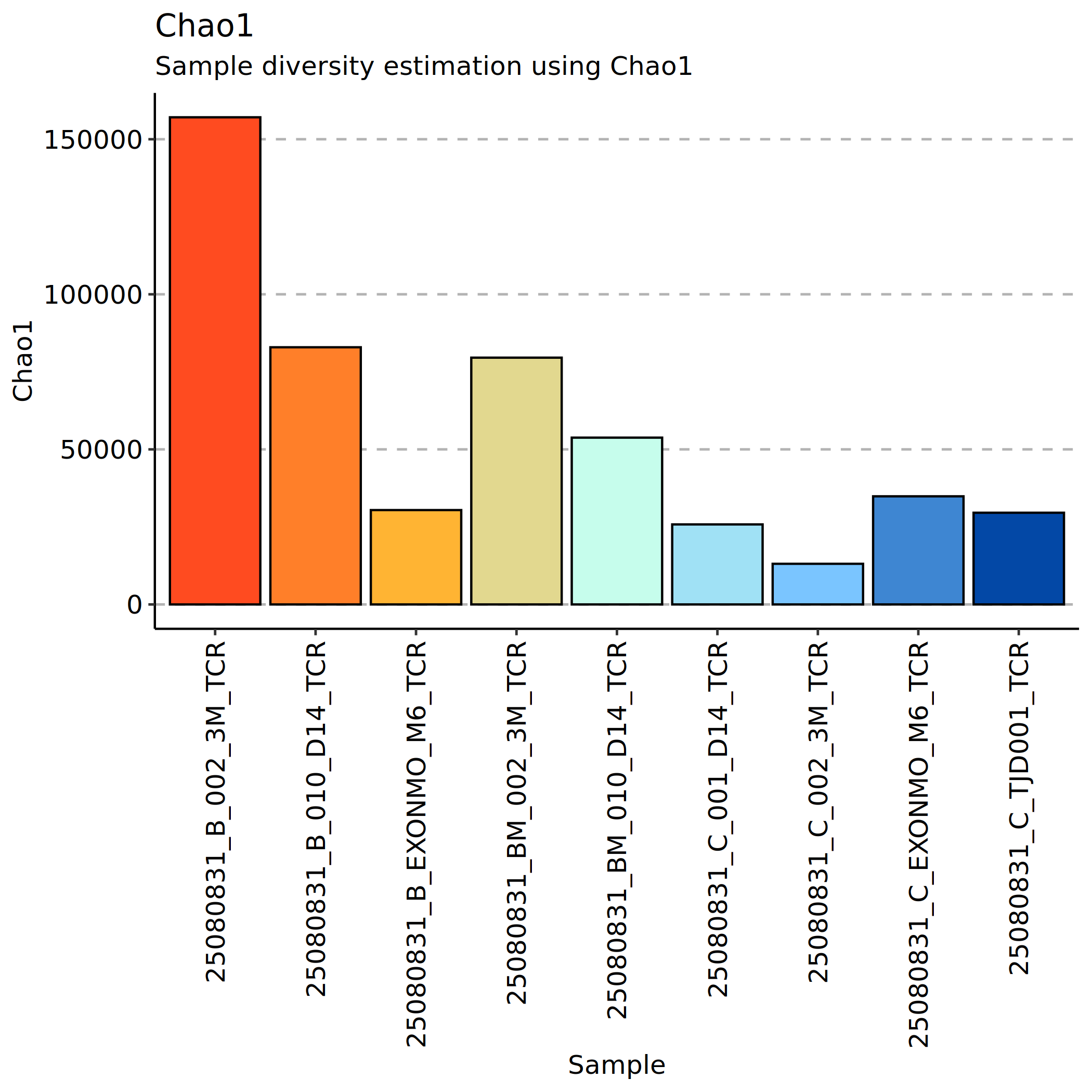 上图展示了各样本的 Chao1 指数。横坐标为样本名称,纵坐标为 Chao1 指数值。每个点代表一个样本的估计克隆丰富度。Chao1 指数越高,表示该样本中克隆型的总数(包括稀有克隆)越多,免疫组库越丰富。该指数对测序深度和稀有克隆的捕捉较为敏感,可用于评估样本采集的充分性和免疫组库的复杂程度。
上图展示了各样本的 Chao1 指数。横坐标为样本名称,纵坐标为 Chao1 指数值。每个点代表一个样本的估计克隆丰富度。Chao1 指数越高,表示该样本中克隆型的总数(包括稀有克隆)越多,免疫组库越丰富。该指数对测序深度和稀有克隆的捕捉较为敏感,可用于评估样本采集的充分性和免疫组库的复杂程度。
3.12.4 多样性指数的生物学意义
- 健康评估:健康个体通常具有较高的克隆多样性,能够应对多样化的抗原挑战。多样性降低可能提示免疫衰老、免疫抑制或免疫缺陷。
- 疾病诊断:某些疾病(如白血病、淋巴瘤)表现为特定克隆的异常扩增,导致多样性显著下降。多样性指数的异常可作为疾病诊断的辅助指标。
- 治疗监测:免疫治疗(如PD-1抑制剂)有效时,通常会观察到克隆多样性的增加或恢复,反映免疫系统功能的改善。
- 预后评估:某些研究表明,较高的克隆多样性与更好的治疗应答和预后相关,可作为预测性生物标志物。
TIP
结果解读要点:
- Inverse Simpson 指数和 Chao1 指数是互补的:Inverse Simpson侧重评估克隆分布的均衡性(是否存在优势克隆),对克隆扩增敏感;Chao1侧重估计克隆的种类总数(包括稀有克隆),反映免疫组库的丰富程度。两者结合可全面评估免疫组库的复杂度和健康状态。
- 多样性指数的解读需要结合临床背景:例如,感染急性期可能出现特异性克隆扩增导致多样性暂时下降,这是正常免疫应答的表现。
- 不同样本类型的多样性基线不同:外周血通常比组织浸润细胞具有更高的多样性。
- 纵向监测多样性变化比单次测量更有意义,可以揭示免疫状态的动态演变。
- 结合其他指标(如克隆扩增、共有克隆)综合分析,可以更全面地理解免疫组库的特征和功能状态。
- 低多样性并非总是病理性的:在特定抗原刺激下,正常的免疫应答也会表现为相关克隆的扩增和整体多样性的下降。
3.13 细胞亚型TCR抗原表位注释分析(TCR)
TCR抗原表位注释分析是通过将检测到的TCR序列与已知的抗原特异性TCR数据库进行比对,从而推断TCR可能识别的抗原表位及其相关的疾病或病理状态。这种分析有助于理解T细胞免疫应答的靶向性,为疾病诊断、免疫治疗和疫苗设计提供重要线索。
3.13.1 生物学意义
TCR抗原表位注释在免疫学研究中具有重要的应用价值:
- 疾病特异性T细胞识别:通过注释TCR序列,可以识别与特定疾病(如肿瘤、病毒感染、自身免疫病)相关的T细胞克隆,揭示疾病特异性免疫应答特征。
- 免疫治疗靶点发现:识别肿瘤特异性或肿瘤相关抗原的TCR,为TCR-T细胞疗法、个性化疫苗等免疫治疗策略提供候选靶点。
- 感染性疾病监测:检测与特定病原体(如病毒、细菌)相关的TCR,评估机体对感染的免疫应答状态和记忆性免疫的建立。
- 自身免疫病机制研究:识别识别自身抗原的TCR,有助于理解自身免疫病的发病机制和组织特异性损伤模式。
- 疫苗效果评估:通过追踪疫苗相关抗原特异性TCR的扩增和持久性,评估疫苗的免疫原性和保护效果。
3.13.2 McPAS-TCR数据库
McPAS-TCR(Manual Curation of Pathology Associated T cell Receptor Sequences)是一个人工策展的病理相关TCR序列数据库,收录了大量经过实验验证的、与特定抗原和疾病相关的TCR序列。该数据库涵盖了:
- 多种疾病类型:包括癌症、病毒感染(如HIV、CMV、EBV、流感等)、自身免疫病、细菌感染等。
- 抗原信息:记录了TCR识别的具体抗原表位序列、抗原来源和MHC限制性。
- TCR序列信息:包含TCR的α链和β链CDR3序列、V/J基因使用情况等详细信息。
- 实验验证依据:所有条目均来自已发表的文献,具有明确的实验证据支持。
通过将样本中检测到的TCR序列与McPAS-TCR数据库进行比对,可以注释出潜在的抗原特异性TCR,并推断其可能参与的病理过程。
3.13.3 疾病相关TCR注释结果
通过与McPAS-TCR数据库比对,可以统计样本中检测到的与不同疾病相关的TCR数量。分析结果展示了各样本中注释到的病理相关TCR的分布情况。
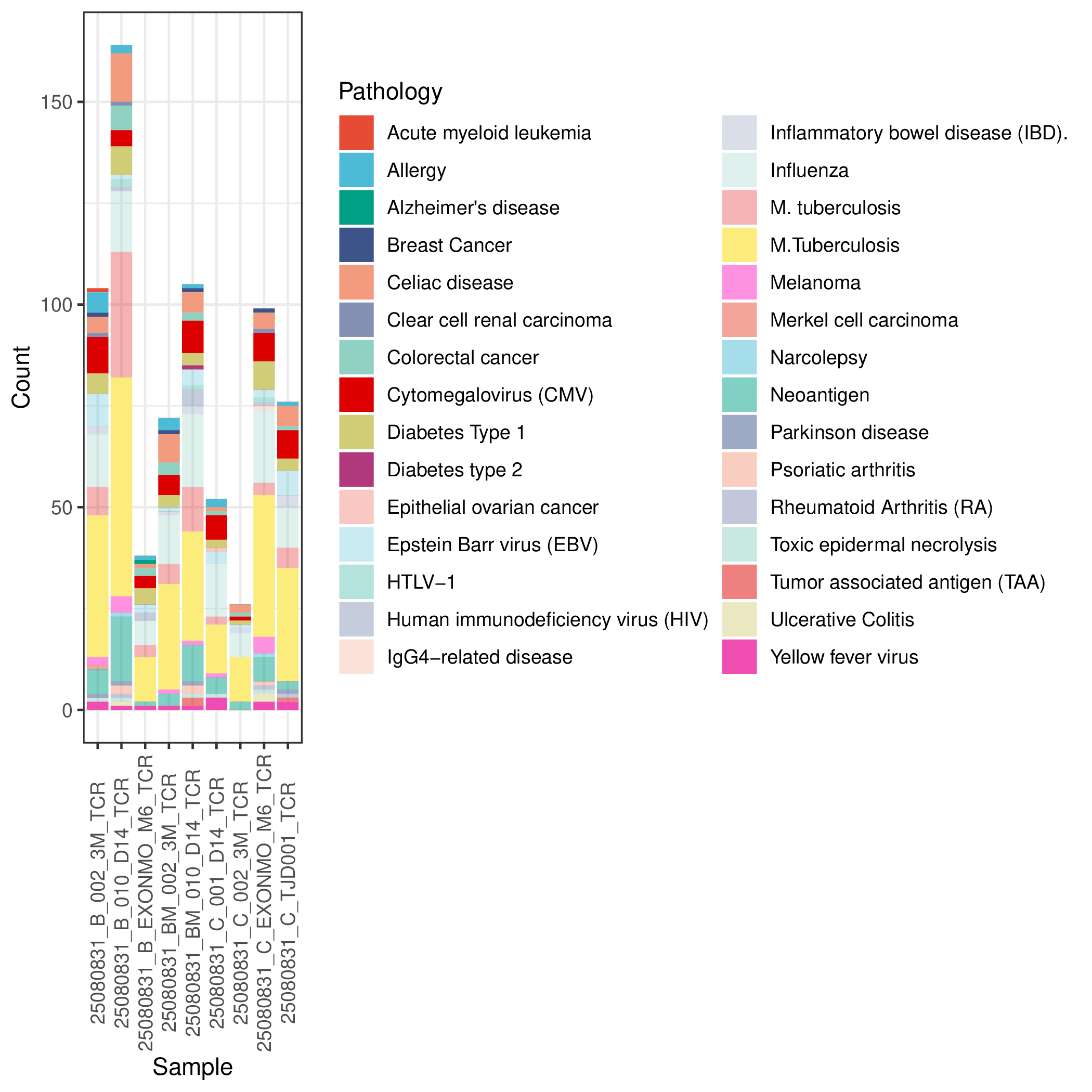 上图展示了各样本中注释到的病理相关TCR数量统计。横坐标为样本名称,纵坐标为检测到的病理相关TCR数量。不同颜色的柱状图代表不同的疾病类型(如癌症、病毒感染、自身免疫病等)。通过该图可以直观地了解:(1)哪些样本富集了特定疾病相关的TCR;(2)样本中主要存在哪些类型的病理相关免疫应答;(3)不同样本间病理相关TCR的组成差异。这些信息有助于理解样本的免疫状态和潜在的疾病关联。
上图展示了各样本中注释到的病理相关TCR数量统计。横坐标为样本名称,纵坐标为检测到的病理相关TCR数量。不同颜色的柱状图代表不同的疾病类型(如癌症、病毒感染、自身免疫病等)。通过该图可以直观地了解:(1)哪些样本富集了特定疾病相关的TCR;(2)样本中主要存在哪些类型的病理相关免疫应答;(3)不同样本间病理相关TCR的组成差异。这些信息有助于理解样本的免疫状态和潜在的疾病关联。
3.13.4 应用场景与注意事项
应用场景:
- 肿瘤免疫分析:识别肿瘤浸润淋巴细胞(TIL)中的肿瘤特异性TCR,评估抗肿瘤免疫应答强度。
- 病毒感染研究:检测与特定病毒(如SARS-CoV-2、EBV、CMV)相关的TCR,评估抗病毒免疫记忆。
- 移植免疫监测:识别与移植排斥或移植物抗宿主病(GVHD)相关的TCR。
- 免疫治疗监测:追踪治疗相关抗原特异性TCR的动态变化,评估治疗应答。
注意事项:
- TCR注释结果为预测性分析,基于序列相似性匹配,需要实验验证才能确认抗原特异性。
- CDR3序列的高度可变性意味着即使序列相似,也可能识别不同的抗原表位。
- 数据库覆盖范围有限,许多TCR序列可能无法找到匹配项,这不代表它们没有生物学功能。
- 注释结果应结合临床背景、组织来源和其他免疫学指标综合解读。
- 同一TCR序列可能在不同疾病中被报道,需要根据研究背景选择最相关的注释。
TIP
结果解读要点:
- 富集特定疾病相关TCR的样本可能存在针对该疾病的活跃免疫应答。
- 病毒相关TCR的检出可能反映既往感染史或疫苗接种史,而非当前活动性感染。
- 肿瘤相关TCR的富集提示存在抗肿瘤免疫应答,但需要进一步验证其功能状态(效应vs耗竭)。
- 注释到的TCR克隆型可作为后续功能验证和免疫治疗开发的候选靶点。
- 结合TCR的克隆扩增状态(参见3.11节)可以更好地评估抗原特异性免疫应答的强度。
3.14 BCR体细胞高频突变分析(BCR)
体细胞高频突变(Somatic Hypermutation, SHM)是B细胞成熟过程中的关键机制,通过在免疫球蛋白变异区基因引入点突变,产生亲和力不同的抗体变体,经过亲和力成熟选择后,产生高亲和力抗体。SHM及序列进化谱系分析可以揭示B细胞的成熟状态、克隆进化关系和抗体亲和力成熟过程,为理解体液免疫应答和抗体开发提供重要信息。
3.14.1 生物学背景
SHM的生物学意义:
- 亲和力成熟:B细胞在生发中心(淋巴结的生发中心)中,通过SHM在BCR变异区引入突变,产生不同亲和力的抗体。高亲和力的B细胞被选择性扩增,低亲和力的被淘汰,这个过程称为亲和力成熟。
- 免疫记忆形成:经历SHM和亲和力成熟的B细胞可以分化为记忆B细胞或浆细胞,提供长期的体液免疫保护。
- 抗体多样性:SHM大大增加了抗体组库的多样性,使机体能够应对变异的病原体。
- 类型转换:SHM通常与类型转换(Class Switch Recombination, CSR)同时发生,B细胞从表达IgM转换为IgG、IgA或IgE,以适应不同的免疫需求。
突变率的生物学含义:
- 低突变率:提示未acquired经历亲和力成熟的初始B细胞(naive B cells)或过渡态B细胞。
- 高突变率:表明经历了充分的亲和力成熟,常见于记忆B细胞和长寿命浆细胞。
- 异常高突变:可能提示慢性抗原刺激、自身免疫病或B细胞恶性肿瘤(如滤泡性淋巴瘤)。
3.14.2 突变频率分析
突变频率(Mutation Frequency)是衡量BCR序列相对于生殖系参考序列的突变率,通常以百分比表示。通过比对BCR序列与生殖系V基因参考序列,计算其变异区中的核苷酸替换比例,从而推断B细胞的成熟状态。
分别对每个细胞群和样本的BCR突变频率进行统计和比较,可以评估不同B细胞亚群的成熟程度和功能状态。

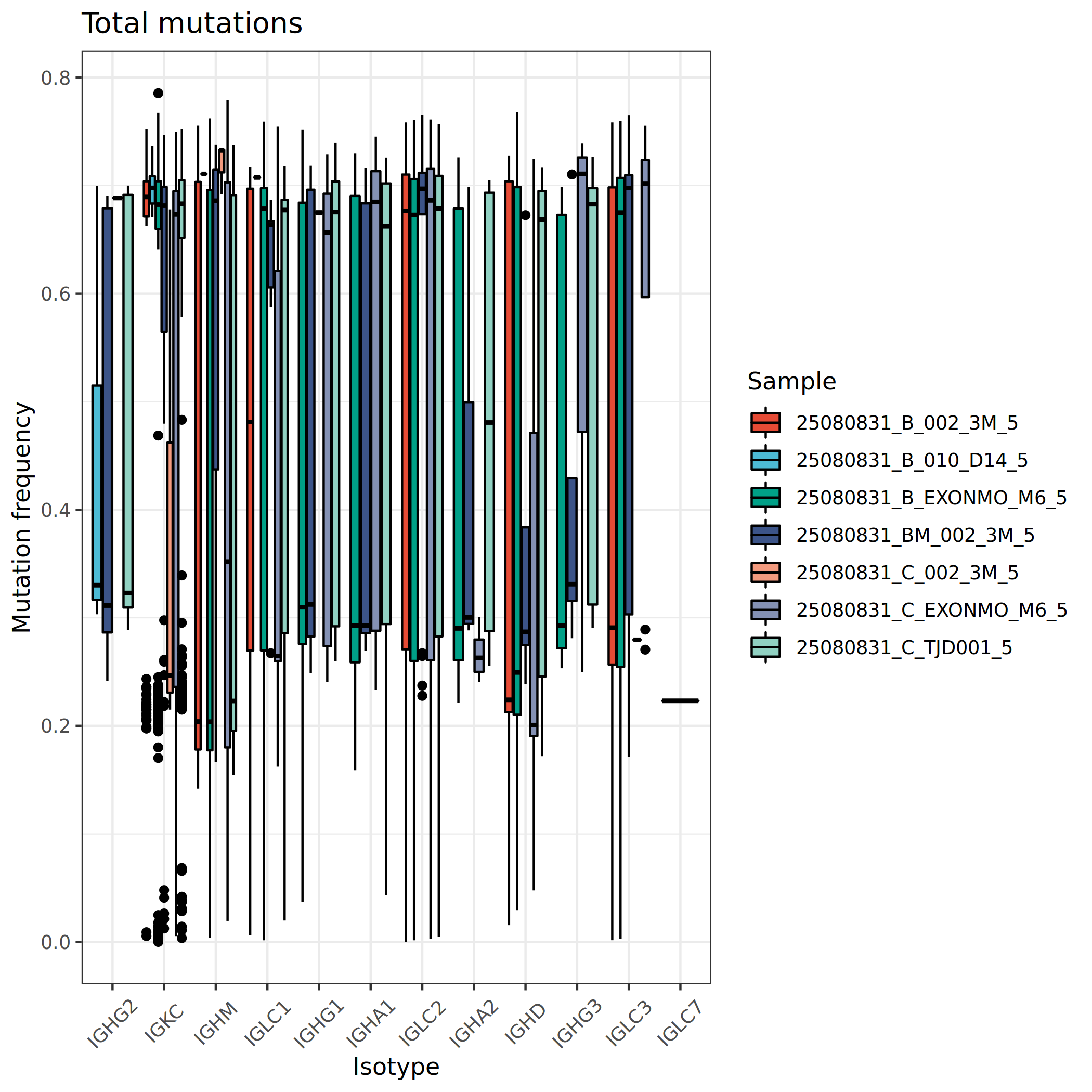 上图展示了不同细胞群和样本中的BCR突变频率。横坐标为样本名称或免疫球蛋白类型,纵坐标为突变频率(百分比)。通过该图可以观察:(1)不同B细胞亚群的成熟程度差异,如记忆B细胞通常具有较高的突变率;(2)不同免疫球蛋白类型的突变特征,如IgG和IgA通常比IgM有更高的突变率;(3)样本间B细胞成熟状态的差异,可能反映疾病状态或治疗影响。
上图展示了不同细胞群和样本中的BCR突变频率。横坐标为样本名称或免疫球蛋白类型,纵坐标为突变频率(百分比)。通过该图可以观察:(1)不同B细胞亚群的成熟程度差异,如记忆B细胞通常具有较高的突变率;(2)不同免疫球蛋白类型的突变特征,如IgG和IgA通常比IgM有更高的突变率;(3)样本间B细胞成熟状态的差异,可能反映疾病状态或治疗影响。
3.14.3 应用场景
SHM分析在以下研究中具有重要应用:
- 抗体发育研究:追踪广谱中和抗体的发育过程,为理性疫苗设计提供指导。
- 自身免疫病:研究自身反应性B细胞的成熟和选择过程。
- 疫苗开发:评估疫苗诱导B细胞应答的质量和成熟程度。
- 感染性疾病:研究抗病毒或抗细菌抗体的亲和力成熟过程。
