Banksy 空间聚类:原理、安装、代码示例与结果解读
时长: 4 分钟
字数: 893 字
更新: 2026-01-26
阅读: 0 次
介绍
NOTE
本文档介绍 Banksy (BANKSY) 空间聚类分析 R 包的原理、安装、使用方法及结果解读,适用于单细胞空间转录组数据的细胞类型识别与空间域分割。
原理简介
IMPORTANT
Banksy 是一种空间组学聚类方法,通过将每个细胞的表达特征与其空间邻域的表达均值及空间梯度特征进行融合,增强细胞特征用于聚类。该方法能够:
- 提高噪声数据中的细胞类型判别能力。
- 区分受微环境影响的细微细胞亚型。
- 识别具有相似微环境的空间结构域。
Banksy 适用于多种空间转录组技术 (如 SeekSpace、Slide-seq、MERFISH、CosMX 等),并可扩展至大规模数据集。
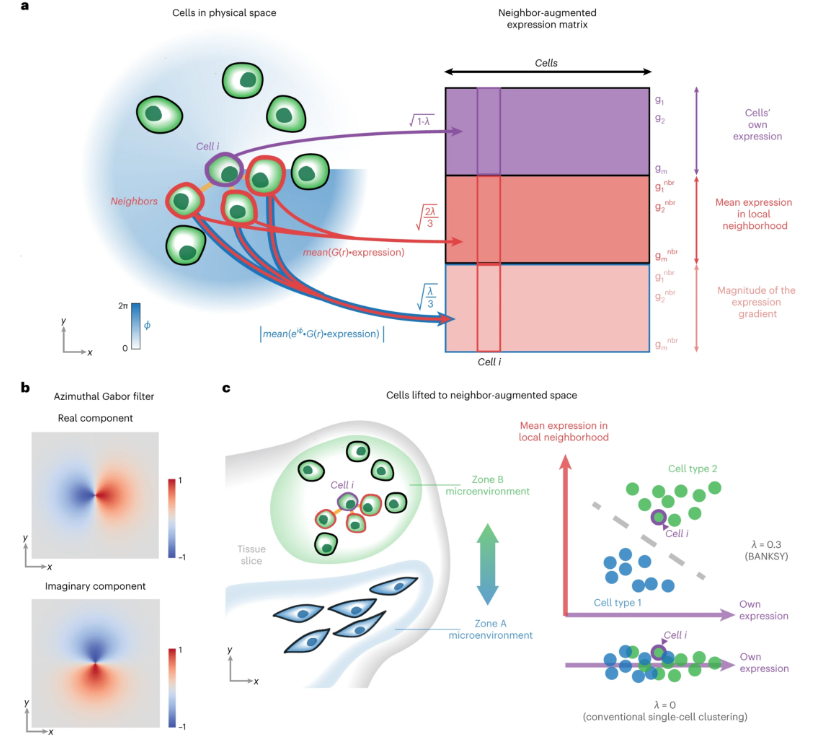
图: Banksy 空间聚类方法示意
数据准备
示例数据
Banksy 官方 R 包自带小鼠海马体空间转录组数据,可直接用于测试。
r
# 加载示例数据
library(Banksy)
data(hippocampus)
gcm <- hippocampus$expression
locs <- as.matrix(hippocampus$locations)自有数据
- 表达矩阵(基因×细胞/空间点)。
- 空间坐标矩阵(细胞/空间点×2)。
环境安装
IMPORTANT
需 R >= 4.4.0,建议使用 Bioconductor 安装。
r
# Bioconductor 安装
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install('Banksy')
# 或从 GitHub 安装最新版
if (!requireNamespace("remotes", quietly = TRUE))
install.packages("remotes")
remotes::install_github("prabhakarlab/Banksy")TIP
推荐同时安装 SpatialExperiment、scuttle、scater、ggplot2、cowplot 等包以便数据处理和可视化。
r
BiocManager::install(c("SpatialExperiment", "scuttle", "scater"))
install.packages(c("ggplot2", "cowplot"))运行示例
r
# 加载所需包
library(Banksy)
library(SpatialExperiment)
library(scuttle)
library(scater)
library(ggplot2)
library(cowplot)
# 构建 SpatialExperiment 对象
se <- SpatialExperiment(assay = list(counts = gcm), spatialCoords = locs)
# 质控与归一化
qcstats <- perCellQCMetrics(se)
thres <- quantile(qcstats$total, c(0.05, 0.98))
keep <- (qcstats$total > thres[1]) & (qcstats$total < thres[2])
se <- se[, keep]
se <- computeLibraryFactors(se)
assay(se, "normcounts") <- normalizeCounts(se, log = FALSE)
# 计算 Banksy 邻域特征
lambda <- c(0, 0.2)
k_geom <- c(15, 30)
se <- Banksy::computeBanksy(se, assay_name = "normcounts", compute_agf = TRUE, k_geom = k_geom)
# 降维与聚类
set.seed(1000)
se <- Banksy::runBanksyPCA(se, use_agf = TRUE, lambda = lambda)
se <- Banksy::runBanksyUMAP(se, use_agf = TRUE, lambda = lambda)
se <- Banksy::clusterBanksy(se, use_agf = TRUE, lambda = lambda, resolution = 1.2)
se <- Banksy::connectClusters(se)
# 可视化
cnames <- colnames(colData(se))
cnames <- cnames[grep("^clust", cnames)]
colData(se) <- cbind(colData(se), spatialCoords(se))
plot_nsp <- plotColData(se, x = "sdimx", y = "sdimy", point_size = 0.6, colour_by = cnames[1])
plot_bank <- plotColData(se, x = "sdimx", y = "sdimy", point_size = 0.6, colour_by = cnames[2])
plot_grid(plot_nsp + coord_equal(), plot_bank + coord_equal(), ncol = 2)参数说明
| 参数 | 说明 |
|---|---|
| lambda | 空间邻域特征权重,0 为非空间聚类,0.2 为推荐值 (细胞分型)。 |
| k_geom | 空间邻居数,推荐 15 和 30。 |
| use_agf | 是否使用邻域均值和梯度特征。 |
| resolution | 聚类分辨率,影响聚类数目。 |
| assay_name | 使用的表达矩阵名称 (如 normcounts)。 |
WARNING
- lambda 参数对不同空间分辨率数据有不同推荐值,详见官方文档。
- 运行大规模数据时建议高性能计算资源。
主要结果说明
- 聚类标签:colData(se) 中的 clust_* 列为不同 lambda 下的聚类结果。
- 降维结果:reducedDims(se) 中 UMAP 等降维坐标。
- 可视化:plotColData、plotReducedDim 等函数可绘制空间聚类分布。
NOTE
Banksy 聚类结果可用于后续空间亚型分析、空间域识别、微环境研究等。
