单细胞 scATAC-seq & scRNA-seq 双组学标准分析:Motif 分析
文档概述
Motif 分析是解析单细胞 ATAC-seq 数据中转录因子调控网络的重要关节。常见分析策略包括:在差异可及性 peak 集合中挖掘富集的 motif,以及对不同细胞群体间进行 motif 活性差异分析。本文档将系统阐述 motif 的基本概念、差异可及性 peak 集合的motif 富集分析方法、motif 活性差异分析。
一、核心概念:什么是 Motif?
(1) Motif 的定义
转录因子需要与DNA开放区域(peaks)结合才能发挥调控作用,每个转录因子通常有偏好的结合序列(如 SP1 偏好 GC 富集序列),这些序列模式就是 Motif,因此可以将Motif(基序)理解为转录因子结合位点的 DNA 序列模式,通常由 6-20 个碱基对组成。如下所示:

(2) Motif 数据库
到目前为止,已经有多个权威数据库整合并完善了不同物种的motif信息,为后续的分析提供了结构化、全面的数据资源。常用的 motif 数据库包括:JASPAR 数据库、HOMER、MEME Suite、TRANSFAC,其中JASPAR 数据库是最常用的转录因子结合位点数据库,包含多个物种的 motif。
TIP
早期对 motif 的确定主要依赖于实验手段(如ChIP-seq、SELEX等)或生物信息学预测,得到的是零散且有限的转录因子结合序列。随着研究深入和大量实验数据的积累,motif 信息不断丰富和标准化。Motif的信息已经相对较健全,直接通过各个数据库就能查阅到对应物种的motif信息。
二、Motif 富集分析
(1) 什么是 Motif 富集?
Motif 富集是指统计特定基因组区域中(如差异peaks),每种特定 motif 出现的频率,并与背景 peaks(如全体 peaks 或 matched control)中的频率进行比较。如果某个 motif 在差异 peaks 中的出现频率显著高于背景,就认为该 motif 在这些区域“富集”。
简单来说,就是统计某些区域(如差异 peaks)里某个 motif 出现了多少次,再和背景区域对比,如果在差异 peaks 里出现得更多,就认为这个 motif 富集
有如下应用:
- 比较不同细胞类型或状态的 peaks 开放程度,筛选出某一类型或状态中特异性开放的 peaks,进而分析其富集的 motif,推测对应的关键调控因子。
- 针对差异 peaks(如疾病组 vs 对照组),进行 motif 富集分析,挖掘调控异常相关的 TF,辅助生物学机制解释和功能归因。
TIP
想要更加直观地理解 对差异peaks进行Motif 富集分析的应用,可以参考实际案例,查阅关于单细胞双组学数据中差异 peak 的 Motif 富集分析部分,详见《单细胞ATAC_RNA多组学差异peaks分析》。
(2) Motif 富集分析的实现:
以Signac为例, Signac使用 AddMotifs() 函数将 motif 信息添加到 Seurat 对象,并用 FindMotifs() 进行 motif 富集分析。下面是关键代码示例(以人类、hg38 为例):
library(Signac)
library(JASPAR2022)
library(TFBSTools)
# 1. 载入 motif 数据库(以人物种为例)
pfm <- getMatrixSet(
x = JASPAR2020,
opts = list(collection = "CORE", tax_group = "vertebrates", all_versions = FALSE)
)
# 2. 添加 motif 信息到 ATAC 对象(例如atac)
DefaultAssay(obj) <- "ATAC"
obj <- AddMotifs(
object = obj, # 你的Seurat/Signac对象
genome = BSgenome.Hsapiens.UCSC.hg38, # 注意加载合适的基因组包
pfm = pfm
)
# 3. 对感兴趣的peaks进行motif富集分析,使用obj中的全部peaks作为背景peaks
motif.results <- FindMotifs(
object = obj,
features = diff_peak # 例如差异分析得到的差异peaks列表
)
head(motif.results)| motif | observed | background | pct.obs | pct.bkg | fold.enrich | pvalue | motif.name | p.adjust |
|---|---|---|---|---|---|---|---|---|
| MA0497.1 | 556 | 8315 | 49.16 | 20.79 | 2.365 | 0 | MEF2C | 0 |
| MA0052.4 | 533 | 7948 | 47.13 | 19.87 | 2.372 | 0 | MEF2A | 0 |
| MA0773.1 | 398 | 4930 | 35.19 | 12.33 | 2.855 | 0 | MEF2D | 0 |
| MA0660.1 | 345 | 4002 | 30.50 | 10.01 | 3.049 | 0 | MEF2B | 0 |
| MA1151.1 | 286 | 3206 | 25.29 | 8.02 | 3.155 | 0 | RORC | 0 |
| MA0592.3 | 350 | 4669 | 30.95 | 11.67 | 2.651 | 0 | MEF2A | 0 |
motif:motif 在数据库中的编号,通常代表一个特定的转录因子的结合序列。observed:在目标 peaks(如差异 peaks)中包含该 motif 的数量。background:在背景 peaks(对照组或全部 peaks)中包含该 motif 的数量。pct.obs:目标 peaks 中带有该 motif 的百分比(= observed/目标 peaks 总数 × 100%)。pct.bkg:背景 peaks 中带有该 motif 的百分比(= background/背景 peaks 总数 × 100%)。fold.enrich:motif 在目标 peaks 中的富集倍数,= pct.obs / pct.bkg,数值越大表明 motif 在差异 peaks 中越富集。pvalue:motif 富集分析的统计显著性原始 P 值,越小意味着富集不太可能是随机出现的。 motif.name:motif 对应的转录因子名称,便于直观识别调控因子。p.adjust:多重假设检验校正后的 P 值(如 FDR 校正),p.adjust < 0.05 常被认为是显著富集。
通过上述指标,可以判定哪些 motif(及其对应转录因子)在差异 peaks 中显著且强烈富集,从而揭示可能参与调控的关键 TF。
TIP
背景peaks:
- 是指在motif富集分析中用作对照的一组peaks,通常代表全体peaks或无显著差异的peaks。其作用是为感兴趣peaks(如差异peaks)中某一motif的出现频率提供一个基准。
- 通过比较差异peaks与背景peaks中某motif的富集程度,可以判断该motif是否在特定生物学状态下有显著富集。
- FindMotif()函数通过background参数可以提供一个特征向量作为背景集,或者提供一个数字来指定随机选择的特征数量作为背景集。
三、Motif 活性分析
(1) 什么是 Motif 活性?
- Motif 活性(Motif Activity)是衡量某个转录因子的 motif 在特定细胞中的潜在结合和调控能力的指标。
- 与 Motif 富集分析不同,motif 活性不是简单地统计 motif 出现的次数,而是考虑该细胞中哪些 peak 含有该 motif,以及这些 peak 的开放程度。
(2) Motif 活性的计算
以 Signac 为例,motif 活性分析的主要流程与 R 代码如下:
library(Signac)
library(Seurat)
library(JASPAR2022)
library(TFBSTools)
library(BSgenome.Hsapiens.UCSC.hg38) # 或根据你的物种调整
# 获取 motif 数据集
pfm <- getMatrixSet(
x = JASPAR2022,
opts = list(collection = "CORE", tax_group = "vertebrates")
)
# 将 motif 信息添加到 ATAC 对象(假设对象名为 obj,Assay 名为 ATAC)
obj <- AddMotifs(
object = obj,
genome = BSgenome.Hsapiens.UCSC.hg38,
pfm = pfm
)
# 运行 chromVAR 计算 motif 活性
obj <- RunChromVAR(
object = obj,
genome = BSgenome.Hsapiens.UCSC.hg38
)查看 motif 活性结果
- 运行完成后,会在对象中新增一个名为 "chromvar" 的 Assay。可以用 Seurat 的 FeaturePlot可视化 motif 活性。
- 例如,可将特定 motif 的活性在 UMAP 上可视化:
FeaturePlot(obj, features = "MA0497.1", assay = "chromvar")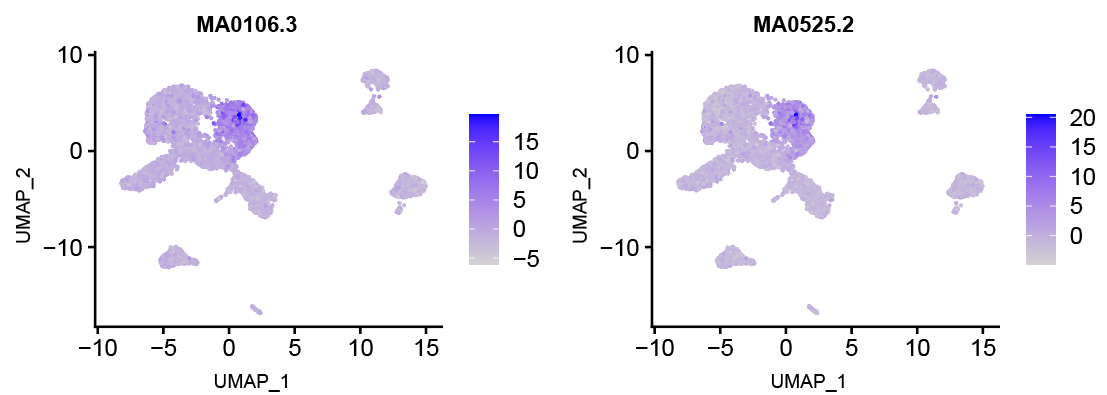
- Motif 活性分数解读
- 偏差分数为正值:该 motif 在该细胞中活跃,TF 可能功能活跃。
- 偏差分数为负值:该 motif 在该细胞中不活跃或受抑制。
- 偏差分数接近零:活性与背景无显著差异。
WARNING
注意事项
- 计算 motif 活性时建议使用合适物种的基因组版本。
- motif 数量或细胞数多时分析耗时较长,可结合并行加速。
四、Motif活性的差异分析
在前文我们介绍了如何在单细胞水平计算每个 motif 的活性分数(chromVAR 偏差分数)。有了这些 motif 活性分数矩阵后,接下来我们常常希望比较不同细胞类型或状态之间,哪些 motif 的活性存在显著差异,从而进一步推断关键转录因子。
直接进行Motif活性差异分析(Differential Motif Activity)
相比只基于差异peak进行的motif富集分析,直接比较细胞类别间的motif活性差异,结果通常具有高度一致性,并且能反映更直观的调控活跃程度。
以 Signac/Seurat 分析框架为例,可以利用 FindMarkers()、FindAllMarkers() 函数直接对 chromVAR 得到的 z-score(即 motif 活性矩阵中的值)进行两组/两类细胞之间的差异分析。此时,常用如下参数来获得“不同类别平均z-score之差”:
differential.activity <- FindMarkers(
object = obj,
ident.1 = 'Pvalb',#比较‘Pvalb’ 与 ‘Sst’ 两组细胞的 motif 活性差异
ident.2 = 'Sst',
only.pos = TRUE,
mean.fxn = rowMeans,
fc.name = "avg_diff"
)TIP
FindAllMarkers()适用于一次性自动比较每个细胞群体与所有其他群体的差异 peaks,非常适合多组分群(如多个细胞类型或状态)的全面差异分析。FindMarkers()适用于两组之间的直接对比(如疾病组 vs 对照组),可实现灵活的一对一差异 peaks 筛查,更便于特定分组之间的精确比较。
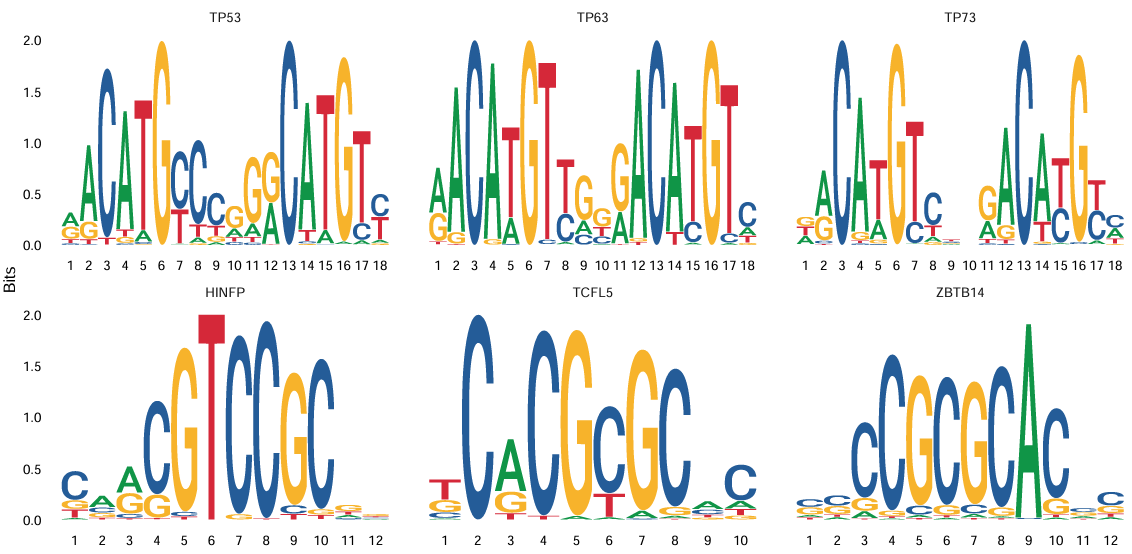
获得差异 motif 后,可用 MotifPlot 对显著 motif 进行可视化:
MotifPlot(
object = obj,
motifs = head(rownames(differential.activity)),
assay = 'peaks'
)
五、Motif活性解读
在多组学数据中,可以同时检查:Motif 活性(ATAC 数据)和TF 表达(RNA 数据)
(1)Motif 活性与 TF 表达一致:
理想情况下,如果某个转录因子在某细胞类型中活跃,其 motif 活性应该高,其 RNA 表达也应该高。
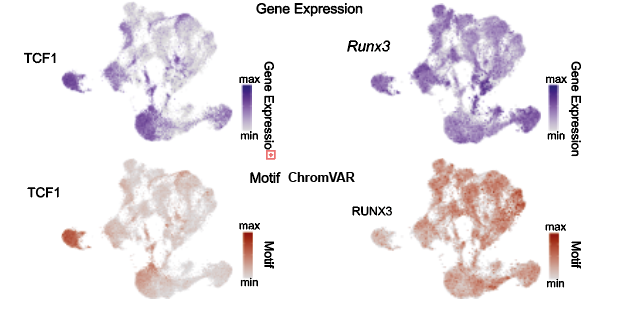
(2)Motif 活性与 TF 表达不一致:
- motif 活性高但 TF 表达低,可能原因:
- 转录因子以非表达依赖的方式调控(如通过共激活因子)
- 转录因子在蛋白水平存在但 RNA 水平较低
- 其他转录因子共享相同的 motif
- TF 表达高但 motif 活性低,可能原因:
- 转录因子可能结合在其他区域(非 peak 区域)
- 转录因子可能以不依赖 DNA 结合的方式起作用
TIP
关键转录因子的识别:
- 在多组学数据中,应重点关注 motif 活性和 TF 表达均高的转录因子。
- 这类 TF 通常是“组合证据”支持的核心调控因子,既具备较强的结合潜能(motif 活性),也有明确的表达(RNA),同时可能直接调控相关Target基因(peak-gene 关联)。
六、Motif 分析的生物学应用
(1) 细胞类型特异性调控因子
识别细胞类型标记 TF:
- 通过比较不同细胞类型的 motif 活性,可以识别每种细胞类型特异的转录因子。
- 这些 TF 可以作为细胞类型的调控标记,类似于基因表达层面的 marker 基因。
- 例如,SPI1/PU.1 是小胶质细胞的标记转录因子;PAX5 是 B 细胞的标记转录因子。
(2) 疾病相关的调控异常
疾病样本中的 Motif 活性异常:
- 比较疾病样本与对照样本的 motif 活性,可以识别哪些转录因子的调控出现异常。
- 这些异常可能提示疾病的调控机制或潜在的治疗靶点。
- 例如,在阿尔茨海默病中,某些转录因子(如 ZEB1、MAFB)的 motif 活性可能异常。
(3) 多组学数据的优势
RNA-ATAC 整合验证:
在多组学数据中,可以同时检查:
- Motif 活性(来自 ATAC):反映转录因子的潜在结合能力。
- TF 表达(来自 RNA):反映转录因子的实际表达水平。
如果某个转录因子的 motif 活性高且表达也高,说明该 TF 在该细胞中活跃且参与调控。
TIP
三证合一的调控分析:
- Peak-Gene-TF Trio 分析:
- 识别差异开放的 peak(差异 peak 分析)。
- 关联 peak 到附近基因(基因注释)。
- 识别 peak 中富集的 motif(motif 分析)。
- 验证 TF 表达与 peak 开放、基因表达的相关性(多组学整合)。
- 通过三证合一,可以更可靠地识别关键转录因子和调控机制。
- Peak-Gene-TF Trio 分析:
七、常见问题
Q1:Motif 活性与 Motif 富集有什么区别?
A:两者相关但不同:
Motif 富集:
- 关注点:特定 peak 集合(如差异 peak)中哪些 motif 显著富集。
- 方法:统计 motif 在目标 peak 集合中的出现频率,与背景比较。
- 结果:得到一个 motif 富集列表,说明哪些 TF 可能调控这些 peak。
- 应用:通常用于差异 peak 分析后的功能解释。
Motif 活性:
- 关注点:每个细胞中每个 motif 的活性水平。
- 方法:考虑该细胞中包含该 motif 的 peak 的开放程度,计算偏差分数。
- 结果:得到一个细胞 × motif 的活性矩阵。
- 应用:可以用于细胞聚类、识别细胞类型特异的 TF、分析 TF 活性的动态变化。
