Monocle3拟时序分析文档
前言
TIP
Monocle3是单细胞轨迹分析领域的先进工具,它在前代版本的基础上进行了重大革新,能够处理百万级别细胞的超大数据集,并能更好地解析复杂的、具有多个分离谱系的生物学过程。
在单细胞研究中,我们捕获的细胞往往处于一个连续变化过程中的不同“快照”。拟时序(Pseudotime)分析的核心任务就是利用算法将这些静态的快照根据其内在的转录组相似性重新排序,从而重建出它们所经历的动态生物学轨迹,如细胞分化、疾病进展或药物响应过程。
Monocle3的核心功能
- 大规模数据处理:能够高效分析百万细胞级别的海量数据集。
- 复杂轨迹构建:擅长处理含有多个不相连轨迹(Partitions)的复杂生物学系统。
- 基于UMAP的轨迹学习:直接在UMAP等低维空间中学习轨迹,更好地保留了细胞的全局结构。
- 基因动态分析:识别沿轨迹或在特定模块中协同变化的基因集,揭示调控过程的核心分子机制。
本篇文档旨在为研究者提供一份详尽的Monocle3技术指南,内容涵盖其核心原理、在SeekSoulOnline云平台上的操作方法、结果解读、实战案例及常见问题,帮助您快速掌握并应用这一强大的分析工具。
Monocle3理论基础
核心原理
Monocle3的核心思想与Monocle2一脉相承,但实现路径截然不同。它认为:细胞的动态过程可以在一个低维的几何空间(如UMAP图)中被描述为一个或多个图(Graph)结构。 通过在这个低维空间中学习细胞的主图(Principal Graph),就可以重建出发育轨迹。
Monocle3的轨迹推断主要分为四步:
降维 (Dimension Reduction):首先,使用PCA和UMAP等标准方法对数据进行降维,将细胞投影到一个二维或三维空间中。这是后续所有分析的基础。
划分分区 (Partitions):Monocle3会自动检测降维空间中是否存在分离的、不相连的细胞群,并将它们划分为不同的“分区”(Partitions)。这使得Monocle3可以同时分析多个独立的轨迹。
学习主图 (Learn Principal Graph):在每个分区内,Monocle3使用SimplePPT算法学习一个能够穿过细胞云中心的主图。这个图的“骨架”就代表了细胞状态转变的主要路径。
细胞排序与指定起点 (Order Cells):用户需要根据生物学先验知识,在图上指定一个或多个“根节点”(Root nodes)作为轨迹的起点。随后,算法会计算每个细胞到最近根节点的最短路径距离,这个距离就被定义为该细胞的拟时序值。
与Monocle2的关键区别
| 特性 | Monocle2 | Monocle3 |
|---|---|---|
| 核心算法 | DDRTree | UMAP + SimplePPT |
| 数据规模 | 适用于数万细胞 | 可扩展至百万细胞 |
| 起点指定 | 可自动推断,但常需手动校正 | 必须手动指定 |
| 轨迹形态 | 通常是单一的、连通的树状结构 | 可包含多个不相连的轨迹(分区) |
| 依赖关系 | 独立流程 | 紧密整合Seurat/Scanpy预处理结果 |
TIP
理解Monocle3与Monocle2的最大区别在于:Monocle2试图在高维空间中直接学习一棵“树”,而Monocle3则先将数据“拍扁”到UMAP图上,再在图上“画”出轨迹。这使得Monocle3速度更快,且能更好地处理复杂的、断开的轨迹。
云平台操作指南
在云平台上,Monocle3的分析流程被设计得直观易用。您无需编写代码,只需通过参数配置界面即可完成分析。
分析前的准备
TIP
Monocle3分析的成功与否,很大程度上取决于输入数据的质量和生物学问题的合理性。在开始分析前,请务必确认:
- 数据已完成预处理:您的单细胞数据已经过标准的质控、降维(PCA、UMAP)、聚类和细胞类型注释。
- 选择了合适的细胞亚群:拟时序分析应在具有潜在分化或转变关系的细胞亚群中进行。将生物学上毫无关联的细胞放在一起进行分析是没有意义的。
- 明确生物学起点:这是Monocle3分析最关键的一步。您必须根据已知的生物学知识,明确哪个细胞群是您所研究过程的起始状态(如干细胞、祖细胞、初始T细胞等)。
参数详解
下表详细列出了云平台Monocle3分析模块的主要参数及其说明。
| 界面参数 | 说明 |
|---|---|
| 任务名称 | 本次分析的任务名称,需以英文字母开头,可包含英文字母、数字、下划线和中文。 |
| root类型 | 选择要分析的细胞类型或聚类对应的标签,例如细胞注释分组celltype。此参数与“root节点”和“细胞类型”配合使用。 |
| root节点 | 核心参数。选择细胞发育过程中的祖细胞或发育前期的细胞类型,例如Plasmablast细胞。 |
| 细胞类型 | 多选,选择要纳入拟时序分析的所有细胞类型。 |
| 拆分因子 | 多选,绘图时用于拆分不同组别的标签。一般选择分组或样本的标签,比如Group或Sample。 |
| 允许存在多个不相连轨迹 | 逻辑值,决定是否允许学习在不同分区(Partitions)中的不相连轨迹。默认允许,适用于存在多个独立发育过程的复杂数据集。 |
| 富集分析 | 逻辑值,选择是否对识别出的基因模块进行GO/KEGG功能富集分析。默认进行。 |
| 物种 | 选择数据对应的物种,用于基因功能富集分析。目前平台支持人和小鼠。 |
| Downsample | 逻辑值,选择是否对细胞进行降采样(随机抽取部分细胞)后再进行分析。适用于超大规模数据集,可显著提升计算速度。 |
| Downsample_num | 整数,如果启用Downsample,此参数用于指定抽取的细胞数量。 |
| 备注 | 自定义备注信息。 |
操作流程
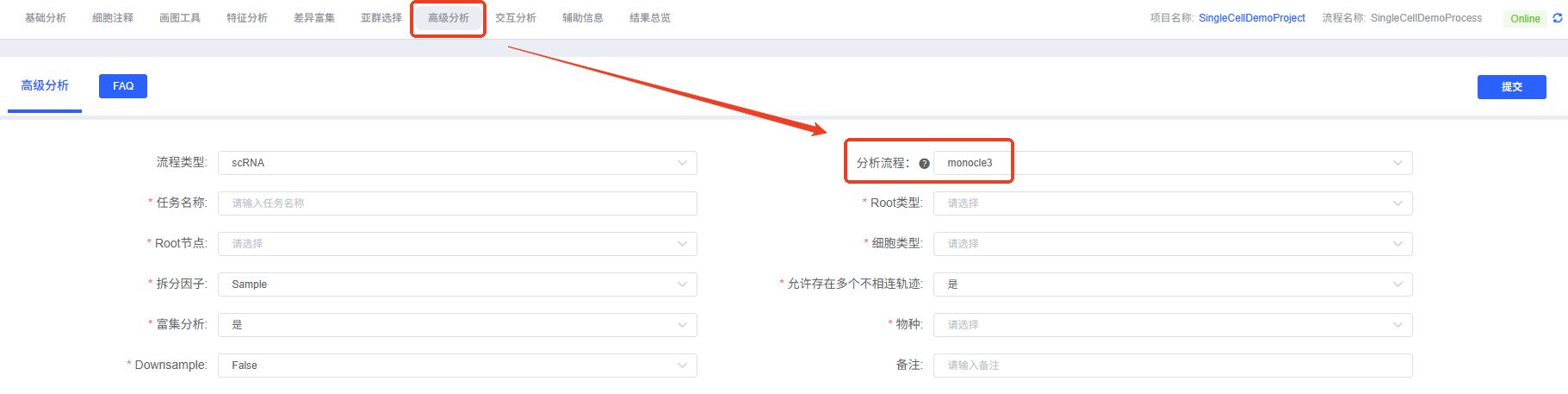
- 进入分析模块:在云平台导航至“高级分析”模块,分析流程选择“monocle3”。
- 创建新任务:为您的分析任务命名,并选择要分析的样本或项目。
- 配置参数:根据上述指南,选择要分析的细胞类型,并务必准确指定轨迹的起点。
- 提交任务:确认参数无误后,点击“提交”按钮,等待分析完成。
- 下载与查看:分析结束后,在任务列表中下载并查看生成的分析报告和结果文件。
结果解读
Monocle3的分析报告包含丰富的图表和数据文件,以下是对核心结果的详细解读。
5.1 细胞轨迹总览图
这是Monocle3分析最核心和最直观的结果。它在UMAP降维图的基础上,叠加了学习到的轨迹“骨架”和计算出的拟时序值。
5.1.1 Pseudotime分布图
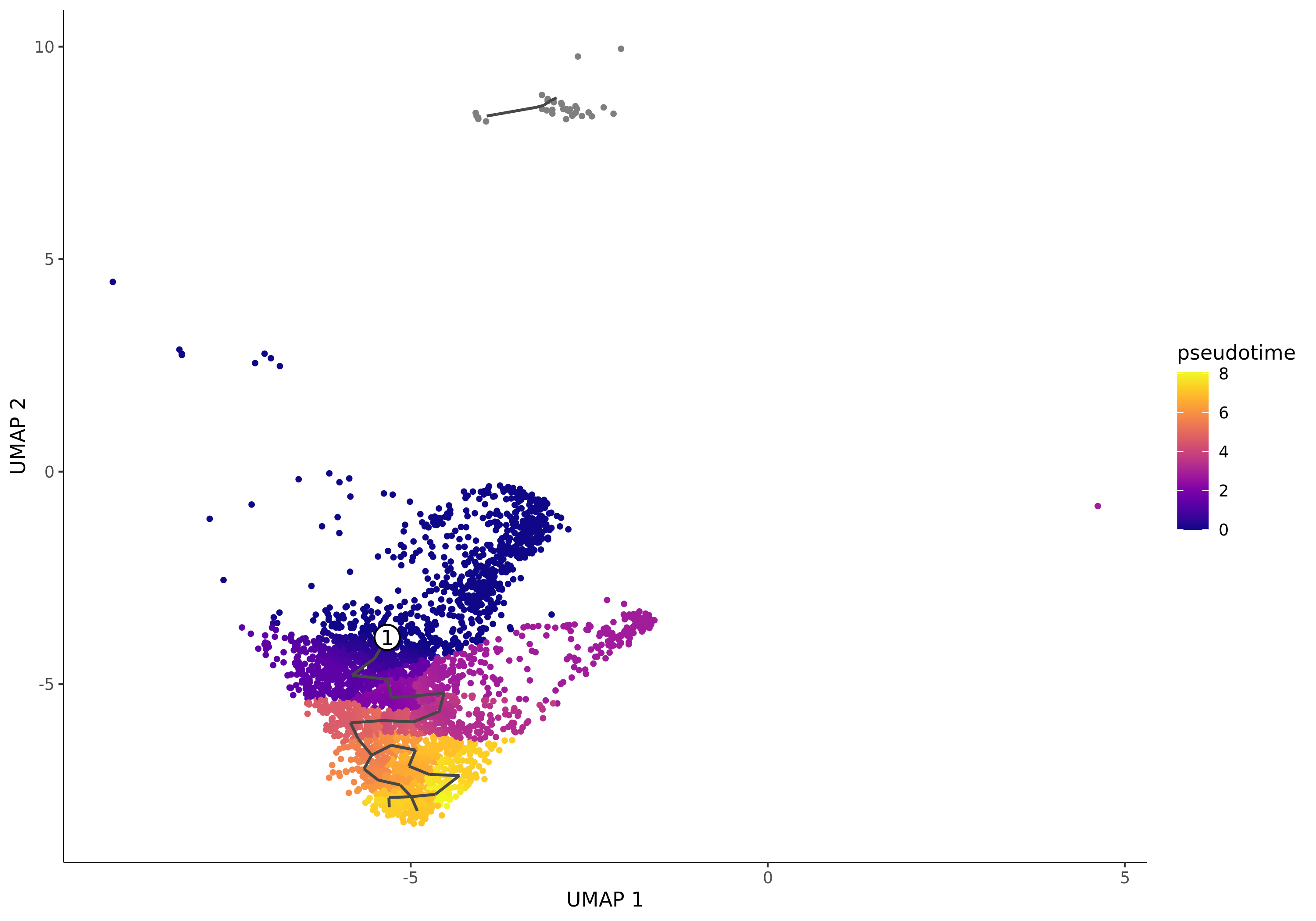
- 图表解读:每个点代表一个细胞。颜色代表该细胞的拟时序值,通常从深色(早期)到亮色(晚期)渐变。拟时序值越小,代表细胞越接近您指定的起点。白底圆圈的编号表示您指定的root节点(起点)。
- 分析要点:
- 观察轨迹的整体形状和颜色梯度,判断分化方向是否符合预期。
- 注意灰色的点,这些点代表与起点不处于同一个“分区”(Partition)的细胞,Monocle3没有为它们计算拟时序值。
5.1.2 细胞群(celltype)轨迹图
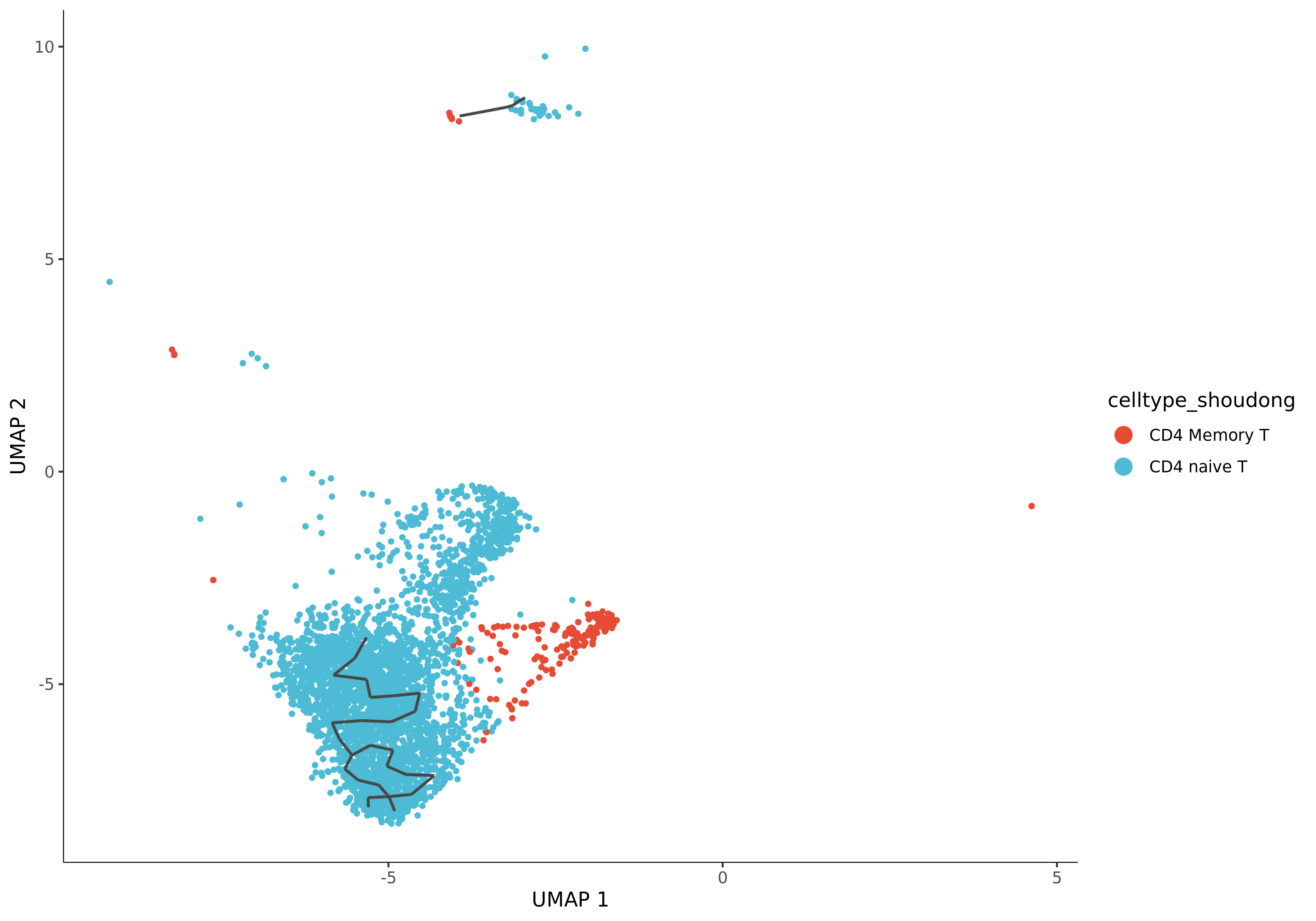
- 图表解读:这是至关重要的一张图。它将您预先注释的细胞类型(Cell Type)映射到轨迹上,用不同颜色表示。
- 分析要点:
- 验证轨迹的生物学意义:检查细胞类型的分布是否符合已知的生物学过程。您指定的起点细胞群是否位于轨迹的开端?终末分化的细胞是否位于轨迹的末端?
- 确定分化路径:通过观察细胞类型的连续排布,可以推断出分化的方向和路径。
报告中还会提供带有分支节点(Branch Point)和终末节点(Leaves)的轨迹图,以及按样本或分组拆分的轨迹图,便于更深入的分析。
5.2 随轨迹变化的基因
Monocle3使用空间自相关分析(Moran's I)来识别在UMAP空间上表现出显著空间共表达模式的基因,这些基因很可能是在轨迹上动态变化的关键基因。
5.2.1 核心基因表达趋势图
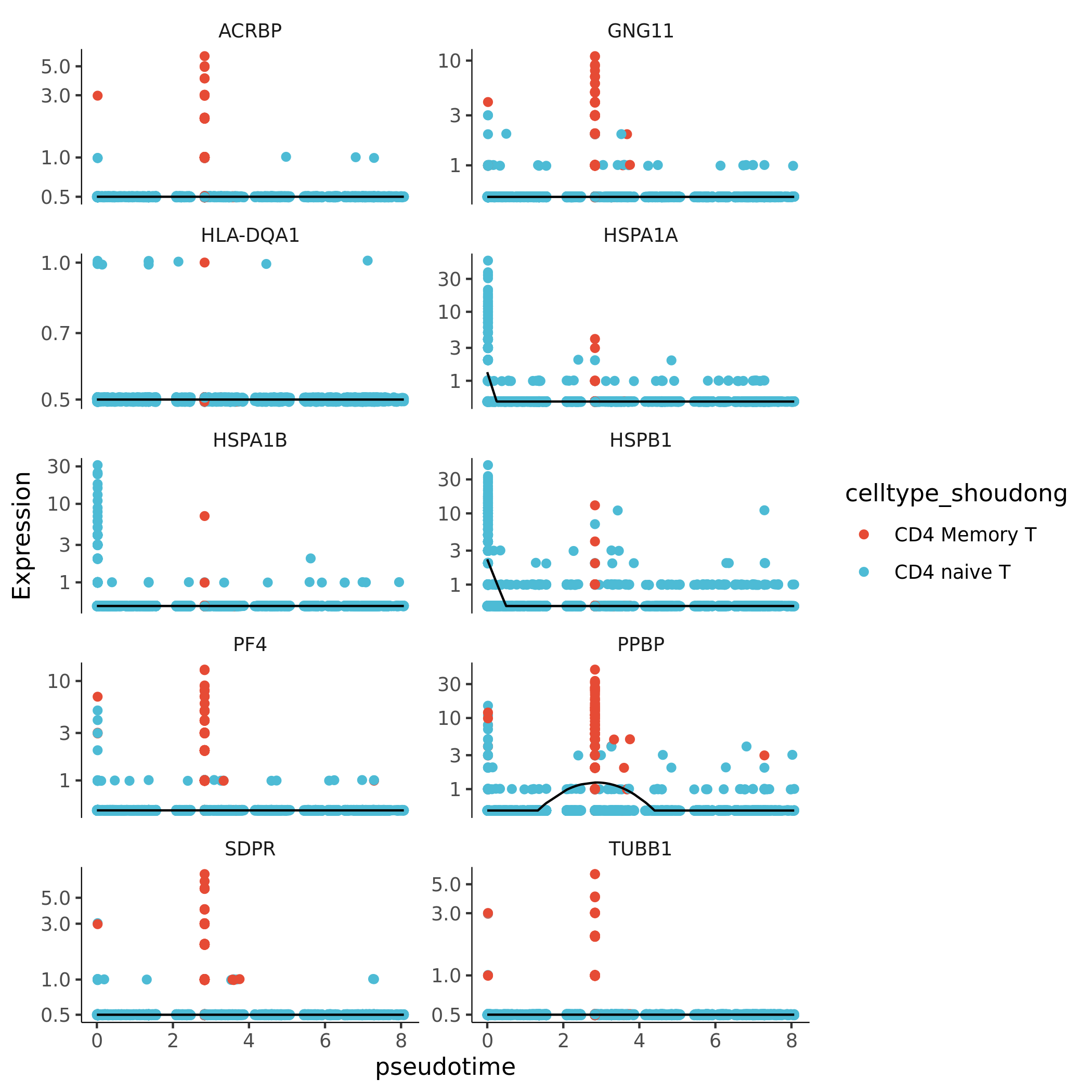
- 图表解读:该图展示了Moran's I指数最高的一组基因(Top10)的表达量如何随拟时序的变化而变化。
- 横坐标:拟时序(Pseudotime)。
- 纵坐标:基因的标准化表达量。
- 每个点:代表一个细胞。
- 曲线:是拟合后的基因表达趋势。
- 分析要点:
- 识别不同的基因表达模式,例如,在发育早期高表达、晚期下调的基因,或是在发育晚期逐渐上调的基因。
5.3 共调控基因模块分析
为了更好地理解基因功能,Monocle3会将具有相似表达趋势的基因聚类成“基因模块”(Gene Modules)。
5.3.1 基因模块表达热图
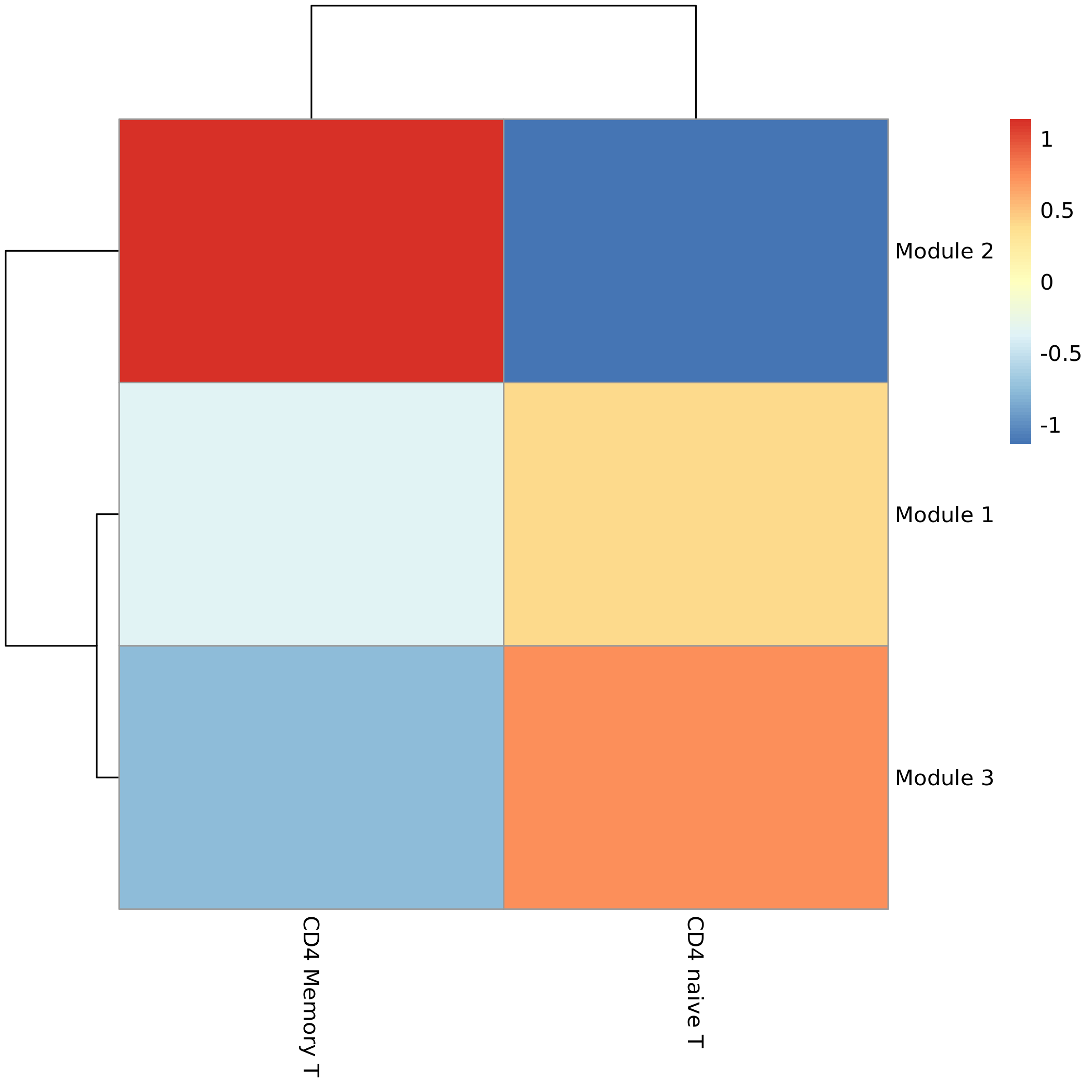
- 图表解读:热图展示了不同基因模块在不同细胞类型中的平均表达水平。
- 横坐标:细胞类型(按轨迹顺序或聚类关系排列)。
- 纵坐标:基因模块。
- 颜色:红色表示该模块在该细胞类型中整体高表达,蓝色表示低表达。
- 分析要点:
- 将特定的基因模块与特定的细胞类型或发育阶段关联起来。例如,某个模块可能只在轨迹起点的干细胞中高表达,而另一个模块则在多个终末分化细胞中高表达。
5.4 基因模块功能富集分析
对每个基因模块进行GO和KEGG功能富集分析,可以揭示不同发育阶段的生物学功能变化。
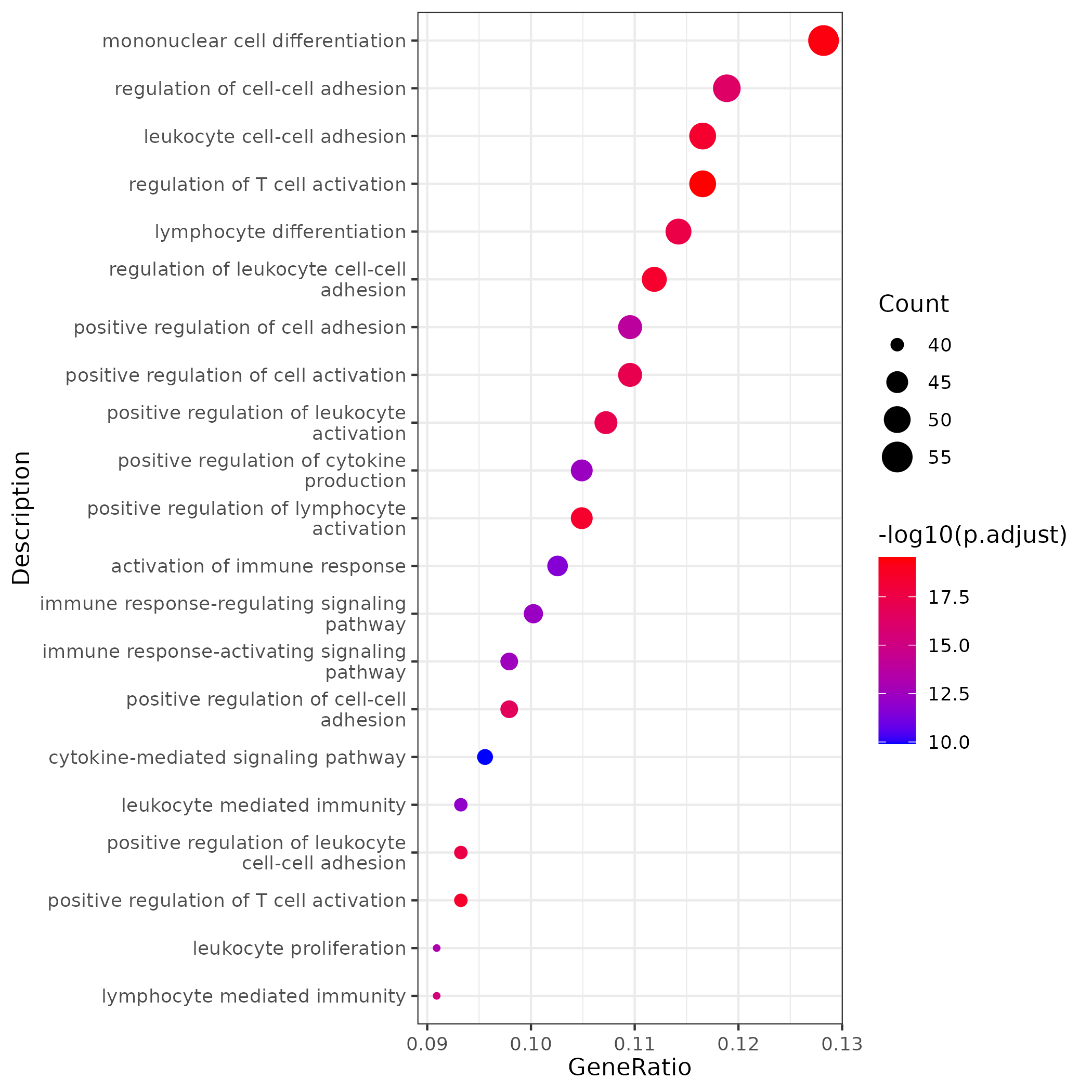
- 图表解读:气泡图展示了某个基因模块显著富集的生物学通路。
- 横坐标:GeneRatio,代表该通路下的基因占模块中总基因的比例。
- 纵坐标:富集的通路名称。
- 颜色:代表富集的显著性(p.adjust),颜色越红越显著。
- 点的大小:代表富集到该通路上的基因数目。
- 分析要点:
- 为每个基因模块赋予生物学功能。例如,与干细胞相关的模块可能富集“细胞周期”通路,而与分化细胞相关的模块可能富集与其特定功能相关的通路。
5.5 结果文件列表
| 目录/文件名 | 内容说明 |
|---|---|
trajectory/ | 轨迹分析核心结果目录 |
*_pseudotime.png/pdf | 拟时序值在UMAP上的分布图。 |
*_trajectory.png/pdf | 叠加了细胞类型的轨迹图。 |
*_genetop10.png/pdf | Top10动态变化基因随拟时序的表达趋势图。 |
gene_module/ | 基因模块分析结果目录 |
*_gene_module.xls | 每个基因所属的模块列表。 |
*_gene_module_heatmap.png/pdf | 基因模块在不同细胞群中的表达热图。 |
enrichment/ | 功能富集分析结果目录 |
go/*_module_go.xls | 所有模块的GO富集分析结果表。 |
kegg/*_module_kegg.xls | 所有模块的KEGG富集分析结果表。 |
go/*_barplot.png/pdf | 各模块GO富集结果柱状图。 |
go/*_dotplot.png/pdf | 各模块GO富集结果气泡图。 |
应用案例
案例:解析胰腺癌中的细胞状态转变
- 文献:Cui Zhou D, et al. Nat Genet. 2022.
- 背景:研究者希望了解在胰腺癌发生过程中,正常的腺泡细胞是如何向导管细胞或肿瘤细胞转变的。
- 分析策略:对来自胰腺癌样本的细胞进行Monocle3拟时序分析,并将腺泡细胞指定为轨迹的起点。
- 核心发现:
- 构建了从腺泡细胞出发的两条不同分化路径。
- 一条路径指向正常的导管细胞状态,其中ADM_Normal细胞作为中间过渡态。
- 另一条路径则指向癌前病变细胞(PanIN),其中ADM_Tumor细胞作为中间过渡态。
- 该分析清晰地揭示了胰腺癌发生过程中可能存在的两条平行的、由不同中间状态介导的细胞演进路径。
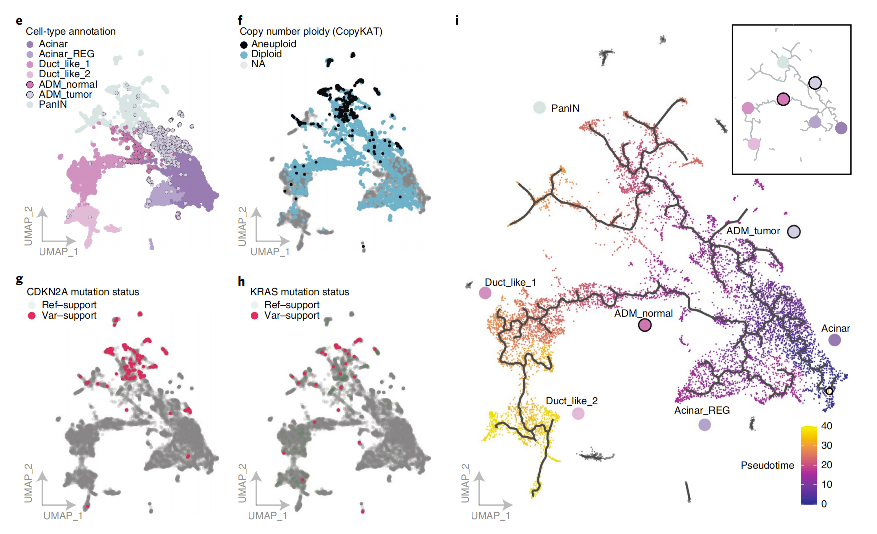 (图注:Monocle3拟时序分析揭示了从腺泡细胞到ADM_Tumor和ADM_Normal状态的独立转变路径。)
(图注:Monocle3拟时序分析揭示了从腺泡细胞到ADM_Tumor和ADM_Normal状态的独立转变路径。)
注意事项
1. 避免过度解读:拟时序轨迹是基于转录组数据的计算推断,不等于真实的细胞谱系。任何关键发现都需要后续的生物学实验(如谱系示踪、功能验证)来证实。
2. 起点选择至关重要:Monocle3的结果完全依赖于用户指定的起点。错误地指定起点将导致整个轨迹的方向和结论都是错误的。务必基于充分的生物学先验知识来选择起点细胞群。
3. 关注基因模块:相比于单个基因,基因模块能更稳健地反映生物学功能。应重点分析与关键细胞状态或轨迹分支相关的基因模块,并对其进行深入的功能富集分析。
4. 结果不是一成不变的:轨迹的构建受到上游降维和聚类的影响。如果轨迹结果不符合预期,可以尝试调整UMAP的参数或聚类的分辨率,这可能会改善轨迹的形态。
常见问题解答(FAQ)
Q1: Monocle3 和 Monocle2 我应该如何选择?
A:
- 选择Monocle3:当您的数据集非常大(>10万细胞),或者您预期存在多个独立的、不相连的分化过程时。Monocle3的速度和可扩展性是其最大优势。
- 选择Monocle2:当您的数据集规模适中,且您预期是一个相对连续、具有清晰分支的树状分化过程时。Monocle2的DDRTree算法在构建清晰的“树”结构方面非常经典和强大。
Q2: 我应该如何选择轨迹的起点(Root cells)?
A: 这是最关键的问题。请遵循以下原则:
- 利用先验知识:根据您的实验设计和生物学背景,选择最“原始”的细胞类型。例如,在发育研究中选择干细胞/祖细胞;在药物刺激实验中选择处理前的细胞(对照组)。
- 利用其他工具辅助:可以使用CytoTRACE等工具预测细胞的分化潜能,得分最高的细胞群通常是很好的起点候选。
- 尝试不同起点:如果不确定,可以尝试选择不同的细胞群作为起点,比较哪种结果更符合已知的生物学事实。
Q3: 为什么我的图中有些细胞是灰色的,没有拟时序值?
A: 这是因为Monocle3首先会将细胞划分为不相连的“分区”(Partitions)。只有和您指定的起点细胞在同一个分区内的细胞才会被计算拟时序。灰色细胞位于其他分区,算法认为它们与您关心的这条轨迹无关。这通常是正常的,但也提示您数据中可能存在多个独立的生物学过程。
Q4: Moran's I 和 Monocle2中的差异表达分析有什么不同?
A:
- Monocle2是寻找随拟时序值变化而显著变化的基因,它关注的是基因表达与“时间”的函数关系。
- Monocle3的Moran's I是寻找在UMAP空间上聚集表达的基因,它关注的是基因表达与“空间位置”的相关性。由于UMAP空间本身就反映了细胞的相似性关系,因此空间共表达的基因通常也就是沿轨迹动态变化的基因,但两者的数学原理不同。
Q5: 我的轨迹图看起来很乱,或者不像我预期的那样,怎么办?
A:
- 检查上游分析:轨迹的质量严重依赖于上游的UMAP降维。尝试调整生成UMAP的参数,生成一个细胞类型分布更合理的UMAP图,再重新运行Monocle3。
- 检查起点选择:确认您的起点选择是否正确。
- 检查细胞群选择:确保您纳入分析的细胞群之间确实存在连续的转变关系。
Q6: Monocle3, CytoTRACE, scVelo有什么区别和联系?我应该如何组合使用?
A: 这三个工具从不同维度解决了细胞动态过程的核心问题,组合使用能构建更完整、更可信的生物学故事。
| 工具 | 核心问题 | 原理 | 优点 | 缺点 |
|---|---|---|---|---|
| Monocle3 | 路径是什么? (What is the path?) | 基于细胞在低维空间(UMAP)的排布,学习主图来重建轨迹。 | 擅长处理大规模、多分支、甚至不相连的复杂轨迹。 | 结果依赖于降维质量和必须手动指定的起点,本身不提供方向性。 |
| CytoTRACE | 起点在哪里? (Where is the start?) | 基于“表达基因的数量与细胞分化潜能成反比”的假设,计算分化潜能得分。 | 提供了一个客观、无偏的方法来识别轨迹的起始细胞群,是验证Monocle3起点选择的黄金标准。 | 仅预测分化潜能,不提供轨迹路径或方向。 |
| scVelo | 方向和速度是什么? (What is the direction & speed?) | 通过RNA速度(spliced/unspliced RNA比例)模型,推断每个细胞未来的转录状态。 | 能揭示细胞状态转变的瞬时方向和速率,为静态的轨迹图赋予了“矢量”信息,能区分循环过程和终末分化。 | 对数据质量要求高(需UMI计数),结果可能受模型假设影响,计算复杂。 |
总结与推荐的工作流:
- 预测起点 (CytoTRACE):首先运行CytoTRACE,在UMAP图上找到分化潜能最高(得分最高)的细胞群。
- 构建轨迹 (Monocle3):运行Monocle3,并将CytoTRACE预测的起点作为Monocle3的“root节点”参数,构建出细胞分化的主要路径。
- 推断方向 (scVelo):在UMAP图上叠加scVelo的RNA速度矢量流,以动态信息验证Monocle3轨迹的方向性,并揭示更精细的细胞状态转变动态。
通过这“三步曲”,您可以构建一个从“确定起点”到“描绘路径”再到“推断方向”的、逻辑严密且证据互补的细胞动态变化故事。
参考文献
- Cao, J., et al. (2019). The single-cell transcriptional landscape of mammalian organogenesis. Nature, 566(7745), 496-502.
- Cui Zhou, D., et al. (2022). Spatially restricted drivers and transitional cell populations cooperate with the microenvironment in untreated and chemo-resistant pancreatic cancer. Nature Genetics, 54(9), 1390-1405.
- Trapnell, C., et al. (2014). The dynamics and regulators of cell fate decisions are revealed by pseudo-temporal ordering of single cells. Nature biotechnology, 32(4), 381-386.
