云平台差异富集分析使用指南
概述
什么是差异富集分析
差异富集分析旨在识别不同细胞群体或条件下显著差异表达的基因,并评估这些基因在功能/通路中的富集,通常分为两步:
- 差异表达分析:发现显著变化基因
- 功能富集分析:解释这些基因的功能与通路背景
NOTE
差异富集分析是单细胞解读的核心环节,务必结合生物学背景合理设置分组与阈值。
生物学意义
差异富集分析在单细胞研究中具有重要价值:
- 细胞功能鉴定:通过差异基因识别细胞类型特异性标记基因,帮助理解不同细胞群体的功能特征
- 生物学过程探索:通过功能富集分析揭示细胞在不同状态下的生物学过程变化
- 疾病机制研究:比较健康与疾病状态下的基因表达差异,发现潜在的疾病相关通路
- 药物靶点发现:识别药物处理前后差异表达的基因和通路,为药物开发提供线索
分析价值
通过差异富集分析,研究人员可以获得:
- 细胞类型标记基因列表,用于细胞注释和功能鉴定
- 活跃的生物学通路信息,揭示细胞功能状态
- 潜在的调控机制,为后续实验提供方向
- 生物学假设,指导深入的功能验证研究
使用说明
新建比较方案开始分析
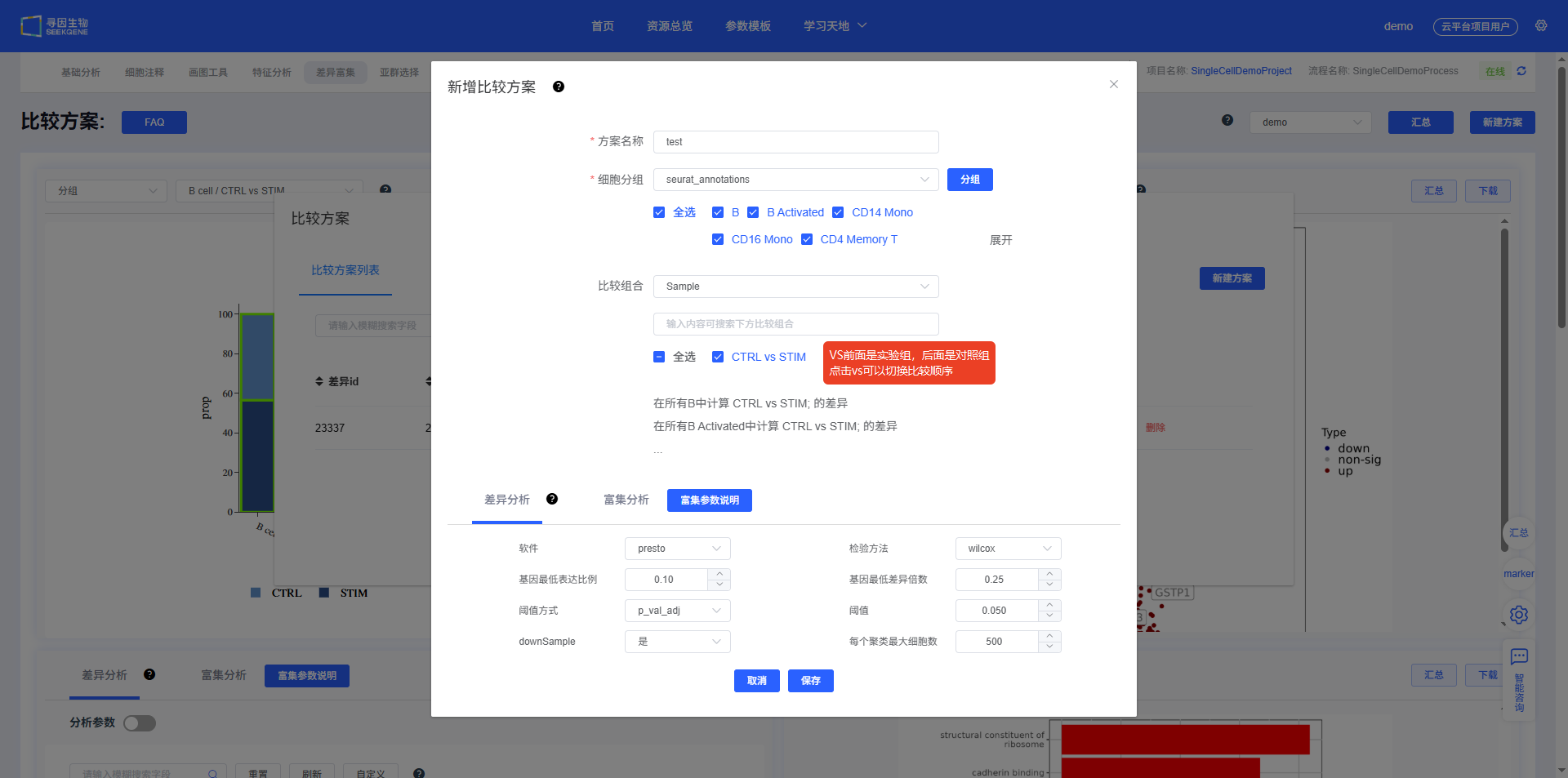
点击页面右上角【新建方案】按钮,弹出比较方案窗口,再点击【新建方案】按钮,弹出新增比较方案窗口。新增比较方案主要包括三部分内容:"方案名称"、"细胞分组"和"比较组合"。
- "方案名称"应填写一个能够清晰反映比较情况、便于区分和查找的名称。
- "细胞分组"选择要分析的对象,可以选择聚类或细胞注释标签,也可以选择样本或样本分组标签,必选。如果没有合适的分组标签,可点击【分组】按钮新建分组标签,建好后即可在细胞分组下拉框中选择新建分组标签。
- "比较组合"选择要进行比较的对象,可以选择聚类或细胞注释标签,也可以选择样本或样本分组标签,非必选。如果比较组合数量超过5组,需要点击【展开】显示全部组合,最多可选择10组比较组合。"vs"前为实验组(比较组),后为对照组(被比较组),点击"vs"可调整比较组合顺序。
创建好比较方案,选择好差异分析和富集分析参数后,点击【保存】即可进行差异富集分析。
比较方案的含义
"细胞分组"选择聚类或细胞注释标签,"比较组合"选择样本或样本分组标签,是2个样本之间同一种细胞类型进行比较。
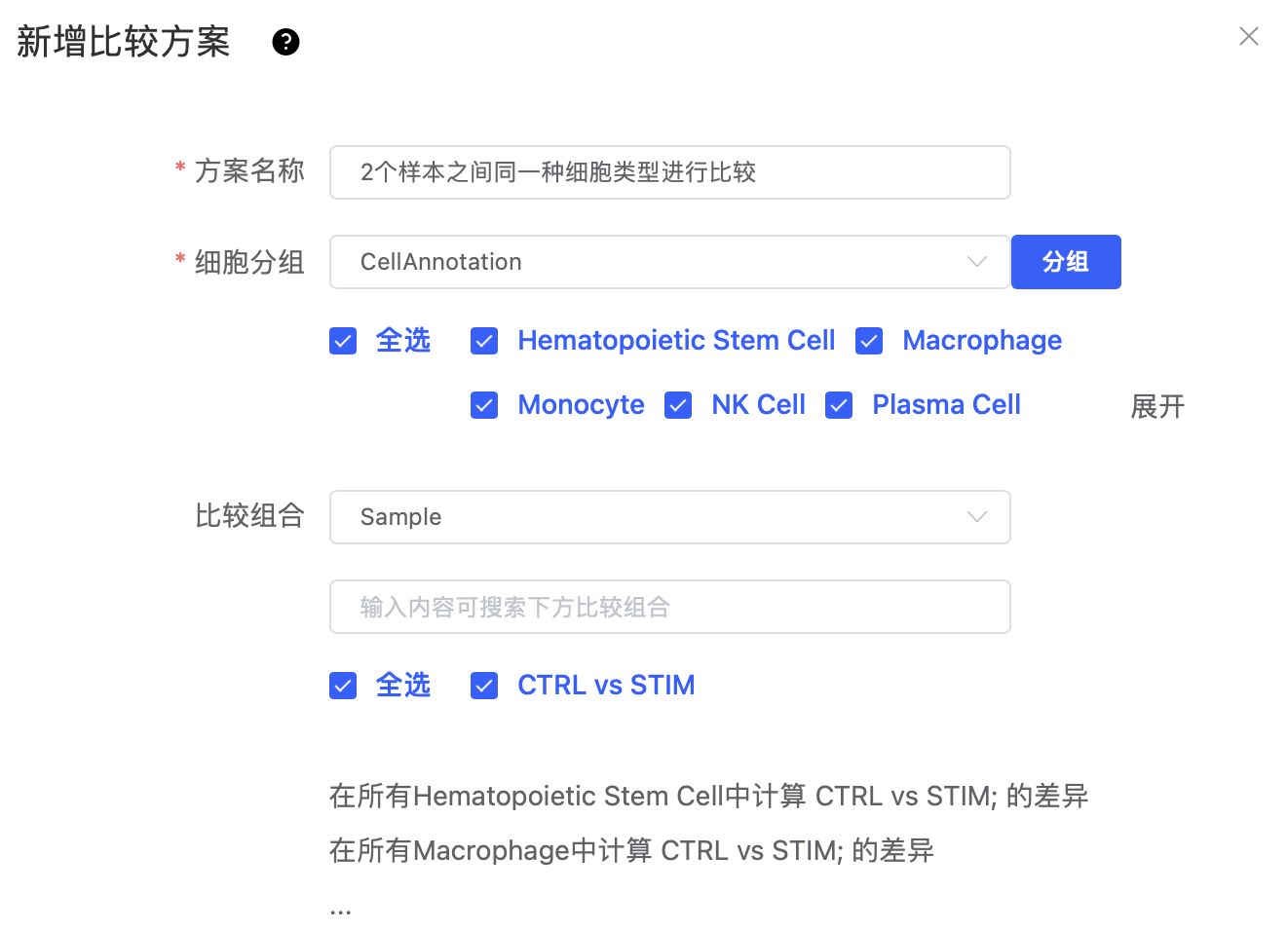
"细胞分组"选择样本或样本分组标签,"比较组合"选择聚类或细胞注释标签,是同一个样本的2种细胞类型进行比较。
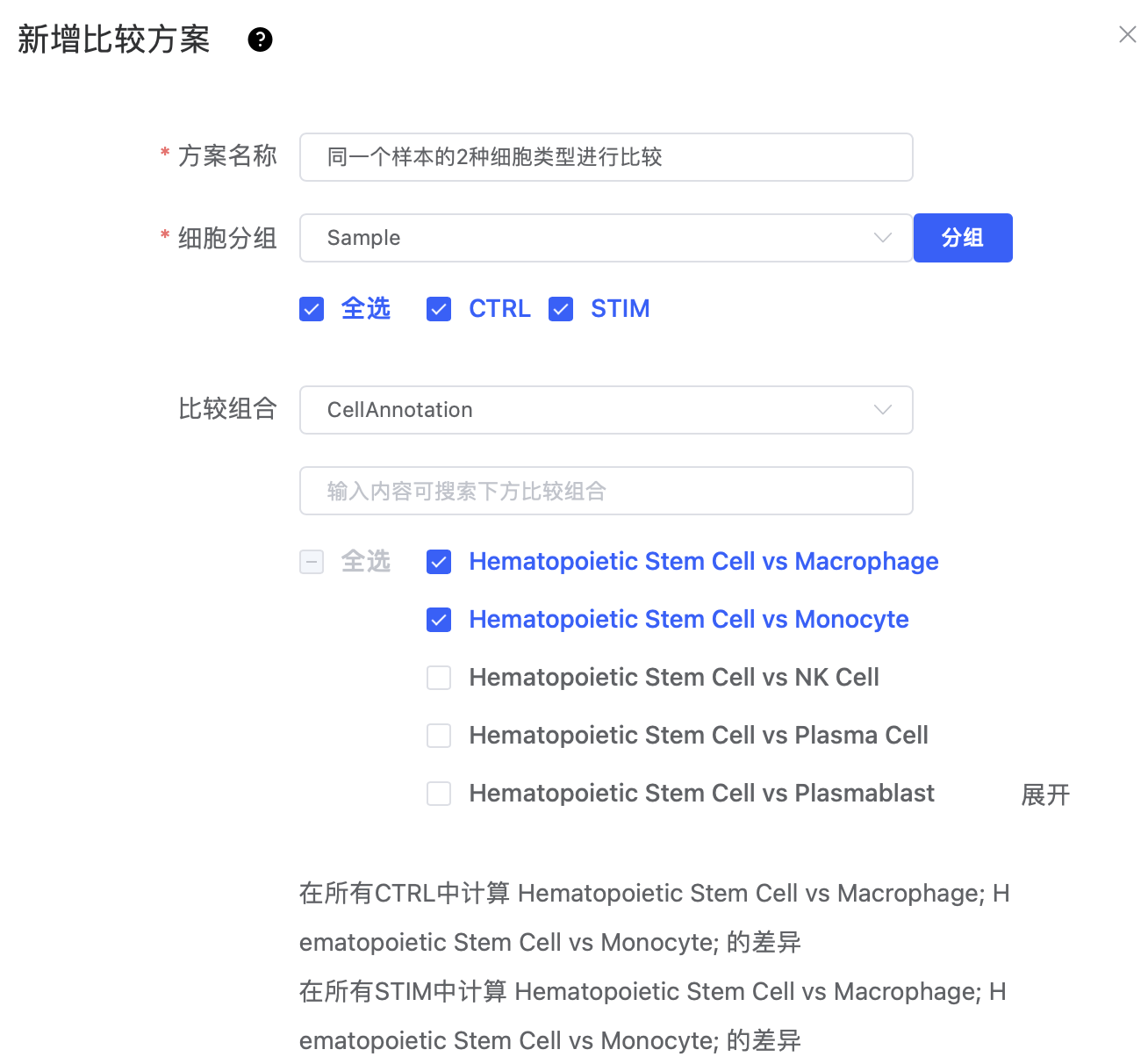
只选择"细胞分组"不选择"比较组合",即可实现每一个细胞分组与其他细胞分组的比较,即1 vs 多。
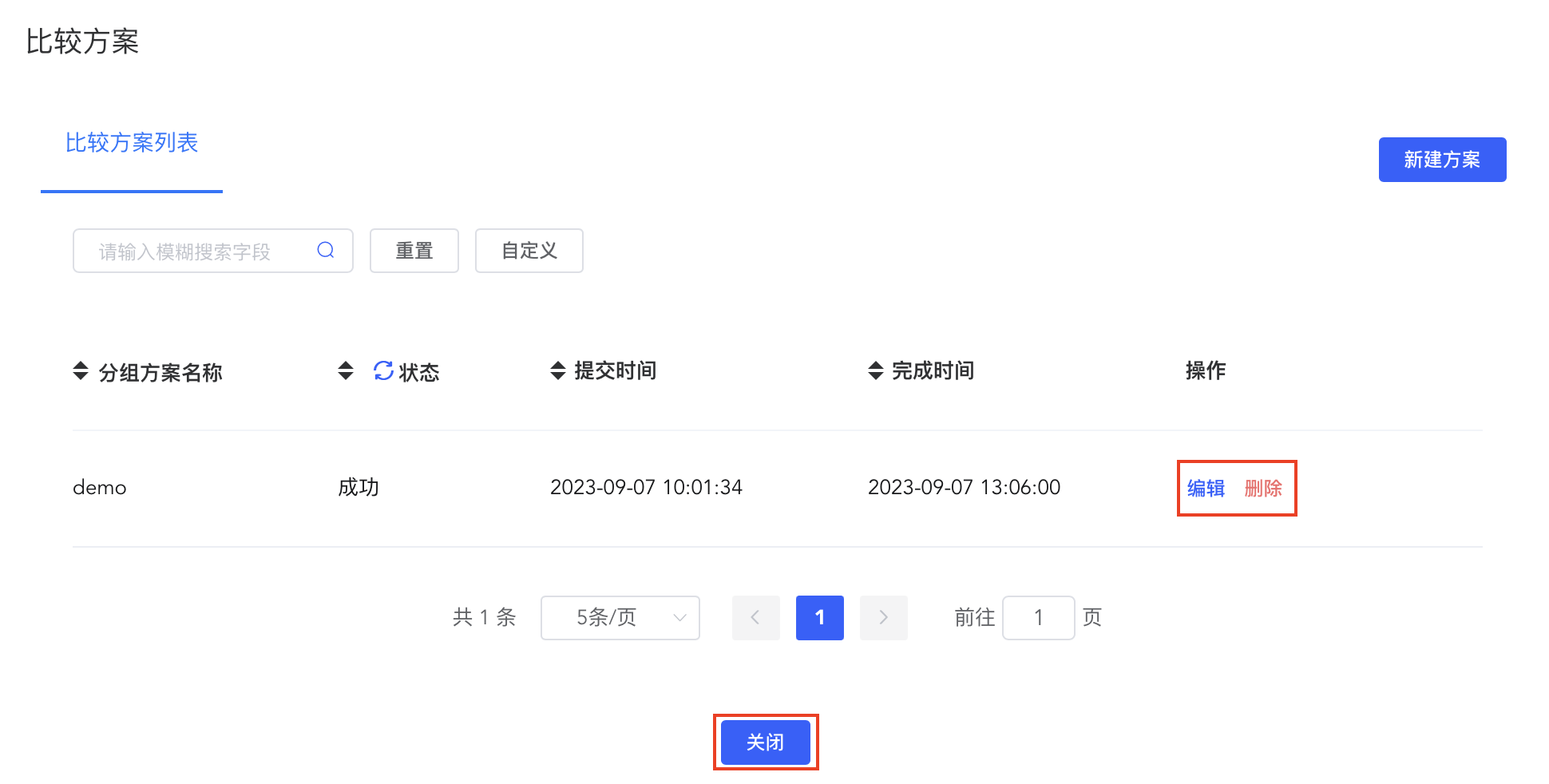
比较方案的修改与删除
比较方案中记录着所有比较方案的详细信息,可点击【编辑】按钮对已有方案进行修改,或点击【删除】按钮对方案进行删除。操作完成后可点击背景或右上方的【关闭】按钮返回主界面。
差异表达分析
分析方法
参考:presto 官方文档,Seurat FindMarkers
| 软件 | 检验方法 | 核心原理 | 优点 | 缺点 | 适用场景与推荐 |
|---|---|---|---|---|---|
| Presto | wilcox | 非参数 Wilcoxon 秩和检验(高效实现,含 auROC 统计) | - 面向大样本高效 - 内存友好 | - 极端稀疏/强不平衡需配合降采样 | ≥1,000 细胞/组的大样本;与 downSample、maxCell 搭配 |
| FindMarkers | wilcox | 非参数检验,比较两组分布中位数与秩次差异 | - 分布假设弱、稳健 | - 对极端不平衡/方差差异敏感 | 默认首选,通用稳健 |
| FindMarkers | bimod | 对数正态的似然比检验 | - 贴合 log-normal 表达 | - 对分布假设敏感 | 表达近似连续且接近对数正态 |
| FindMarkers | roc | ROC/auROC 基于可分性评估标志基因 | - 直观评估可分性 | - 不返回p值;解释偏向可分性 | 标志物筛选、辅助评估 |
| FindMarkers | t | 学生 t 检验,比较两组均值 | - 简单快速 | - 需正态性与方差齐性;对离群点敏感 | 近似正态、样本量适中 |
| FindMarkers | negbinom | 负二项分布检验(计数、过度离散) | - 适配 UMI 计数/过度离散 | - 计算较慢、参数估计复杂 | 原始计数、离散度较高 |
| FindMarkers | poisson | 泊松分布检验(稀疏计数) | - 简单刻画稀疏计数 | - 忽略过度离散 | 稀疏、近似泊松场景 |
| FindMarkers | LR | 似然比检验(嵌套模型比较) | - 可纳入协变量 | - 模型设定要求高 | 需要模型化与协变量时 |
| FindMarkers | MAST | GLM 模型,考虑零膨胀特性 | - 适配零膨胀单细胞表达 | - 计算复杂、耗时 | 存在大量零表达、需严谨建模 |
参数说明
- 基因最低表达比例:设置基因在细胞中表达的最小比例阈值(默认0.1,即10%),表达该基因的细胞比例在任何一组中低于该阈值时,则该基因将不会列入差异基因列表。可以通过过滤低表达的基因加快分析速度。
- 基因最低差异倍数:设置上下调差异倍数的最小绝对值(average Log2 fold change)。默认为0.25,表示仅保留cluster间表达差异倍数log2FC ≥ 0.25和log2FC ≤ -0.25的基因。可根据差异基因数量适当调整该阈值,增加基因最低差异倍数会加快分析速度,但可能会丢失部分基因。该值范围为大于0。
- 显著性阈值方式(p 值/校正 p 值):设定判定基因表达差异是否显著的筛选阈值,默认p_val_adj < 0.05。若差异基因过多,可适当调低阈值,如0.01;若不以该条件筛选,请填写1。p_val_adj范围为[0,1]。
- DownSample:是否进行细胞抽样,默认是。通过降低细胞数量加快分析速度。
- 每个聚类最大细胞数:设置每个细胞群中最大细胞数。针对细胞抽样情况(即downSample等于是),细胞总数高于该阈值的细胞群,随机选择该阈值个细胞进行差异分析,细胞总数低于该阈值的细胞群,则使用所有的细胞进行差异分析。
功能富集分析
分析方法
| 方法 | 函数 | 核心 | 输入 | 输出/可视化 | 适用场景 |
|---|---|---|---|---|---|
| ORA(过度表示分析) | clusterProfiler::enricher | 超几何/Fisher:评估差异基因中的过度表示 | 差异基因列表 + TERM2GENE(GO/KEGG/Reactome/MSigDB) | 富集表;DotPlot、BarPlot | 已有差异基因集合,聚焦显著条目 |
| GSEA(基因集富集分析) | clusterProfiler::GSEA | ES/NES + 置换评估整体协调变化 | 全量有序基因(如按 avg_log2FC)+ TERM2GENE | NES、p.adjust;ES 曲线;Dot/Bar | 信号弱或整体变化,避免硬阈值 |
数据库与数据集
- 系统支持包括人、小鼠、大鼠、斑马鱼、果蝇、线虫、猫、狗、牛、鸡、马、猕猴、猪、鸭嘴兽、负鼠、绿安乐蜥、爪蟾、酵母、黑猩猩等上百个物种的数据库。可以参考我的数据库查看感兴趣的物种。
- 数据库与数据集需根据物种和研究目的灵活选择,如功能注释优先GeneOntology,通路分析可选KEGG或Reactome。
| 数据库 | 可选数据集 |
|---|---|
| GeneOntology | Molecular Function(分子功能)、Biological Process(生物过程)、Cellular Components(细胞组分) |
| Pathway | KEGG |
| MSigDB | H(Hallmark)、C1(定位基因集)、C2(已知功能集)、C3(调控靶点集)、C4(计算预测集)、C5(本体集)、C6(致癌特征集)、C7(免疫特征集)、C8(细胞类型集)、H_C2_C5(H、C2、C5 联合库) |
| Reactome | Reactome 通路库 |
参数说明
- pvalueCutoff:判定富集显著性的p值阈值,通常需满足p < 0.05。
- qvalueCutoff:校正后p值(FDR)的显著性阈值,通常需满足p < 0.05(大规模数据可降低qvalueCutoff至0.01))。
- minGSSize:最小基因集大小(过滤过小基因集,降低假阳性),默认 10。
- maxGSSize:最大基因集大小(过滤过大基因集,避免广谱功能干扰),默认 500。
结果解读
差异表达分析结果
顶部:通过左上角“分组/比较”与方案下拉框联动控制;柱状图中点击高亮单元可快速定位到右侧对应结果。
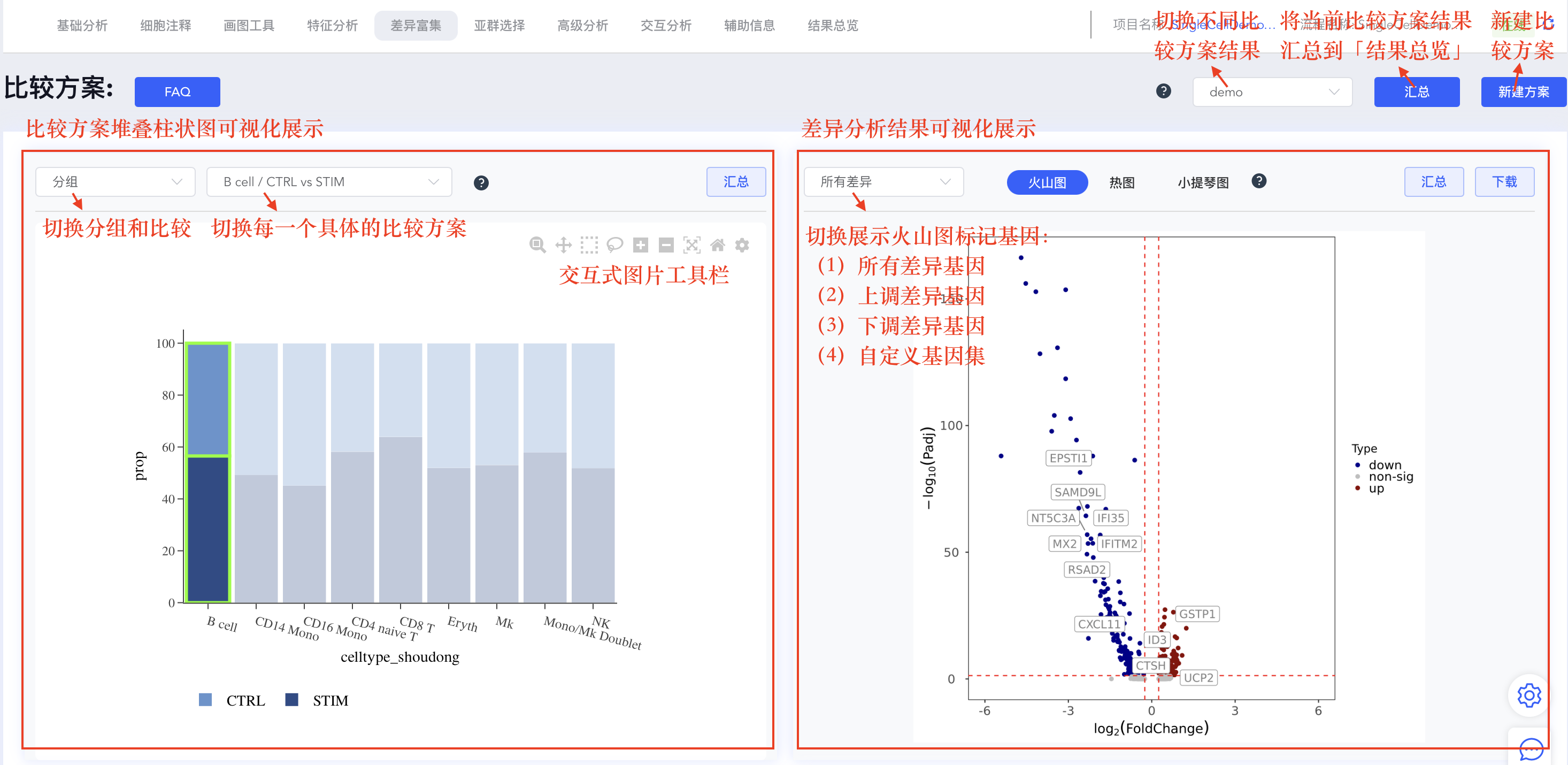
火山图
火山图是差异表达分析结果的重要可视化工具:
- X轴:对数倍数变化(log2FC)
- Y轴:-log10(p值)
- 颜色:
- 红色:显著上调基因
- 蓝色:显著下调基因
- 灰色:无显著差异基因
- 阈值线:垂直虚线表示倍数变化阈值,水平虚线表示显著性阈值
热图
热图显示差异基因在不同细胞群体中的表达模式:
- 行:差异表达基因
- 列:细胞群体
- 颜色:基因表达水平(红色表示高表达,蓝色表示低表达)
- 聚类:基因和细胞群体的聚类结果
小提琴图
小提琴图显示基因表达在不同细胞群体中的分布:
- X轴:细胞群体
- Y轴:基因表达水平
- 形状:表达分布的密度曲线
- 箱线图:中位数、四分位数等统计信息
底部:整体火山图与整体气泡图(随“分组/比较”视角切换做聚合展示)。
| 视角 | 图片 |
|---|---|
| 分组 | 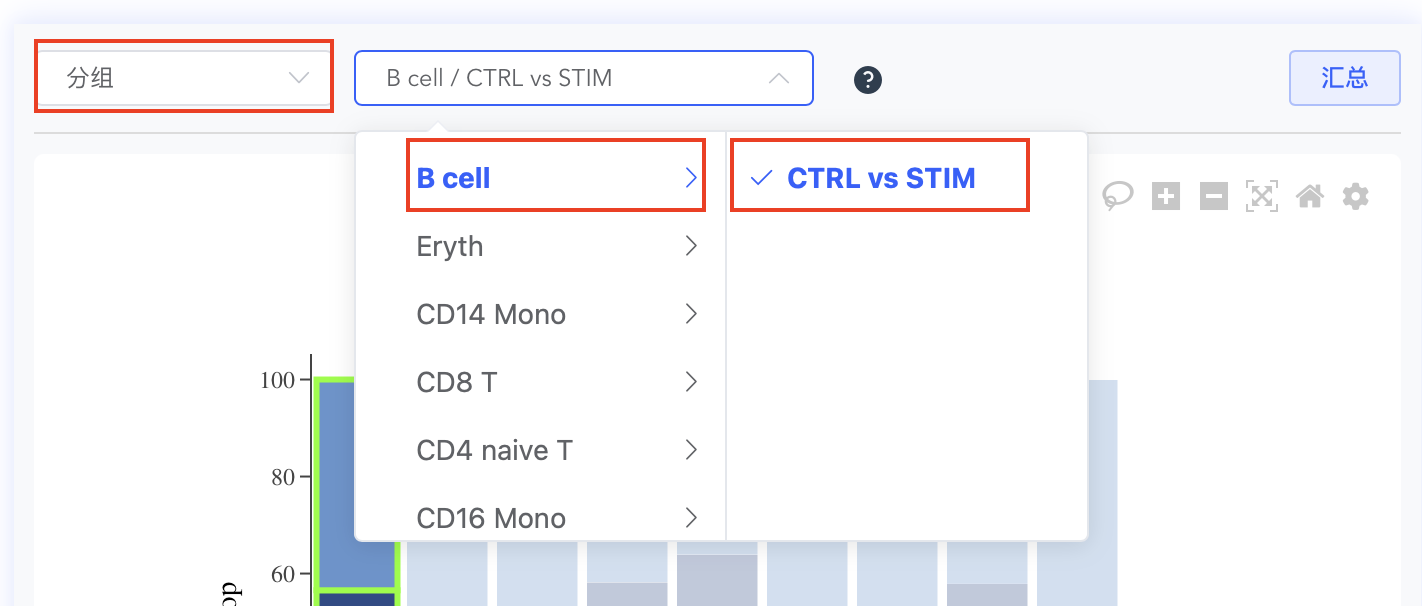 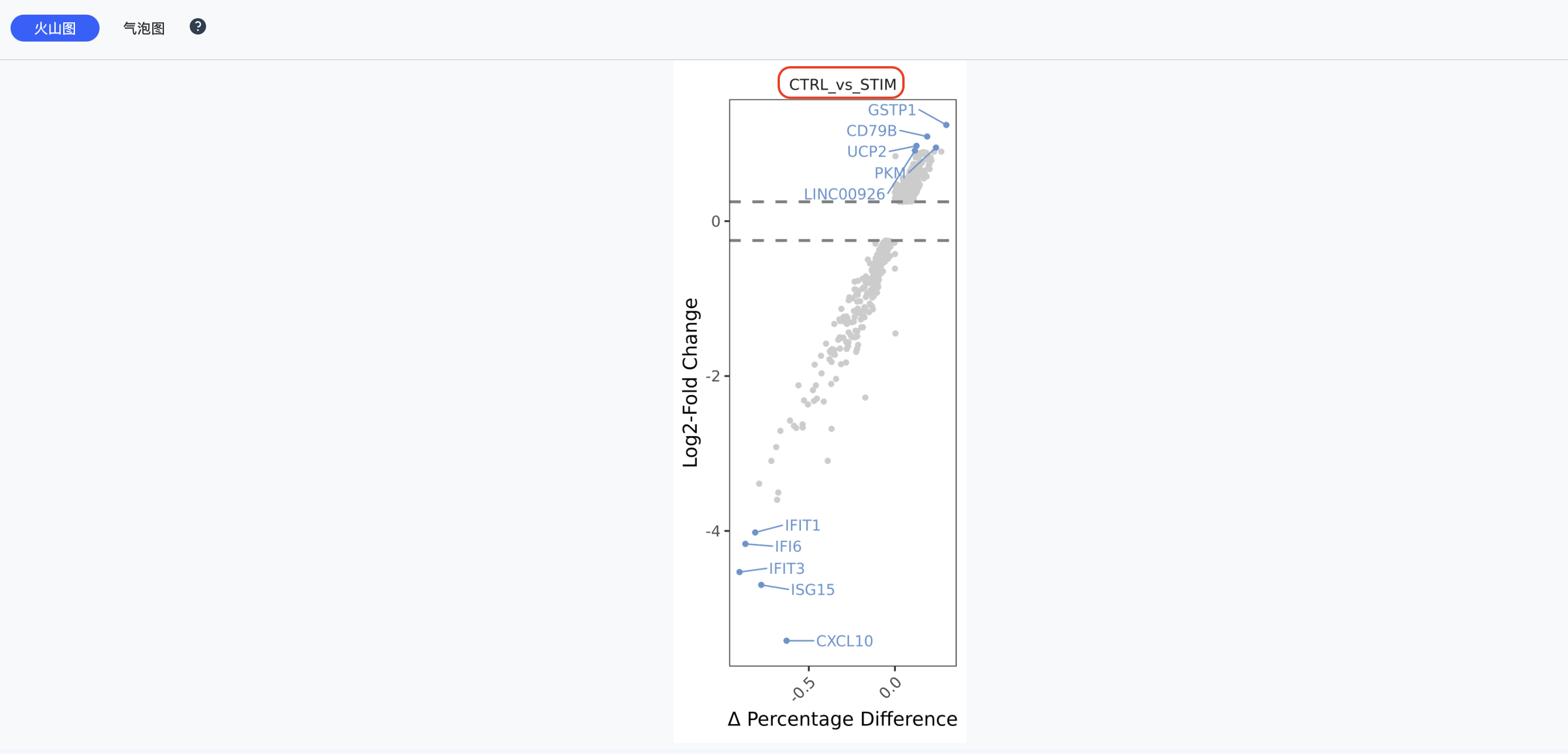 |
| 比较 | 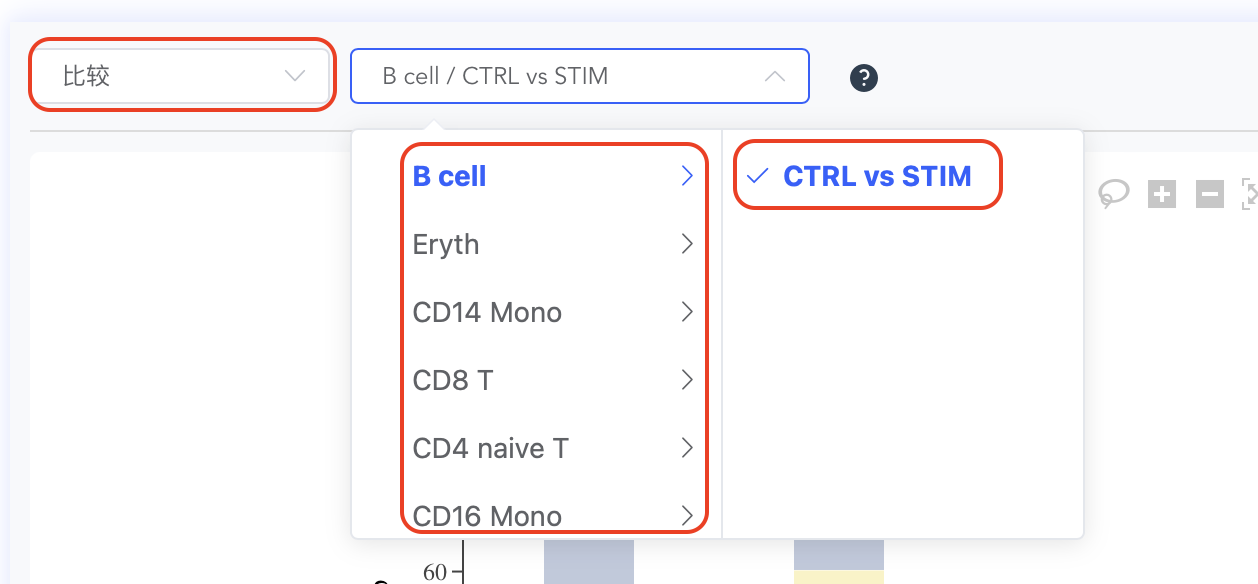 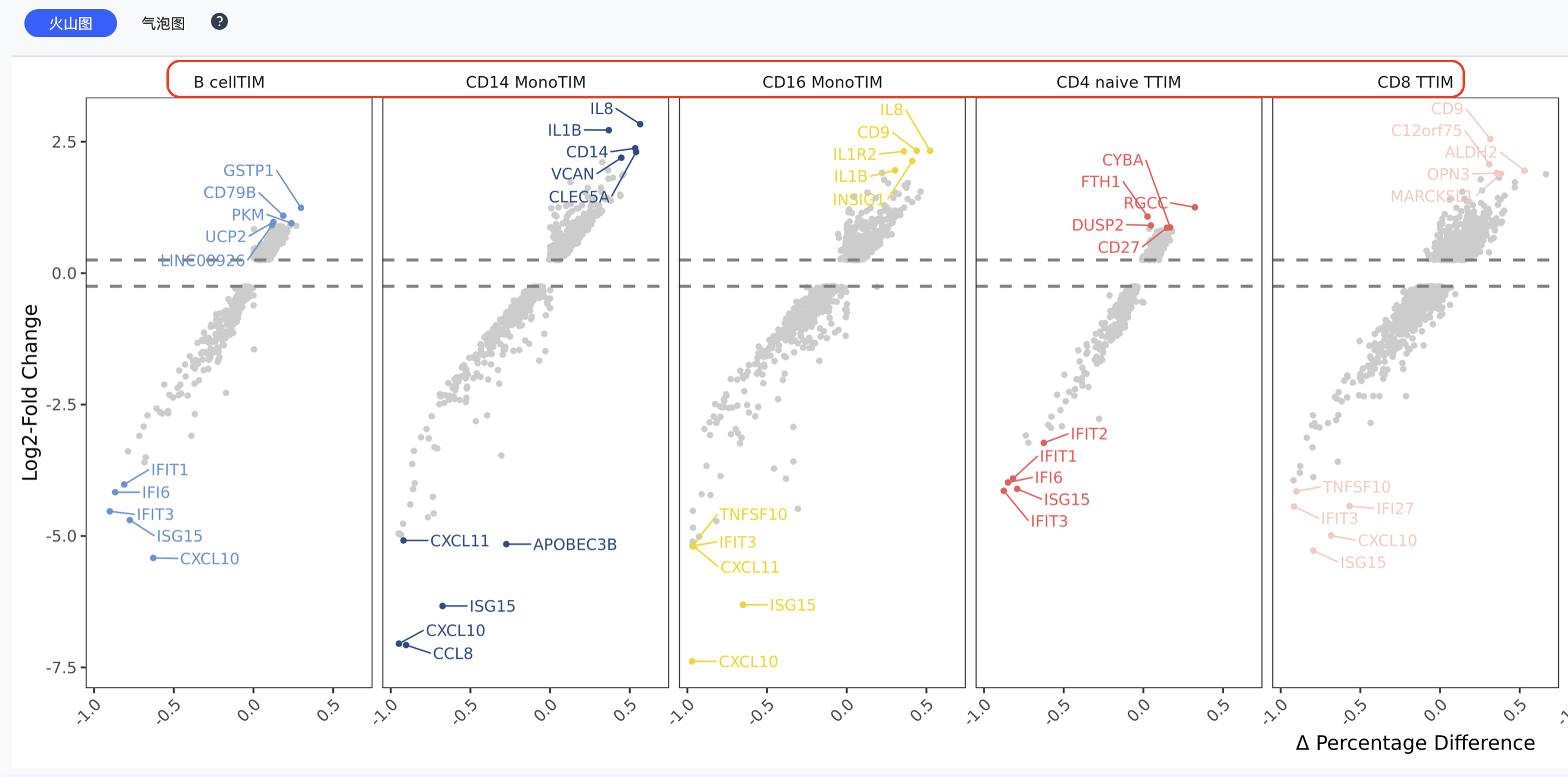 |
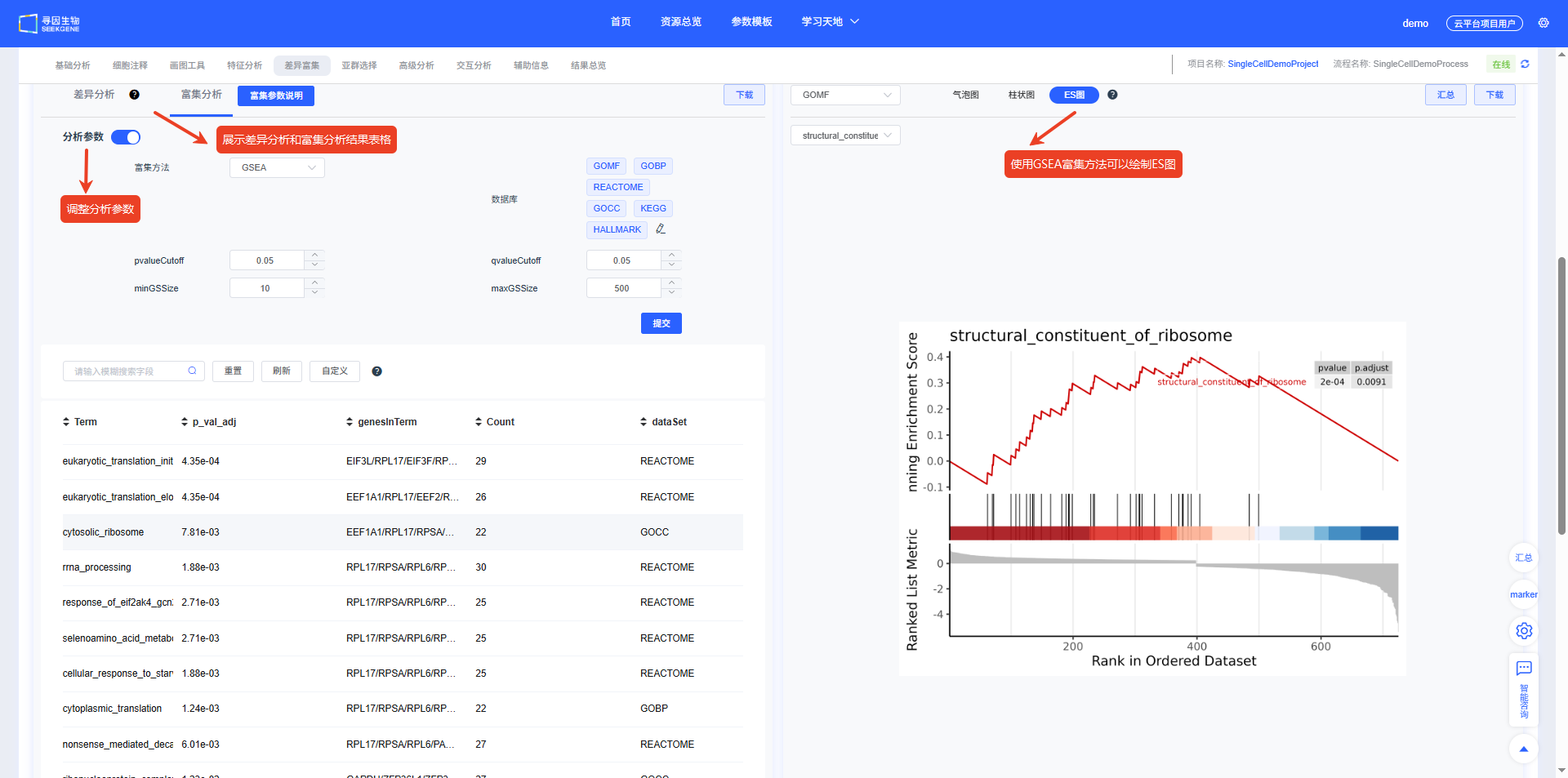
富集分析结果
中部:
- 左侧表格:差异分析和富集分析结果表格,支持搜索、筛选、排序;点击【分析参数】可修改检验方法/阈值并重算。点击下载可下载差异分析或富集分析的表格。
- 右侧图区:富集 DotPlot、BarPlot、ES 图(按所选数据库与通路关注集展示)。
气泡图
气泡图是富集分析结果的主要可视化形式:
- X轴:基因比例(GeneRatio)
- Y轴:富集通路名称
- 气泡大小:富集基因数量
- 颜色:显著性水平(p值或q值)
条形图
条形图显示富集通路的排名和显著性:
- X轴:富集评分或基因数量
- Y轴:富集通路名称
- 颜色:显著性水平
- 排序:按富集程度或显著性排序
GSEA 富集评分图
GSEA结果包含富集评分图:
- X轴:基因排序位置
- Y轴:富集评分
- 曲线:富集评分曲线
- 垂直线:富集基因的位置
- 热图:基因表达水平
TIP
差异富集结果可结合画图工具进一步可视化,便于结果解读和汇报。
总结
云平台差异富集分析工具提供了完整的分析流程,从差异表达分析到功能富集分析,帮助研究者深入理解单细胞数据的生物学意义。通过合理设置参数和正确解读结果,可以获得高质量的生物学发现。
常见问题解答
Q1:差异分析方法怎么选?
- 默认使用 Wilcoxon(FindMarkers: wilcox),分布假设弱、通用稳健。
- 样本量大(≥1,000/组)或性能要求高:优先 Presto(高效 Wilcoxon/auROC)。
- 计数数据过度离散:考虑 negbinom。
- 零表达多、需建模:考虑 MAST。
- 近似正态、样本量适中:可用 t 检验。
- 需纳入协变量:用 LR(似然比)。
Q2:差异基因太多/太少如何调整?
- 太多:增大 logFC 阈值(如 0.25→0.5)、提高 min.pct(0.05→0.1)、收紧 p_val_adj(0.05→0.01)。
- 太少:减小 logFC 阈值、降低 min.pct、放宽 p_val_adj(0.05→0.1 或 1)。
- 大样本偏慢:开启 downSample,并设置 max.cells.per.ident(如 3,000–10,000)。
Q3:p 值还是校正 p 值?
- 推荐优先使用 校正 p 值 p_val_adj ≤ 0.05;多组比较或通路众多时可更严格(≤0.01)。
- 若仅做探索且希望不过滤,可临时将阈值设为 1。
Q4:ORA 和 GSEA 什么时候用?
- 已有明确的差异基因集合、聚焦显著条目:用 ORA(enricher)。
- 整体信号弱或担心阈值筛选丢信息:用 GSEA(按全基因排序)。
- 数据库选择:功能注释优先 GO(BP/MF/CC);通路分析常用 KEGG/Reactome;也可选 MSigDB 子集。
Q5:运行很慢怎么办?
- 选择 Presto;启用 downSample 并设置 max.cells.per.ident;
- 缩小数据库/通路集合范围;
- 检查方案是否包含过多比较组合,必要时分批执行。
Q6:结果与预期不一致怎么排查?
- 核对“分组/比较”是否选对标签与方向(vs 左侧为实验组)。
- 查看是否开启抽样导致波动;调整阈值后重算对比。
- 关注批次/个体差异,必要时改用更稳健方法或提高阈值。
Q7:富集数据库没找到对应物种?
- 平台内置上百个个常见物种库;如未覆盖,请确认物种设置是否正确,或自行上传新建数据库,详细操作参考我的数据库。
TIP
建议结合生物学背景合理设置参数,并在页面中使用“分析参数”进行重算与对比;解读页面提供的图表与结果。
