SCENIC分析技术培训文档
前言
IMPORTANT
SCENIC(Single-Cell rEgulatory Network Inference and Clustering)是单细胞转录组学中用于推断基因调控网络和细胞状态的重要工具。它能够帮助研究者理解细胞异质性背后的转录调控机制,识别细胞类型特异性的调控因子。
在单细胞研究中,我们不仅关注细胞的分类和标记基因,更希望深入了解驱动细胞状态转变的转录调控网络。SCENIC通过整合转录因子与靶基因的共表达关系以及DNA motif分析,构建细胞特异性的调控网络,为解析细胞功能和发育提供重要线索。
SCENIC的核心功能
- 基因调控网络推断:基于基因共表达模式推断转录因子与靶基因的调控关系
- 调控活性评估:通过AUCell算法计算每个细胞中调控子(Regulon)的活性状态
- 细胞状态识别:基于调控网络活性对细胞进行分组和注释
- 调控子特异性分析:识别在特定细胞类型中特异性活跃的调控因子
本篇文档旨在为单细胞研究者提供一份详尽的SCENIC技术指南,内容涵盖其基本原理、在SeekSoulOnline云平台上的操作方法、结果解读、实战案例及常见问题,帮助您快速掌握并应用该工具。
SCENIC理论基础
核心原理
SCENIC的核心思想是:通过识别转录因子与其靶基因的共表达模块,并结合DNA motif分析验证调控关系,构建细胞特异性的基因调控网络。这一过程可以概括为三个主要步骤:
- 基因调控网络推断 (GRN inference):使用GENIE3或GRNBoost算法基于基因表达相关性推断转录因子(TF)与潜在靶基因之间的调控关系
- 调控子修剪与富集分析 (Regulon pruning and enrichment):利用RcisTarget对推断的调控关系进行motif富集分析,去除假阳性调控关系,形成可靠的调控子(Regulon)
- 调控子活性评估 (Regulon activity scoring):使用AUCell算法计算每个细胞中每个调控子的活性状态(AUC值)
关键算法详解
1. GENIE3/GRNBoost - 基因调控网络推断
- 原理:基于随机森林或梯度提升算法,计算每个转录因子与其潜在靶基因表达模式的相关性
- 输出:TF-target调控关系及重要性得分(importance),得分越高表示调控关系越可靠
2. RcisTarget - motif富集分析
- 原理:基于已知的转录因子结合位点数据库,对推断的调控关系进行motif富集分析
- 作用:验证调控关系的可靠性,去除间接调控和假阳性结果
- 输出:经过修剪的调控子(Regulon),包含TF、靶基因及其motif信息
3. AUCell - 调控子活性评估
- 原理:基于基因集富集分析(GSEA)思想,计算每个细胞中调控子靶基因集合的富集程度
- 输出:每个细胞中每个调控子的活性得分(AUC值),用于后续分析
调控子特异性分析指标
1. RSS (Regulon Specificity Score)
- 定义:调控子在特定细胞类型中的特异性得分
- 计算:基于AUC值在不同细胞类型间的分布差异
- 应用:识别细胞类型特异性的关键调控因子
2. Z-score
- 定义:调控子在特定细胞类型中的标准化活性得分
- 计算:基于调控子在目标细胞类型与其它细胞类型间AUC值的差异
- 应用:量化调控子在不同细胞类型中的相对活性
云平台操作指南
在云平台上,SCENIC分析流程被设计得直观易用。您无需编写代码,只需通过参数配置界面即可完成分析。 
分析前的准备
IMPORTANT
SCENIC分析的成功与否,很大程度上取决于输入数据的质量和生物学问题的合理性。在开始分析前,请务必确认:
- 数据已完成预处理:您的单细胞数据已经过标准的质控、降维、聚类和细胞类型注释。
- 选择了合适的细胞亚群:SCENIC分析应在具有生物学意义的细胞亚群中进行,如已注释的细胞类型或功能相关的细胞簇。
- 确保数据规模适中:对于超过数万个细胞的数据集,建议开启Downsample进行分析以避免内存不足。
参数详解
下表详细列出了云平台SCENIC分析模块的主要参数及其说明。
| 界面参数 | 说明 |
|---|---|
| 任务名称 | 本次分析的任务名称,需以英文字母开头。 |
| 物种 | 选择该分析流程数据对应的物种名称,目前支持人、小鼠和果蝇。 |
| 分组因子 | 选择要分析的细胞类型或聚类对应的标签,例如celltype。 |
| 细胞类型 | 多选,选择要纳入分析的具体细胞类型或聚类。 |
| 拆分因子 | 多选,绘图时用于拆分不同组别的标签,如Group或Sample。 |
| Downsample | 是否对大数据集进行随机下采样。 |
| Downsample_num | 每个细胞亚群下采样保留的细胞数目。 |
| 基于历史结果分析 | 选择"Regulon画图",可基于已完成的分析任务重新绘图。 |
| 历史任务名称 | 选择一个已成功的SCENIC任务。 |
| Regulon | 在历史任务结果中,选择要重新绘图的Regulon名称。 |
| 备注 | 自定义备注信息。 |
重要注意事项
CAUTION
- 大数据集处理:当细胞总数超过数万时,若
Downsample参数设置为False,分析可能会因内存不足而失败。强烈建议开启Downsample进行分析。 - Metadata规范:请确保RDS文件中的metadata列名和内容不包含中文或特殊字符(如
&),否则可能导致流程错误。 - 物种匹配:确保选择的物种与实际数据匹配,否则会影响motif数据库的准确性。
操作流程
- 进入分析模块:在云平台导航至"高级分析"模块,选择"scenic"。
- 创建新任务:为您的分析任务命名,并选择要分析的样本或项目。
- 配置参数:根据上述指南,选择要分析的细胞类型、分组信息等。
- 提交任务:确认参数无误后,点击"提交"按钮,等待分析完成。
- 下载与查看:分析结束后,在任务列表中下载并查看生成的分析报告和结果文件。
结果解读
SCENIC的分析报告包含丰富的图表和数据文件,以下是对核心结果的详细解读。
1. 结果文件列表
| 文件名 | 内容说明 |
|---|---|
adj_grn_filter.csv | 共表达模块基于motif分析筛选之后的表格,可查看TFs及其靶基因相关性。 |
reg_cxt.csv | TFs-motif分析结果,根据motifID可搜索motif logo图片。 |
auc_mtx.csv | Regulon在各细胞中AUC打分矩阵文件。 |
auc_thresholds.csv | 判定Regulon在细胞中是否开放的阈值。 |
binary_mtx.csv | auc_mtx.csv文件基于auc_thresholds转换的二进制文件。 |
celltype_rss.csv | 进行分析的细胞群各Regulon特异性得分结果。 |
celltype_rss_top5reg.csv | 进行分析的细胞群筛选特异性得分top5 Regulon。 |
celltype_z.csv | 进行分析的细胞群各Regulon Z值结果。 |
2. 调控子活性可视化
AUC热图
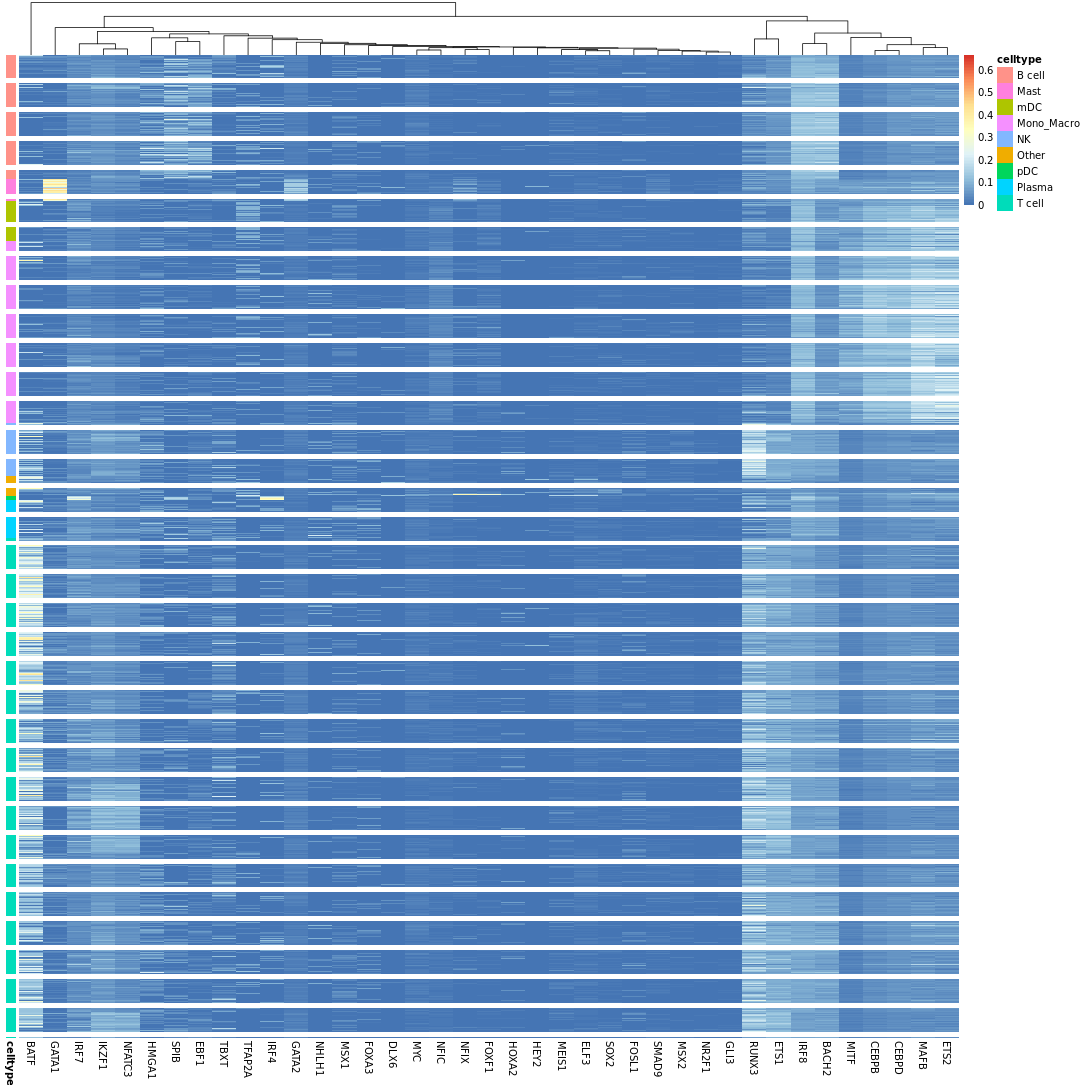
- 图表解读:展示各细胞中调控子活性(AUC)的热图。
- 颜色含义:红色表示高活性,蓝色表示低活性。
二值化热图
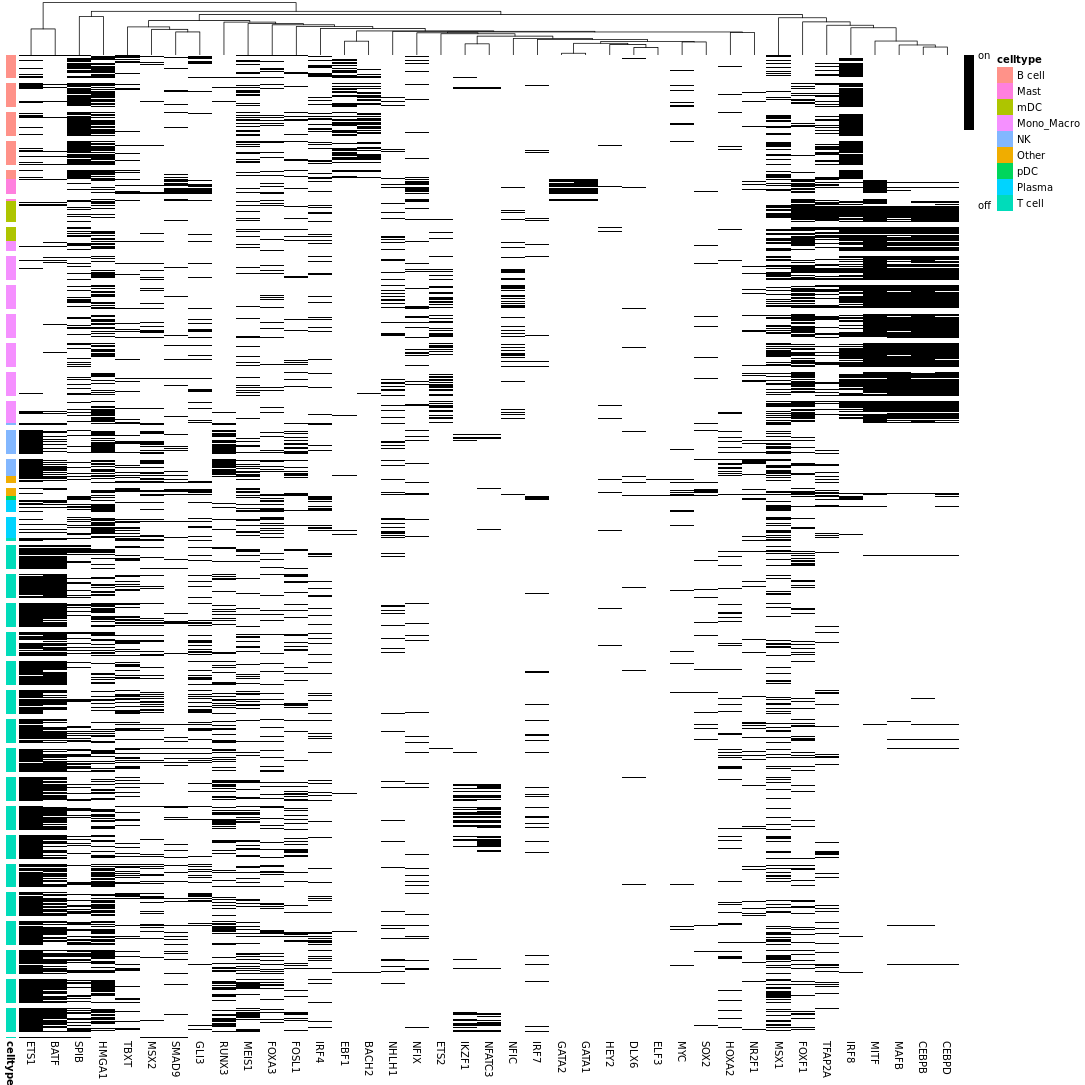
- 图表解读:基于AUC阈值转换的二值化热图。
- 颜色含义:黑色表示调控子处于活跃状态,白色表示调控子处于非活跃状态。
降维图
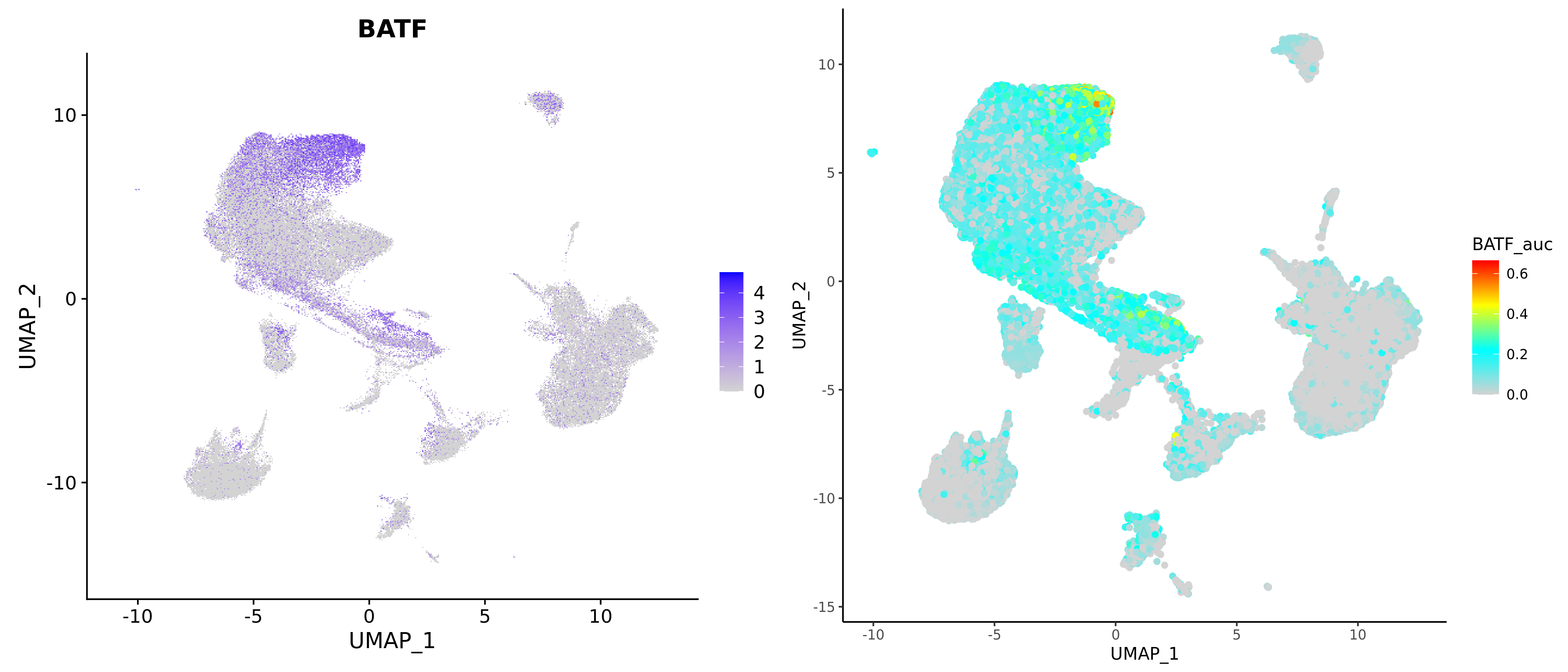
- 图表解读:在UMAP/tSNE图上展示调控子活性的空间分布。
- 左右图对比:
- 左图:调控子靶基因的表达水平
- 右图:调控子活性(AUC)的空间分布
应用案例
案例一:肿瘤微环境中关键转录因子的识别
- 文献:Aibar S, González-Blas CB, Moerman T, et al. Nat Methods. 2017.
- 背景:目标是识别在肿瘤浸润免疫细胞中具有特异性调控活性的转录因子(Regulon),以寻找潜在的治疗靶点或生物标志物。
- 分析策略:在注释好的免疫细胞群(如CD8+ T细胞、巨噬细胞)中运行SCENIC完整流程(GENIE3/GRNBoost → RcisTarget → AUCell),计算每个Regulon在细胞中的AUC并基于RSS得分筛选细胞类型特异性Regulons;将结果与差异表达基因和临床表型(如免疫检查点表达)进行联合分析。
- 核心发现:
- 在肿瘤相关巨噬细胞中识别出若干具有高RSS的调控子(例如STAT3相关Regulon),其AUC在肿瘤样本中显著上调。
- 这些Regulon的靶基因富集于“炎症反应”“免疫抑制”路径,与临床样本的免疫抑制表型一致。
- 部分高RSS的转录因子经过文献回顾也被报道与肿瘤免疫微环境调控相关,支持结果的可靠性。
案例二:发育或分化过程中调控网络的动态重构
- 文献:Van de Sande B, Flerin C, Davie K, et al. Nat Protoc. 2020.
- 背景:研究细胞命运决策期间转录调控网络如何随时间发生重构,识别负/正调控主导因子。
- 分析策略:对不同发育阶段或分化时间点的单细胞数据分别运行SCENIC,比较不同时间点的Regulon AUC矩阵与RSS分布,关注随时间显著变化的Regulon并结合motif信息验证直接调控可能性。
- 核心发现:
- 识别到在分化早期活性高的若干Regulon(如某些干细胞维持相关TF),其AUC随分化进程下降;与此同时出现一组分化终末状态相关的Regulon AUC上升。
- motif富集分析支持部分Regulon为直接调控关系,提示这些TF可能在命运决定中发挥因果作用。
案例三:药物/处理条件下Regulon响应的鉴定与验证
- 文献:Huynh-Thu VA, Irrthum A, Wehenkel L, et al. PLoS One. 2010.
- 背景:评估药物处理对细胞调控网络的影响,筛选出药物诱导或抑制的关键Regulon。
- 分析策略:合并处理组与对照组数据,运行SCENIC并计算每个细胞的AUC;对组间AUC进行差异分析,筛选显著改变的Regulon并通过富集分析和外部数据(如ChIP-seq)验证其可能直接靶向关系。
- 核心发现与验证:
- 发现某些Regulon在处理组中AUC显著下降,提示药物可能通过抑制特定TF活性发挥作用。
- 结合ChIP-seq或公开数据库验证后,确认部分靶基因为该TF的直接靶基因,从而为药物作用机制提供分子证据。
注意事项与最佳实践
TIP
避免过度解读:SCENIC结果是基于转录组数据的计算推断,不等于真实的调控关系。任何关键发现都需要后续的生物学实验(如ChIP-seq、报告基因实验)来证实。
常见问题解答 (FAQ)
Q1: SCENIC分析需要多长时间?
A: 分析时间取决于数据规模和计算资源配置。一般来说:
- 小数据集(1,000-5,000细胞):1-2小时
- 中等数据集(5,000-20,000细胞):2-6小时
- 大数据集(>20,000细胞):6-24小时或更长 建议开启Downsample以加快分析速度。
Q2: NES值和AUC值的意义是什么?
A:
- NES (Normalized Enrichment Score):标准化富集分数,用于评估motif富集的显著性。NES>3通常被认为是显著富集。
- AUC (Area Under the Curve):曲线下面积,用于计算NES,同时也反映了调控子活性水平。
Q3: RSS和Z-score有什么区别?
A:
- RSS (Regulon Specificity Score):调控子特异性得分,衡量调控子在特定细胞类型中的特异性。值越高表示调控子在该细胞类型中越特异。
- Z-score:标准化活性得分,衡量调控子在特定细胞类型中的相对活性。值越高表示调控子在该细胞类型中活性越强。
Q4: 如何验证SCENIC分析结果的可靠性?
A: 可通过以下方式验证结果可靠性:
- 生物学验证:结合已知文献和数据库验证关键调控关系
- 实验验证:通过ChIP-seq、报告基因实验等方法验证关键调控因子
- 交叉验证:使用不同的数据集或分析方法验证结果一致性
参考文献
Aibar S, González-Blas CB, Moerman T, et al. SCENIC: single-cell regulatory network inference and clustering. Nat Methods. 2017 Nov;14(11):1083-1086. doi: 10.1038/nmeth.4463. Epub 2017 Oct 9. PMID: 28991892; PMCID: PMC5937676
Huynh-Thu VA, Irrthum A, Wehenkel L, et al. Inferring regulatory networks from expression data using tree-based methods. PLoS One. 2010 Sep 28;5(9):e12776. doi: 10.1371/journal.pone.0012776. PMID: 20927193; PMCID: PMC2946910
Van de Sande B, Flerin C, Davie K, et al. A scalable SCENIC workflow for single-cell gene regulatory network analysis. Nat Protoc. 2020 Jul;15(7):2247-2276. doi: 10.1038/s41596-020-0336-2. Epub 2020 Jun 19. PMID: 32561888
He Y, Chen Q, Dai J, et al. Single-cell RNA-Seq reveals a highly coordinated transcriptional program in mouse germ cells during primordial follicle formation. Aging Cell. 2021 Jul;20(7):e13424. doi: 10.1111/acel.13424. Epub 2021 Jun 26. PMID: 34174788;PMCID: PMC8282241
Wang JJ, Ge W, Zhai QY, et al. Single-cell transcriptome landscape of ovarian cells during primordial follicle assembly in mice. PLoS Biol. 2020 Dec 22;18(12):e3001025. doi: 10.1371/journal.pbio.3001025. PMID: 33351795; PMCID: PMC7787681
Zhao B, Jiang X. hsa-miR-518-5p/hsa-miR-3135b Regulates the REL/SOD2 Pathway in Ischemic Cerebral Infarction. Front Neurol. 2022 Apr 11;13:852013. doi: 10.3389/fneur.2022.852013. PMID: 35481271; PMCID: PMC9038098
