Scoring基因集打分
前言
TIP
Scoring模块是一个灵活、高效的基因集打分工具,它整合了多种主流的打分算法(如AUCell, UCell, singscore等),能够帮助研究者快速评估一个基因集在所有细胞中的整体表达活性,是探索和验证特定生物学功能(尤其是免疫相关功能)的利器。
在单细胞研究中,我们常常需要回答这样的问题:
- 我感兴趣的某个通路(如“凋亡”或“干扰素反应”)在哪些细胞群中更活跃?
- 我发现的一组特征基因(Marker Gene)是否在某个特定的细胞亚群中整体高表达?
- 相比于对照组,处理组的肿瘤细胞在“上皮间质转换(EMT)”这个功能上有什么变化?
基因集打分(Gene Set Scoring)分析正是为了解决这类问题而设计的。它将单个基因的表达信息整合为通路的活性得分,从而在更高的功能层面理解细胞的状态和异质性。Scoring模块内置了大量与免疫、癌症相关的基因集数据库,并提供了丰富的可视化功能,让研究者可以方便、快捷地进行探索性功能分析。
Scoring 基因集打分理论基础
核心原理
Scoring模块整合了多种算法,用户可以任选其一进行分析:
- AUCell:使用曲线下面积(Area Under the Curve, AUC)来计算输入基因集的关键子集是否在每个细胞的表达谱顶部富集。由于该方法基于基因排序,因此不受基因表达单位和标准化方法的影响。
- UCell:与AUCell类似,也是一种基于排序的算法,使用Mann-Whitney U统计量,计算速度快,结果稳健。
- singscore:同样基于基因表达排序,但采用了不同的计算方式。
- AddModuleScore:Seurat包内置的经典打分方法,通过计算基因集内基因的平均表达水平(并减去随机抽取的背景基因的表达)来打分。
分析时,Scoring模块会使用选定的算法,计算每个细胞在指定基因集上的得分,最终生成一个“功能活性矩阵”(基因集 × 细胞),用于后续的可视化和统计分析。
云平台操作指南
分析前的准备
TIP
基因集打分分析的价值在于将高维的基因表达数据转化为更易于解读的功能活性信息。开始分析前,请确认:
- 数据已完成预处理:您的单细胞数据已经过标准的质控、降维、聚类和细胞类型注释。
- 选择了合适的基因集:基因集的质量直接决定了分析结果的可靠性。您可以选择平台内置的经典通路数据库(如Hallmark, KEGG, GO),也可以根据研究需要上传自定义的基因列表。
- 明确生物学问题:您希望通过打分来回答什么问题?是比较不同细胞类型的功能差异,还是观察处理前后某个通路的变化?清晰的问题将指导您更好地解读结果。
参数详解
下表详细列出了云平台Scoring分析模块的主要参数及其说明。
| 界面参数 | 说明 |
|---|---|
| 任务名称 | 本次分析的任务名称,需以英文字母开头,可包含英文字母、数字、下划线和中文。 |
| 分组因子 | 选择要分析的细胞类型或者聚类对应的标签。例如celltype。 |
| 细胞类型 | 多选,选择要纳入分析的具体细胞类型或聚类。 |
| 物种 | 支持人、小鼠等。选择“user-defined”时,可上传自定义基因集。 |
| 自定义基因集 | 当<物种>选择“user-defined”时,点击【模版下载】,按照模版上传自定义基因集(.gmt格式)。 |
| 分析方法 | 选择基因集打分的算法,包括 AUCell、UCell、singscore 或 AddModuleScore。 |
| 拆分因子 | (非必填)选择一个meta.data中的分类标签,将图形按该标签拆分为不同的子图,便于组间比较。 |
| 降维图 | 使用 umap 或 tsne 图展示打分结果。 |
| 备注 | 自定义备注信息。 |
操作流程
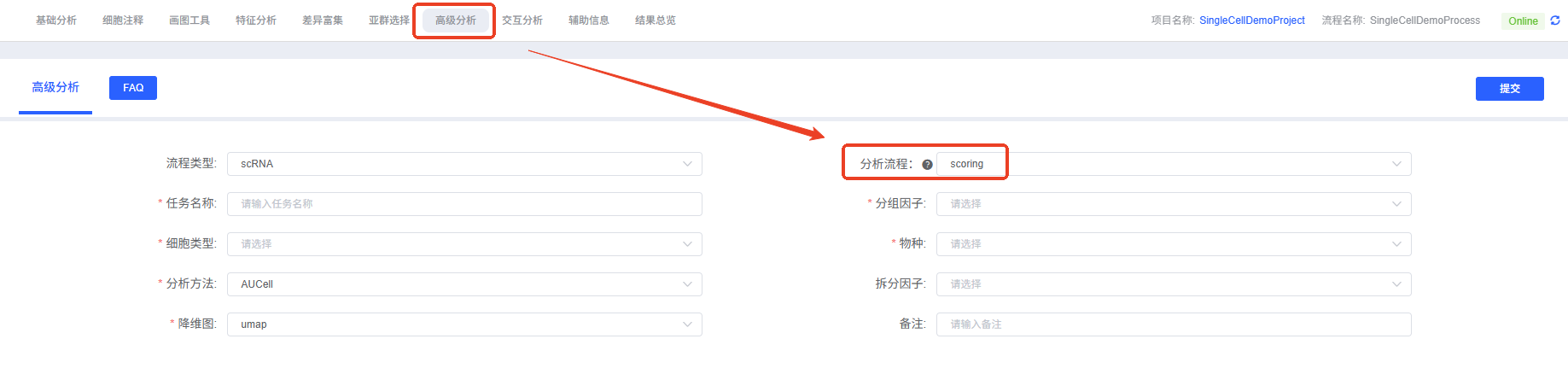
- 进入分析模块:在云平台导航至“高级分析”模块,选择“scoring”。
- 创建新任务:为您的分析任务命名,并选择要分析的样本或项目。
- 配置参数:根据上述指南,选择算法、基因集、要分析的细胞群等。
- 提交任务:确认参数无误后,点击“提交”按钮,等待分析完成。
- 下载与查看:分析结束后,在任务列表中下载并查看生成的分析报告和结果文件。
结果解读
Scoring模块提供了丰富的可视化结果,帮助您从不同维度解读通路活性。
各基因集在不同细胞群间的AUCell得分降维图
这是最直观的结果之一。它将特定基因集的得分映射到UMAP/tSNE降维图上,直观展示该功能在不同细胞群中的富集情况。
- 左图:按细胞群着色,用于定位不同的细胞类型。
- 右图:按基因集得分着色,颜色越深,代表该通路/功能的活性越强。
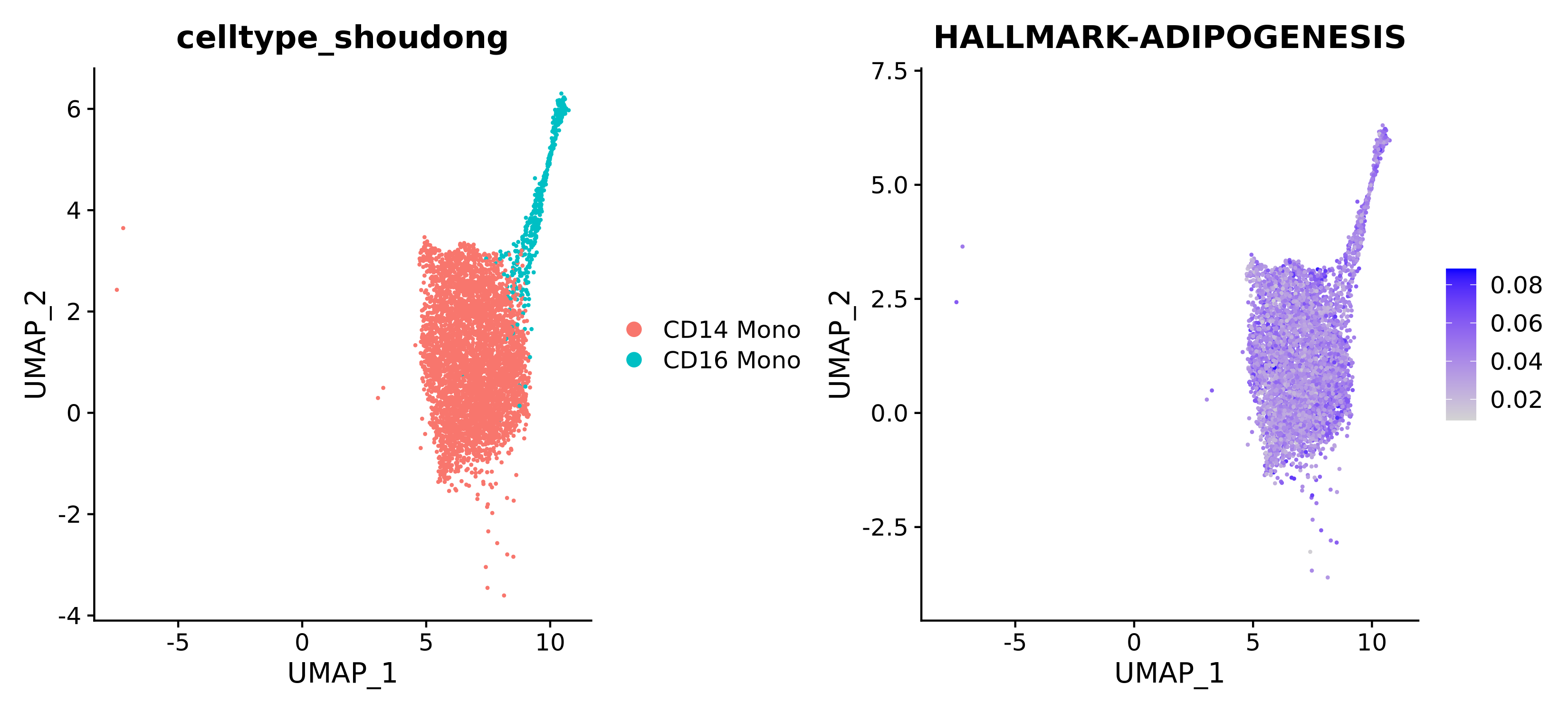
| 注释 | 左图中不同颜色代表不同细胞群,右图中颜色越深代表得分越高,颜色越浅代表得分越低。 |
|---|
各基因集在不同细胞群间的AUCell得分小提琴箱线图
用于精确比较不同细胞群在该基因集上的得分分布。小提琴图展示了得分的密度分布,箱线图则展示了中位数、四分位数等统计信息,便于进行组间差异的统计检验。
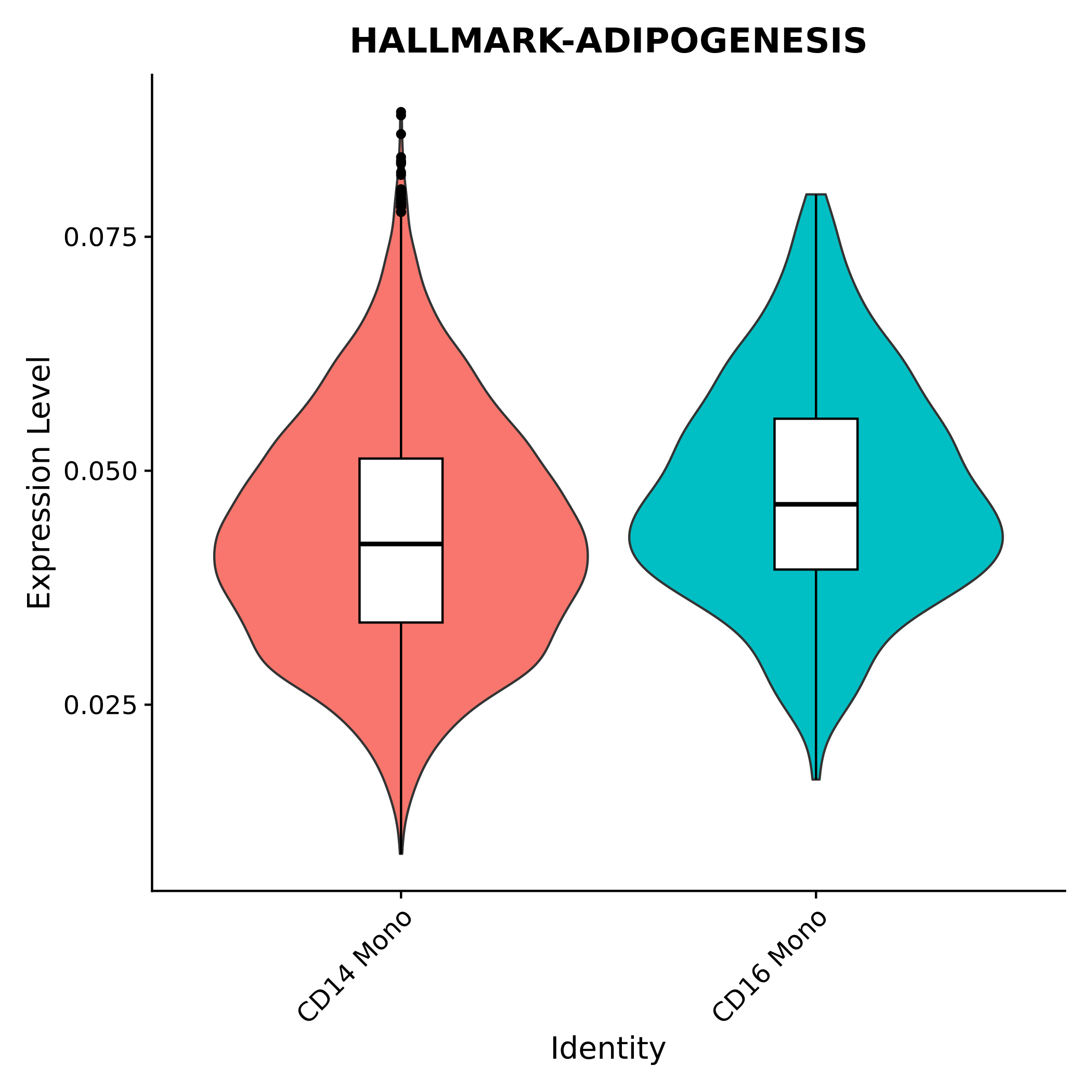
| 注释 | 同时以小提琴图和箱线图展示得分情况,不同颜色代表不同细胞群。 |
|---|
结果文件列表
| 文件名 | 内容说明 |
|---|---|
*.csv | 核心数据:结果数据表格文件,记录了每个细胞在各个基因集上的得分。 |
*_dim.png/pdf | 各基因集在不同细胞群间的得分降维图。 |
*_vln.png/pdf | 各基因集在不同细胞群间的得分小提琴箱线图。 |
split/ | 若设置了拆分因子,此目录下会存放按分组或样本拆分后的各类可视化结果。 |
应用案例
案例:揭示ALDH家族在胶质瘤中的多重作用
- 文献:Wang Z, et al. Front Immunol. 2022.
- 背景:醛脱氢酶(ALDH)家族被认为在多种癌症中扮演重要角色,但其在胶质瘤中的具体功能尚不明确。
- 分析策略:研究者利用单细胞数据,通过计算三个与细胞增殖、迁移和免疫相关的GO通路的AUCell分数,来评估ALDH家族的功能。
- 核心发现:
- 功能活性评估:通过AUCell打分,他们发现ALDH高表达的肿瘤细胞在“细胞增殖”、“细胞迁移”和“免疫应答”相关的通路上得分显著更高。
- 结论:该研究巧妙地利用基因集打分的方法,将复杂的基因表达与特定的生物学功能联系起来,证实了ALDH家族可能通过调节肿瘤细胞增殖、迁移和免疫景观来影响胶质瘤的进展,为后续的功能验证提供了有力证据。
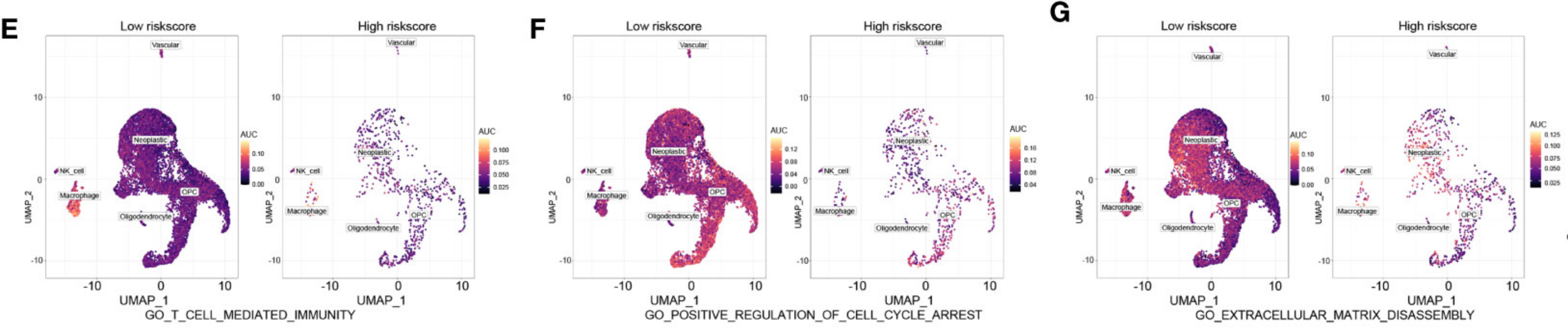 (图注:利用AUCell分数评估ALDH家族在胶质瘤中的功能活性。)
(图注:利用AUCell分数评估ALDH家族在胶质瘤中的功能活性。)
注意事项
1. 算法选择:不同的打分算法各有优劣。AUCell和UCell是目前较为推荐的基于排序的方法,它们对数据标准化的依赖性较低。AddModuleScore则更直观,但可能受高表达基因的影响较大。
2. 基因集选择:结果的生物学意义完全取决于您选择的基因集。请使用来自权威数据库(如MSigDB, KEGG)或文献报道的、经过验证的基因集。对于自定义基因集,需要有充分的生物学理由。
3. 得分是相对值:基因集得分是一个相对值,用于比较不同细胞或细胞群之间的活性高低,其绝对值本身没有太多意义。
常见问题解答 (FAQ)
Q1: Scoring、GSVA、scMetabolism有什么区别?
A: 这三者都是基因集富集或打分的工具,但侧重点不同:
- Scoring:核心是单细胞基因集打分。它为每个细胞计算一个通路活性得分,擅长展示通路活性在不同细胞间的异质性分布。它整合了多种打分算法,尤其适合探索性分析和免疫相关功能研究。
- GSVA:核心是样本/细胞群水平的基因集变异分析。它将基因表达矩阵转换为基因集富集分数矩阵,常用于寻找不同细胞群之间差异显著的通路,适合做差异通路分析。
- scMetabolism:专注于代谢通路的打分和可视化。它内置了KEGG上的所有代谢通路,并提供了多种代谢相关的可视化方法,是研究细胞代谢异质性的专用工具。
Q2: 如何解读AUCell分数?分数高低代表什么?
A: AUCell分数反映了某个基因集在单个细胞的基因表达排序列表中的富集程度。
- 分数高:意味着该基因集中的大部分基因在这个细胞中表达水平相对较高(排在前面),表明该基因集所代表的生物学功能在这个细胞中可能比较活跃。
- 分数低:意味着该基因集中的基因在该细胞中表达水平相对较低,表明该功能可能不活跃。 重要的是比较相对大小,而不是绝对值。例如,可以比较肿瘤细胞和正常细胞的“增殖”通路得分,来判断哪群细胞增殖更活跃。
Q3: 我可以上传自己的基因列表进行分析吗?
A: 可以。您需要将您的基因列表整理成.gmt格式的文件。.gmt文件是一个以制表符分隔的文本文件,每一行代表一个基因集,格式如下: GeneSetName Description Gene1 Gene2 Gene3 ...
- 第一列是基因集名称。
- 第二列是基因集的描述(可以留空)。
- 从第三列开始,是该基因集包含的所有基因ID(需与您数据中的基因ID类型匹配,如Symbol)。 在云平台操作时,将<物种>参数选为“user-defined”,然后上传您的.gmt文件即可。
参考文献
- Aibar S, et al. SCENIC: single-cell regulatory network inference and clustering. Nat Methods. 2017 Nov;14(11):1083-1086.
- Wang Z, et al. The ALDH Family Contributes to Immunocyte Infiltration, Proliferation and Epithelial-Mesenchymal Transformation in Glioma. Front Immunol. 2022 Jan 7;12:756606.
- Fan C, et al. irGSEA: the integration of single-cell rank-based gene set enrichment analysis. Brief Bioinform. 2024 May 23;25(4):bbae243.
