画图工具
“画图工具”提供了添加多条件标签的功能,您可以用它绘制各种图表,包括小提琴图、降维图、FeaturePlot、DotPlot、堆叠柱状图和饼图等。无论是展示特定基因或细胞类型的表达模式,还是探索数据分布,这些图表都能提供直观的可视化效果,助您更好地理解数据并进行进一步分析和解释。
NOTE
“画图工具”支持多种主流可视化类型,建议根据分析目标选择合适的图表类型。
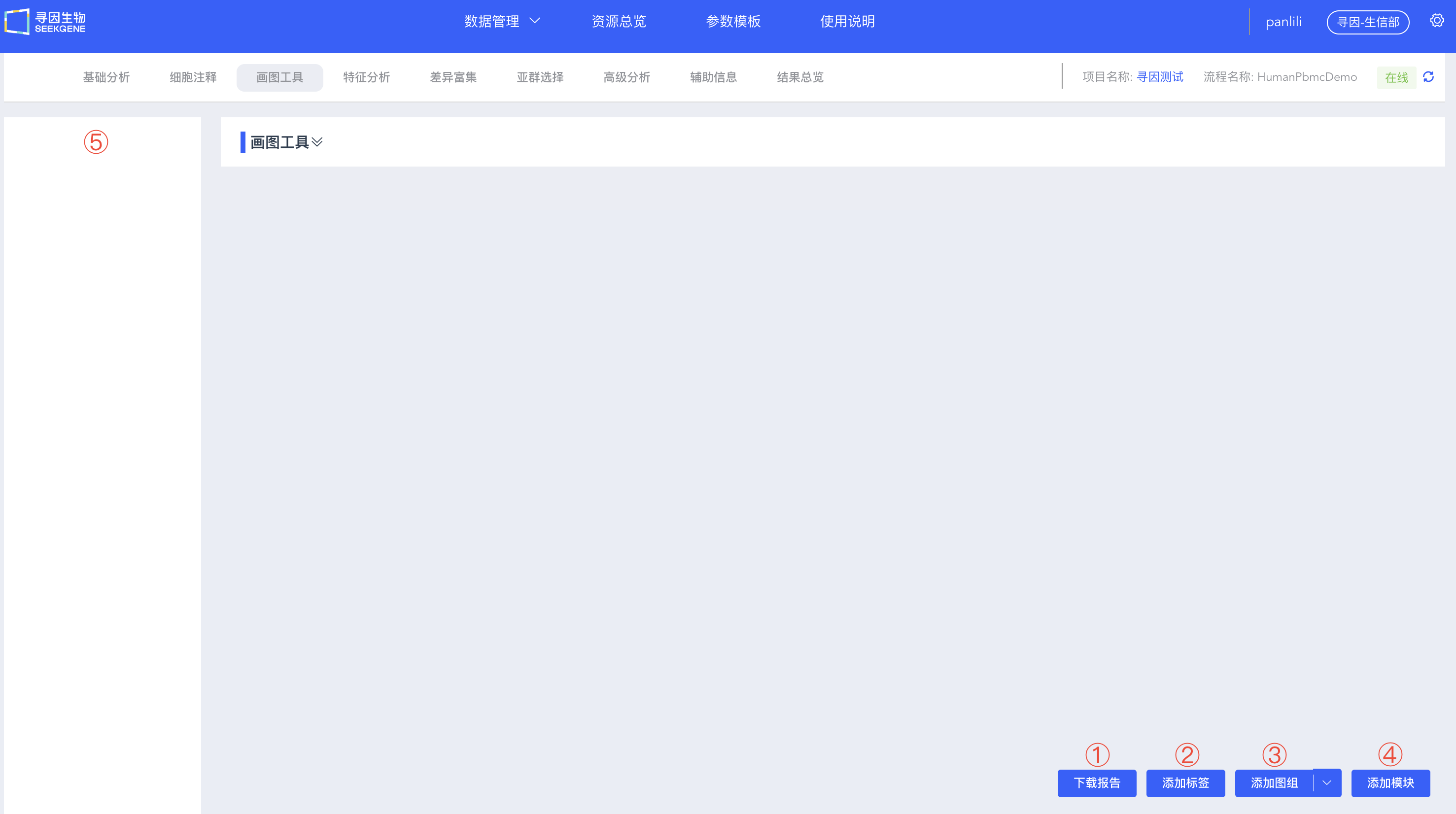
下载报告
点击【下载报告】,即可将“画图工具”的所有图表结果以报告形式下载到本地计算机。请注意,下载过程中可能需要等待大约半分钟时间。
添加标签
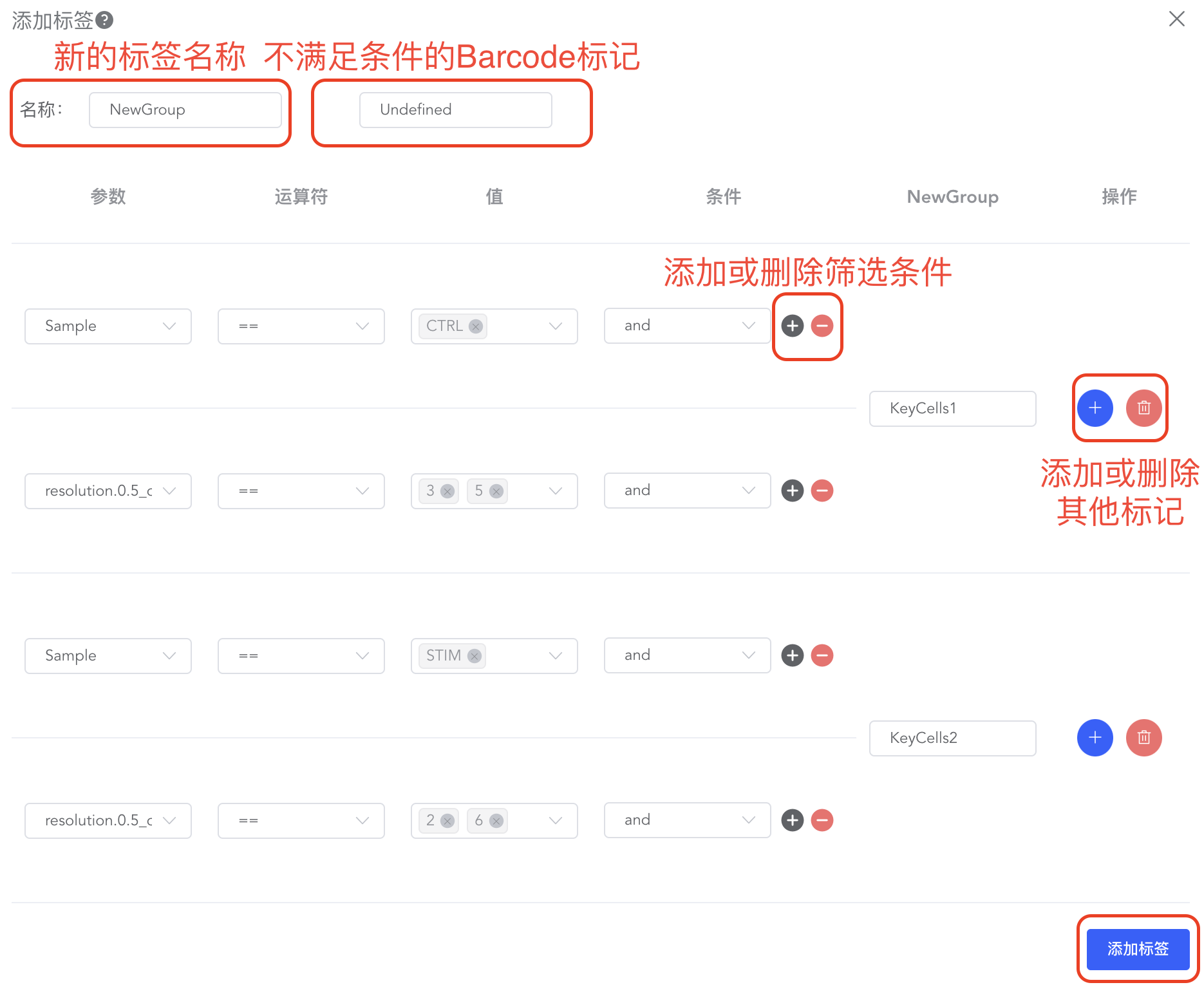
点击【添加标签】,弹出添加标签页面,按照条件对Barcode进行标记。
- 在“名称”处对新标签进行命名。
- “运算符”包括等于“==”和不等于“≠”,“条件”包括交集“and”和并集“or”。通过“运算符”和“条件”设置多个条件共同定位某群关键细胞。
- 不满足条件的Barcode默认标记为“Undefined”,也可自定义标记。
- 点击【添加标签】,完成新标签添加,即可在“画图工具”中根据新标签进行绘图分析。
添加图组
手动添加
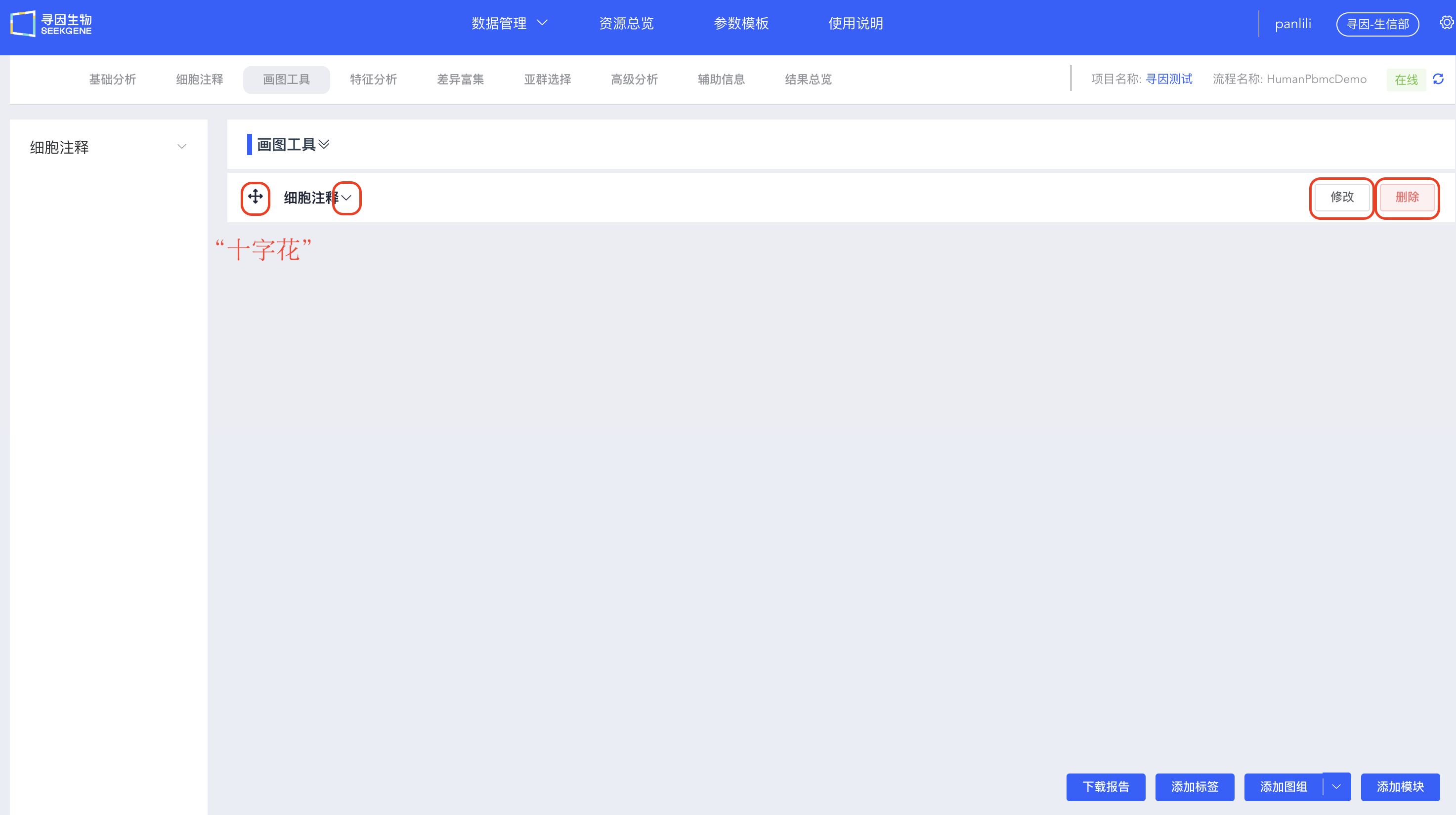
点击【添加图组】,弹出输入图组名称窗口,输入图组名称并点击【确定】,即可在“画图工具”中看到新命名的图组。

长按并上下拖动“十字花”,可调整图组顺序。
点击图组名称右侧的下拉框,展开或收缩图组结果。收缩状态下,长按并拖动“十字花”调整图组顺序。
点击【修改】,弹出输入图组名称窗口,输入新名称并点击【确定】,修改当前图组名称。
点击【删除】,删除当前图组。
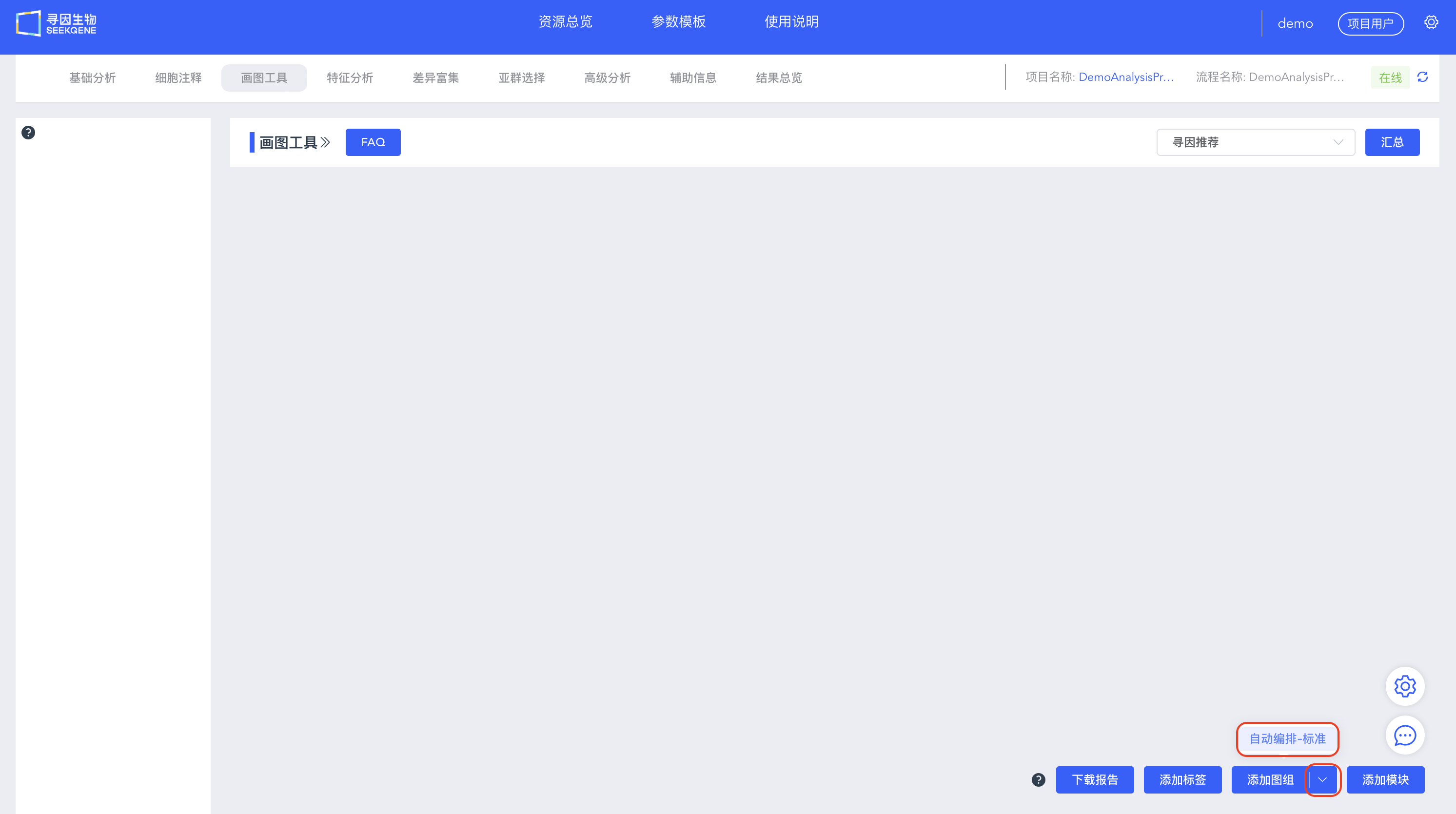
自动编排
点击【添加图组】右侧的下拉框,选择【自动编排-标准】,填写合适参数,自动创建“质控”和“细胞注释”图组,自动绘制降维图、mito、nFeature_RNA和nCount_RNA等多种结果图片。
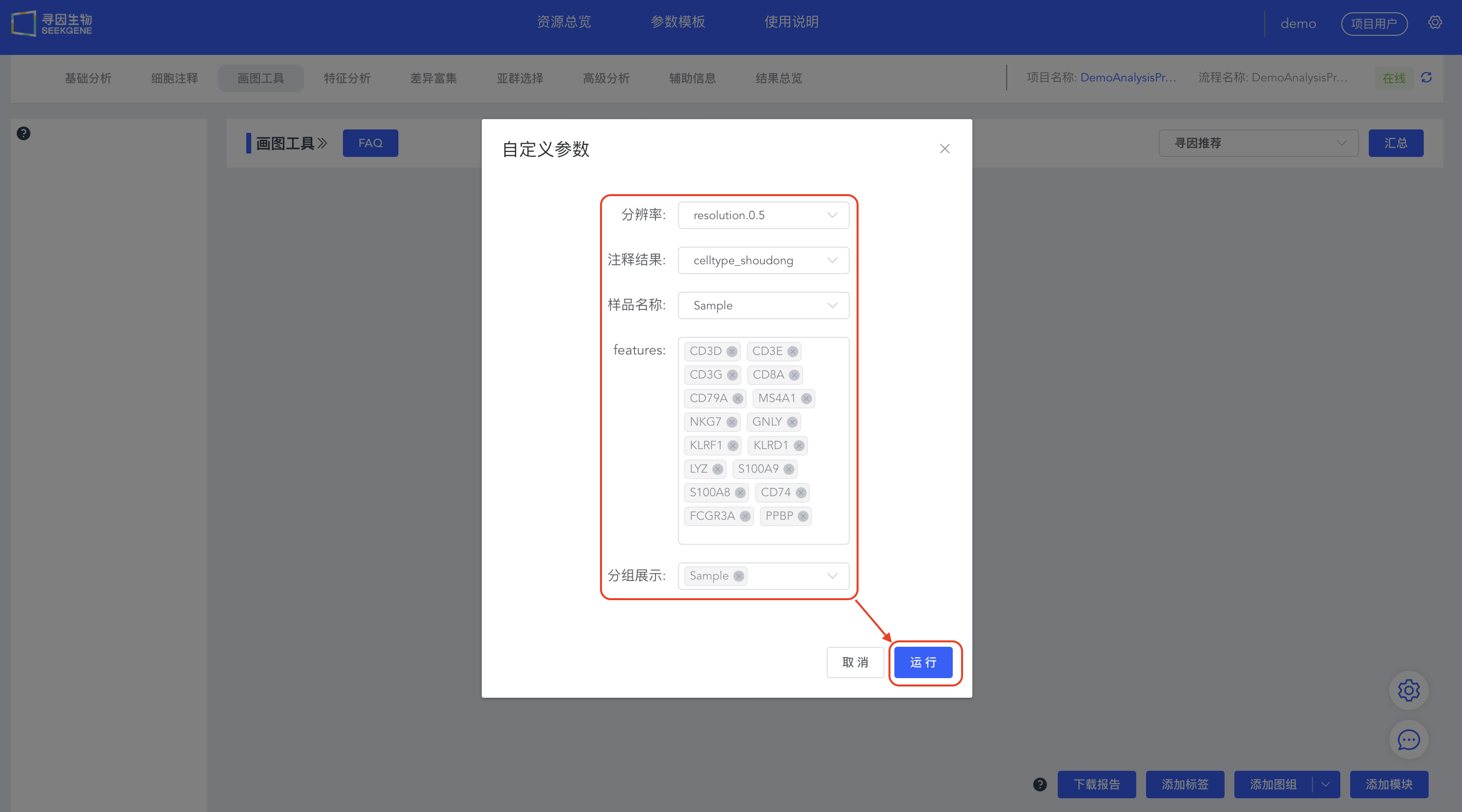
添加模块
配色方案
点击寻因推荐下拉框,即可在画图工具模块选择不同配色方案,包括npg、lancet、aaas、jco、gsea、ucscgb、igv和寻因推荐。
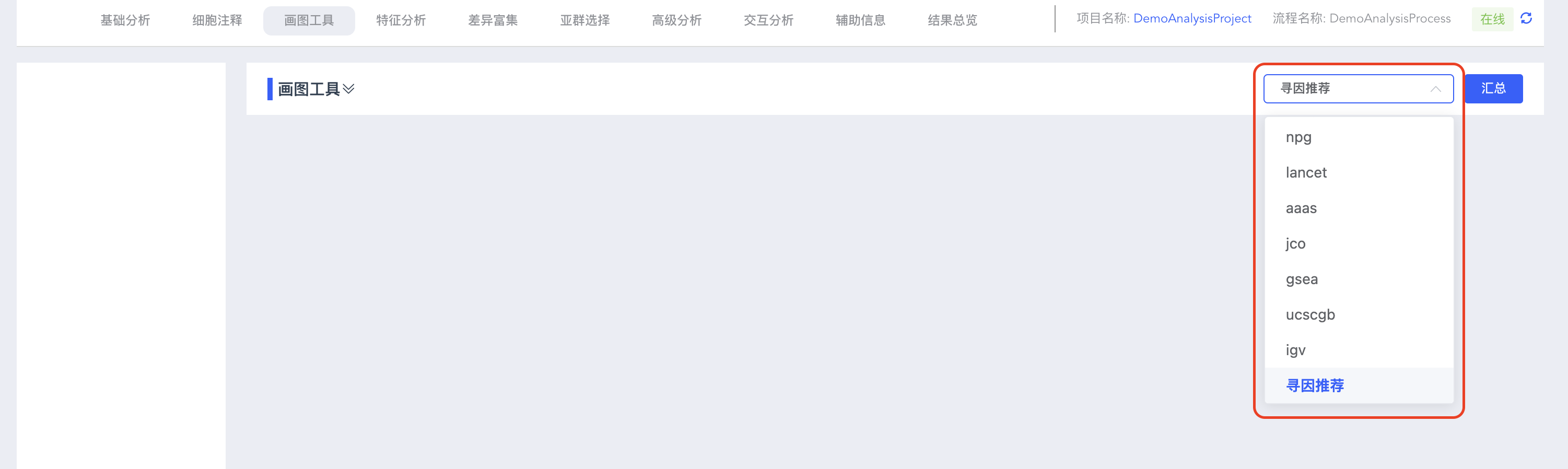
添加模块
点击【添加模块】,在“标题”处输入图表标题,在“描述”处对图表信息进行描述,选择合适的图表类型(小提琴图、降维图、FeaturePlot、DotPlot或分组统计图)进行绘图分析。
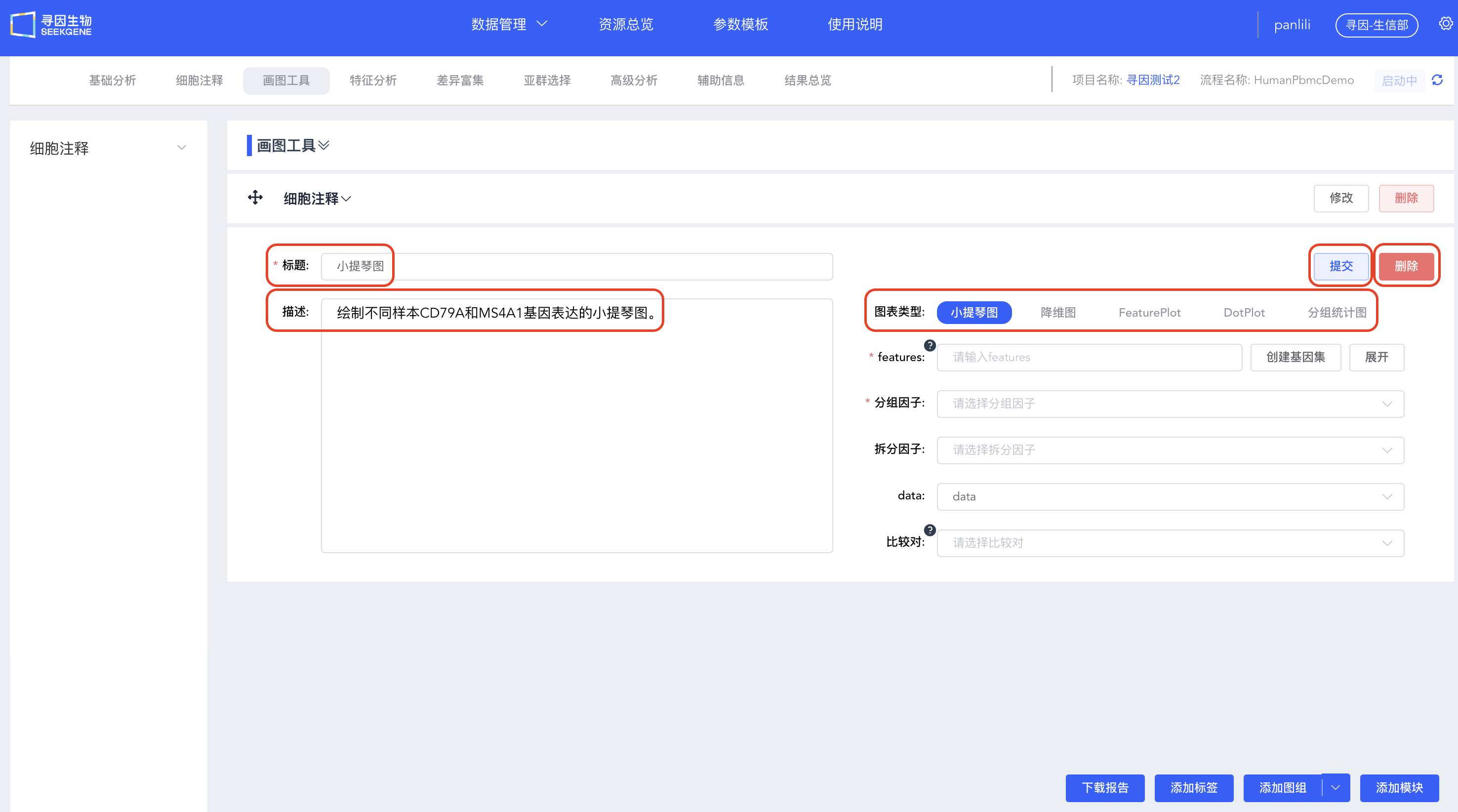
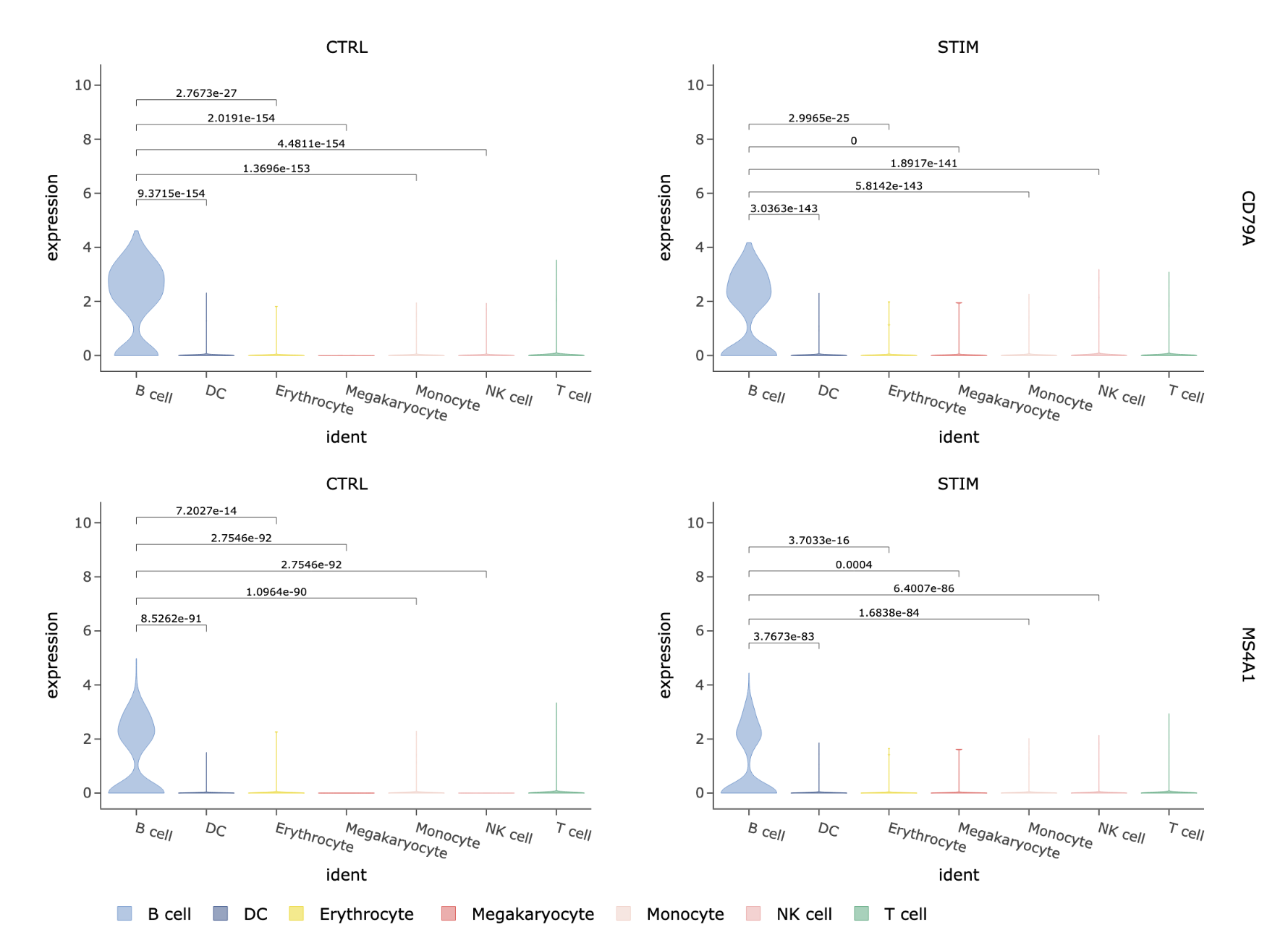
小提琴图
可按分组类型绘制基因表达量小提琴图。
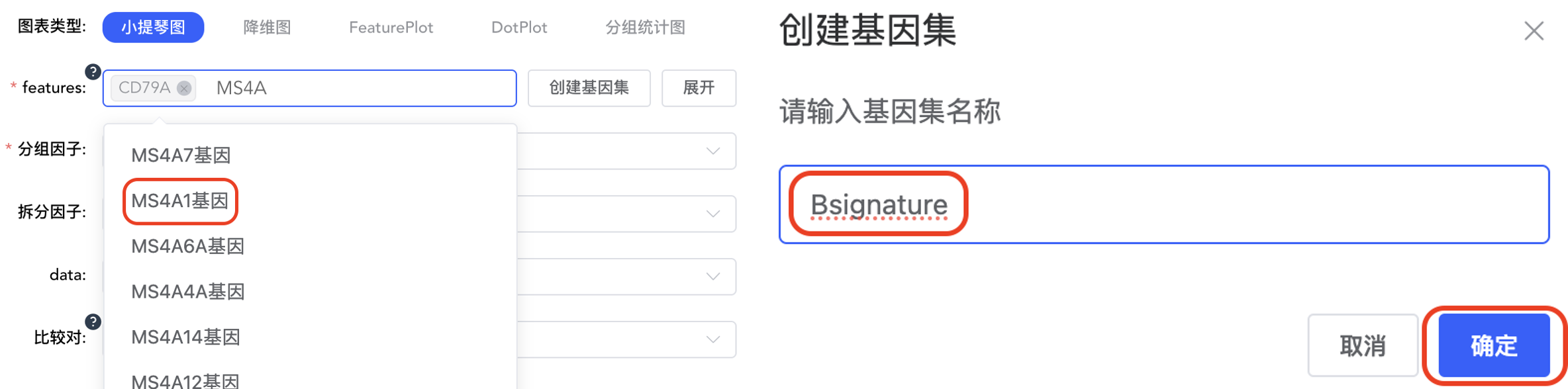
features:输入要绘制的基因,下拉框模糊匹配搜索,选择要绘制的基因,可连续输入多个基因。点击【创建基因集】,弹出输入基因集名称窗口,输入基因集名称,点击【确定】完成基因集创建。输入要绘制的基因集名称,下拉框模糊匹配搜索,选择要绘制的基因集,快速对基因集进行绘图。点击【展开】查看全部基因名称,点击【折叠】折叠基因名称。
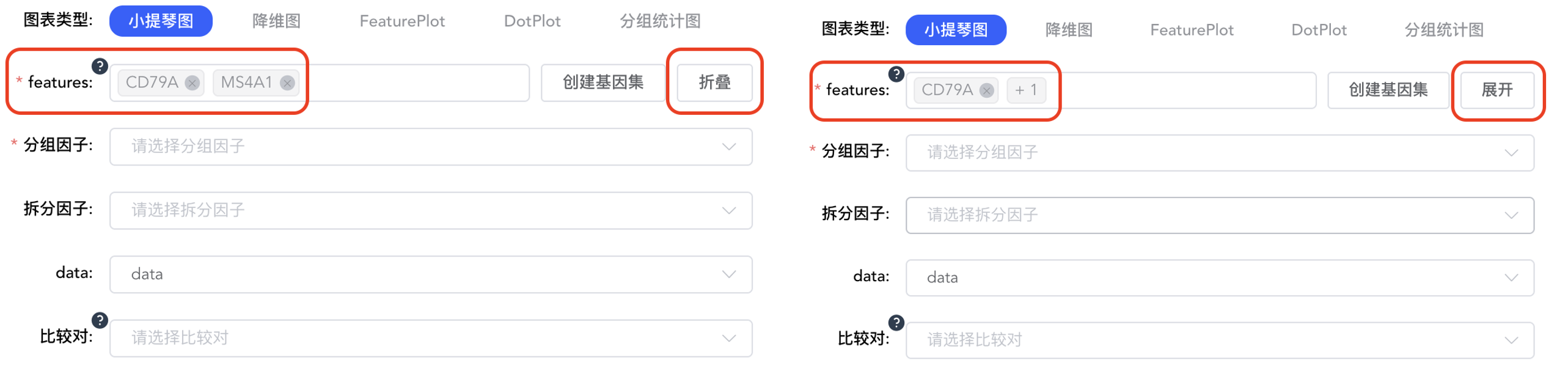
分组因子:对meta.data中的分类标签和【添加标签】中新添加的标签,选择一个来按组(颜色)对细胞进行分类,必填。可根据二级标签,只展示部分分组(颜色)。
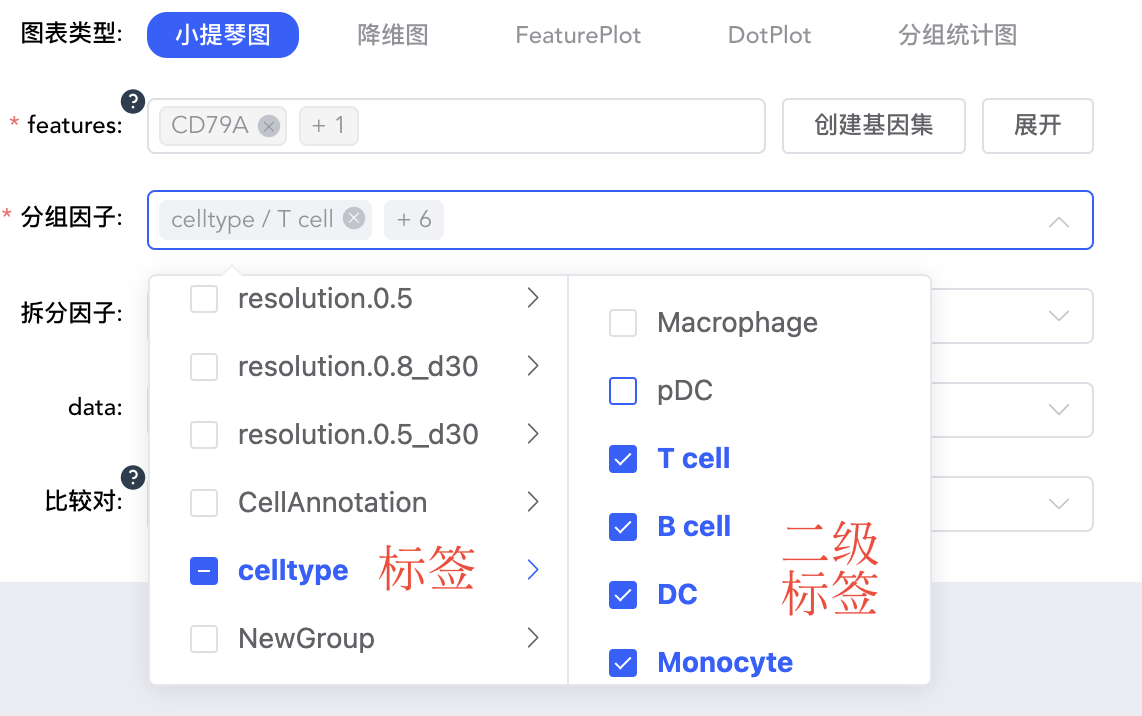
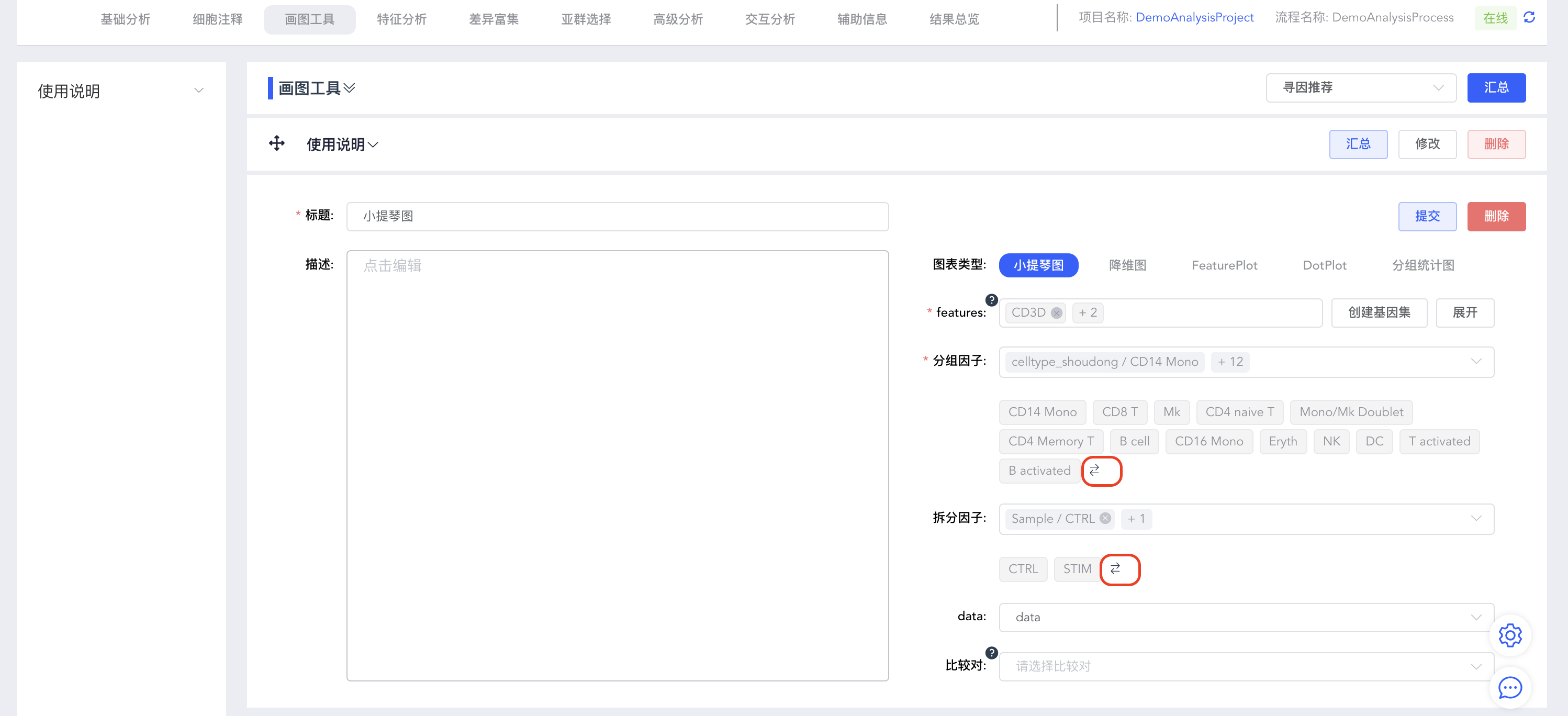
拆分因子:对meta.data中的分类标签和【添加标签】中新添加的标签,选择一个将图形拆分为不同子图,非必填。可根据二级标签,只展示部分分组。点击分组因子和拆分因子后的箭头可快速调整顺序。
data:选择使用哪个数据槽(slot)的基因表达数据进行可视化展示,默认是data。
比较对:当分组因子有多个时,可标注两两比较p值,非必填。可根据二级标签,只展示部分分组的差异比较。
选择合适的绘图参数,点击【提交】,绘制小提琴图。
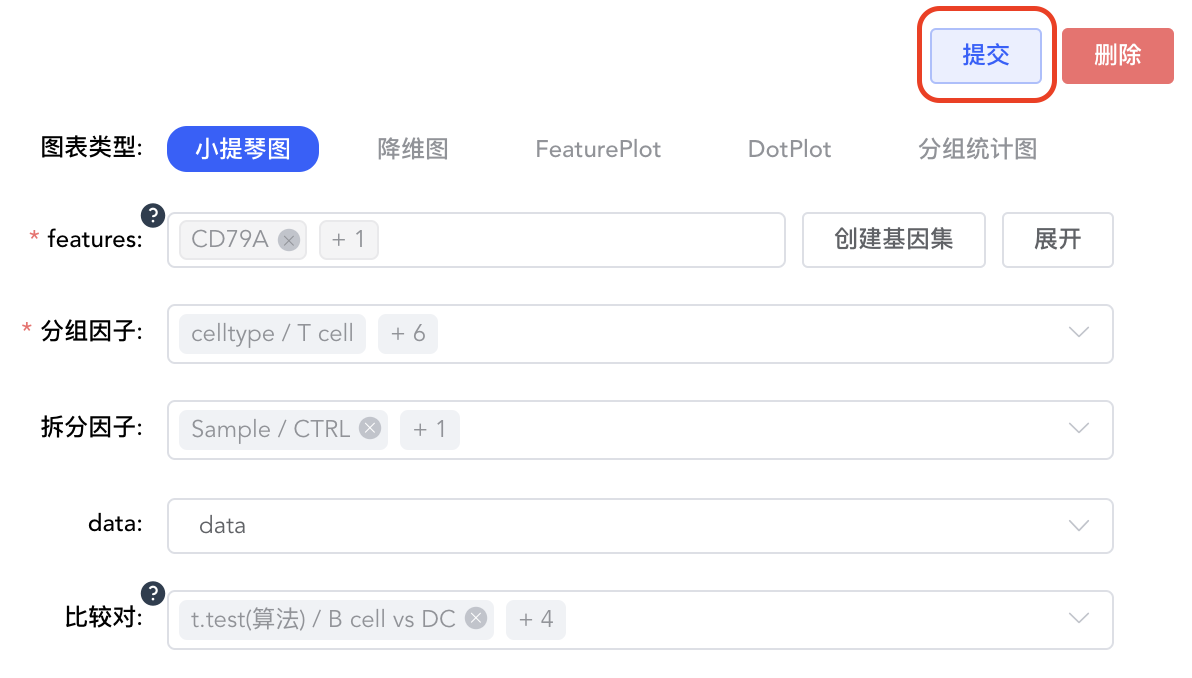
点击【删除】,删除当前小提琴图结果。
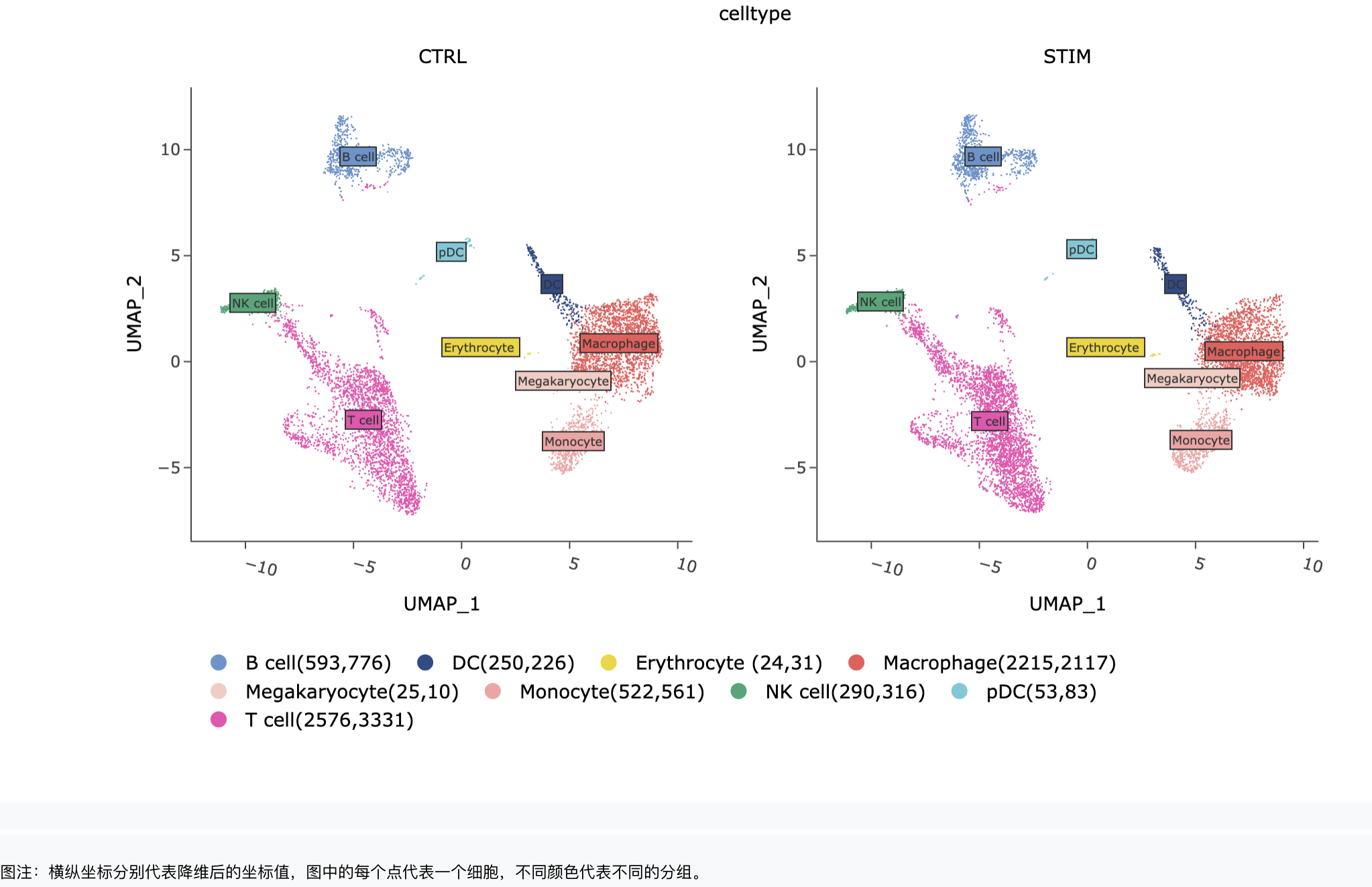
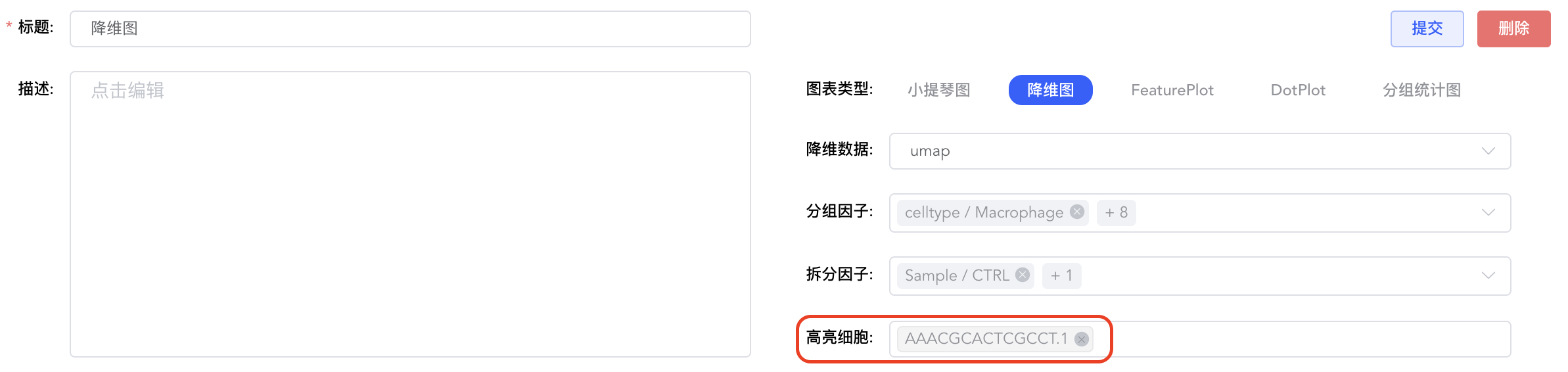
降维图
可按分组或样本拆分情况绘制降维图。
降维数据:umap方式、tsne方式、pca方式。
分组因子:对meta.data中的分类标签和【添加标签】中新添加的标签,选择一个来按组(颜色)对细胞进行分类,必填。可根据二级标签,只展示部分分组(颜色)。
拆分因子:对meta.data中的分类标签和【添加标签】中新添加的标签,选择一个将图形拆分为不同子图,非必填。可根据二级标签,只展示部分分组。
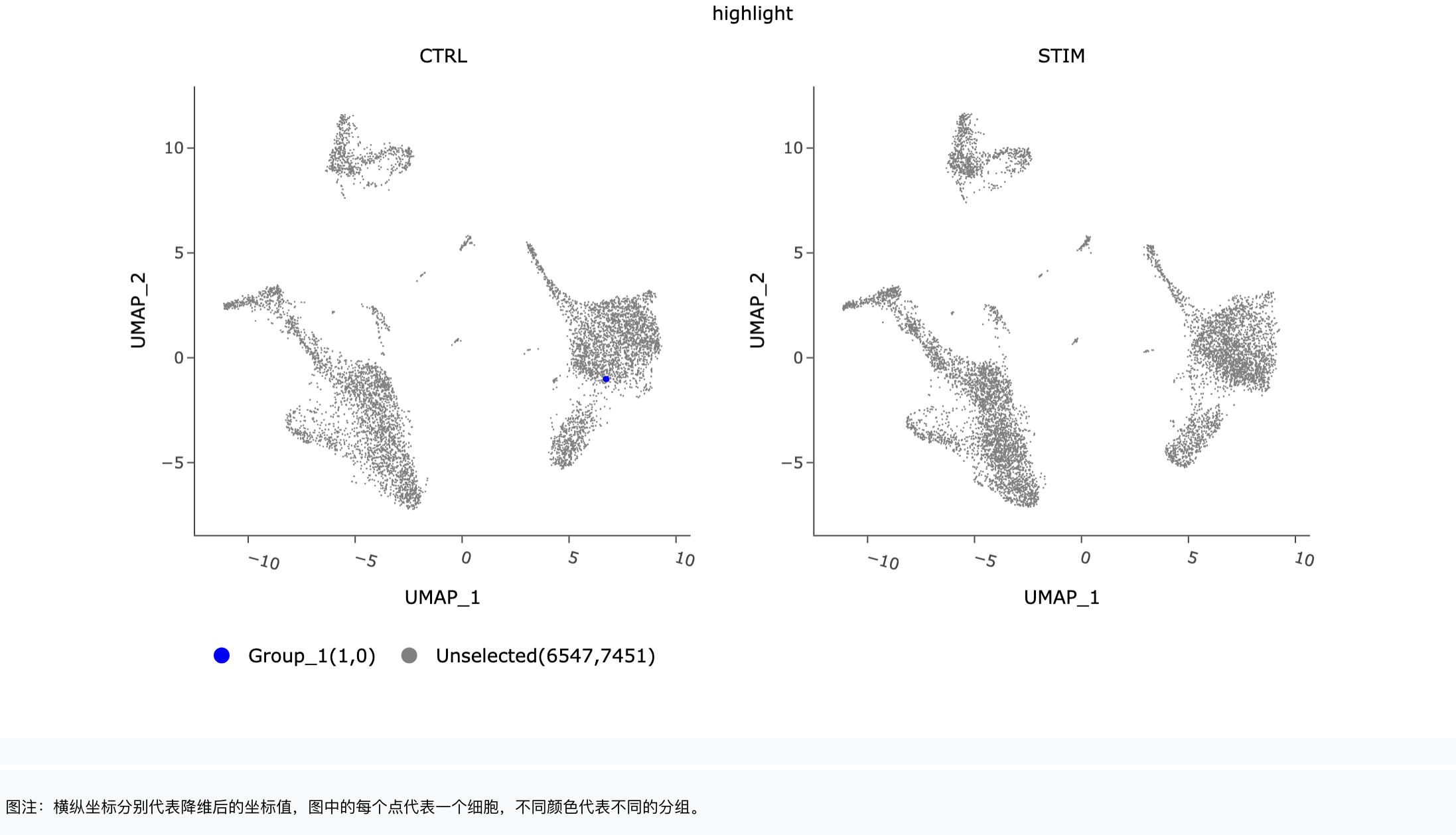
高亮细胞:输入关注细胞的Barcode,高亮显示该细胞,非必填。
常规降维图:
高亮细胞降维图:
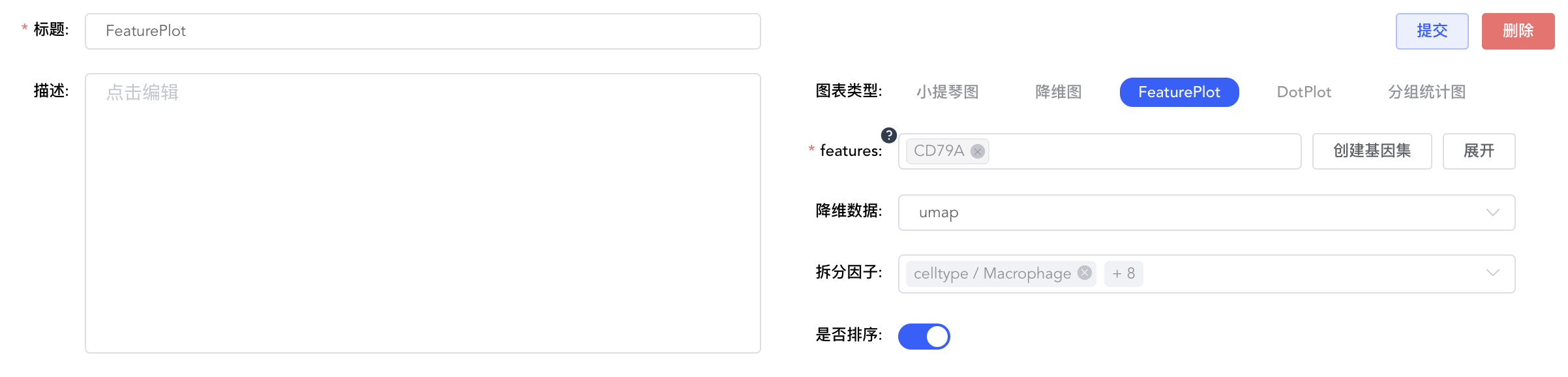
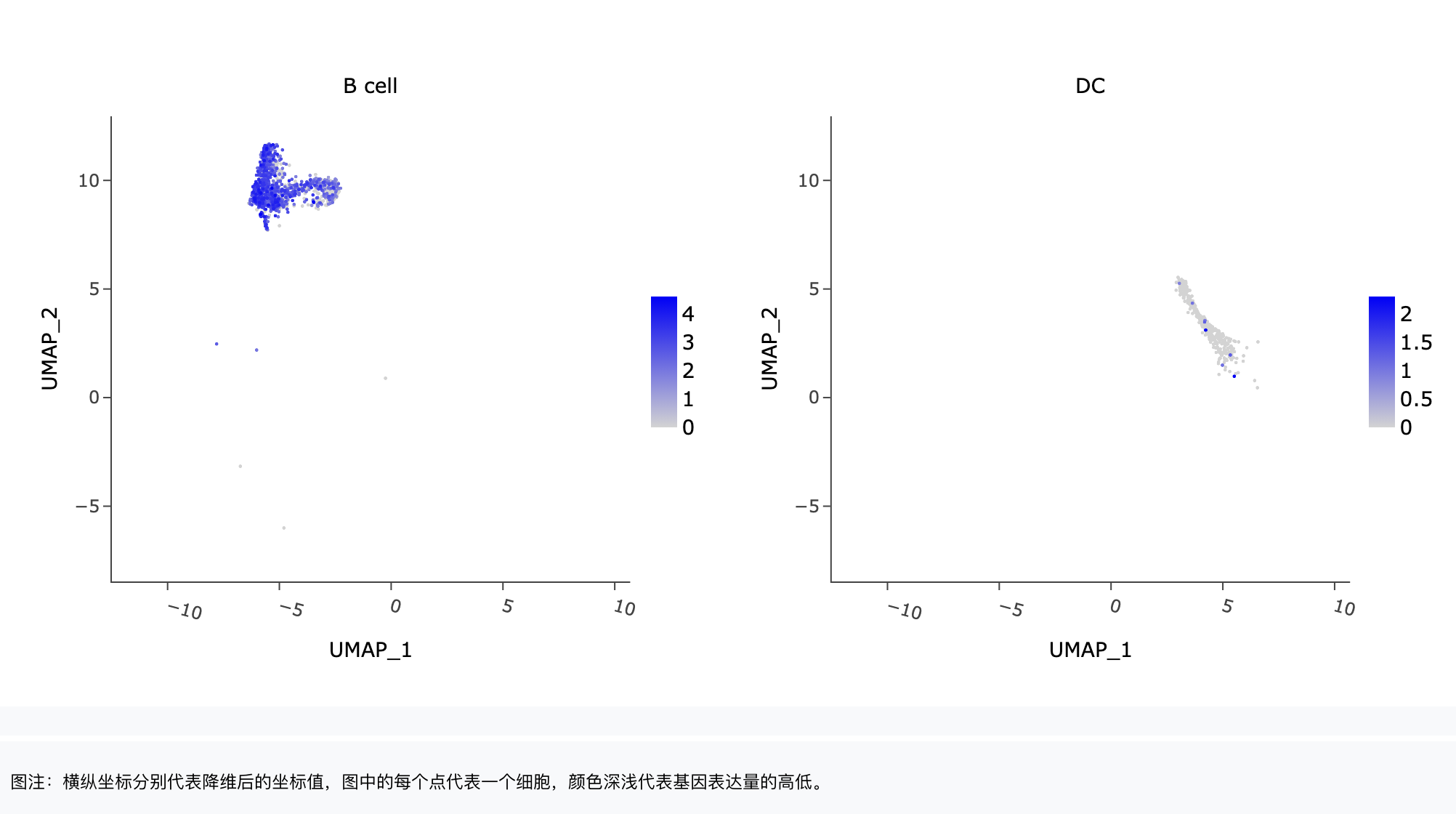
FeaturePlot
可按分组类型绘制基因表达量FeaturePlot图。
features:输入要绘制的基因,下拉框模糊匹配搜索,选择要绘制的基因,可连续输入多个基因。点击【创建基因集】,弹出输入基因集名称窗口,输入基因集名称,点击【确定】完成基因集创建。输入要绘制的基因集名称,下拉框模糊匹配搜索,选择要绘制的基因集,快速对基因集进行绘图。点击【展开】查看全部基因名称,点击【折叠】折叠基因名称。
降维数据:umap方式、tsne方式、pca方式。
拆分因子:对meta.data中的分类标签和【添加标签】中新添加的标签,选择一个将图形拆分为不同子图,非必填。可根据二级标签,只展示部分分组。
是否排序:是否按照表达量顺序绘制细胞,默认是按照表达量顺序绘制。
DotPlot
可按分组类型绘制基因表达量dotplot图。
features:输入要绘制的基因,下拉框模糊匹配搜索,选择要绘制的基因,可连续输入多个基因。点击【创建基因集】,弹出输入基因集名称窗口,输入基因集名称,点击【确定】完成基因集创建。输入要绘制的基因集名称,下拉框模糊匹配搜索,选择要绘制的基因集,快速对基因集进行绘图。点击【展开】查看全部基因名称,点击【折叠】折叠基因名称。
分组因子:对meta.data中的分类标签和【添加标签】中新添加的标签,选择一个来按组对细胞进行分类,必填。可根据二级标签,只展示部分分组。
拆分因子:选择meta.data中的分类标签或【添加标签】中新添加的标签,在一个气泡图中拆分展示,非必填。可根据二级标签,只展示部分分组。
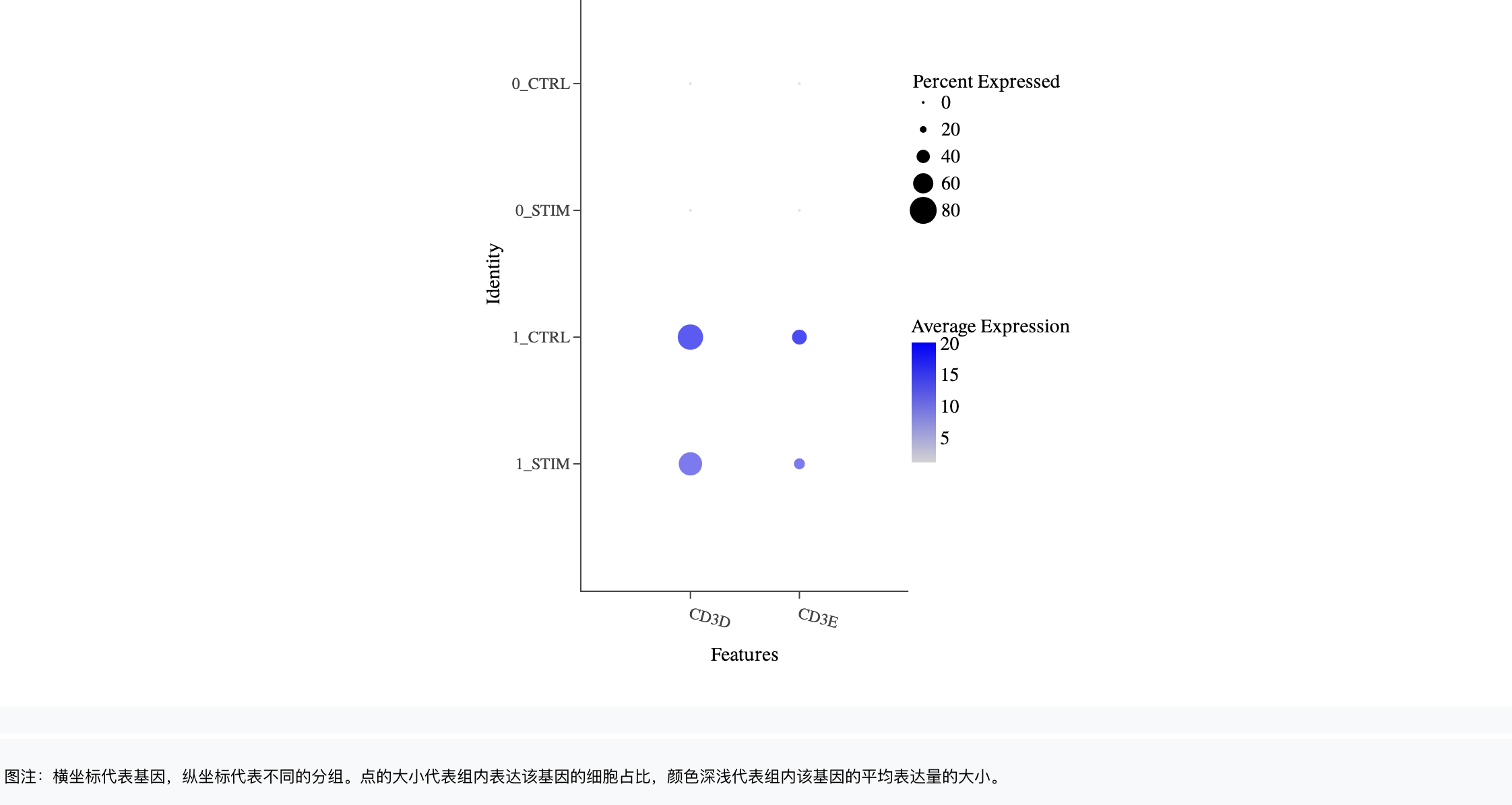
分组统计图
堆叠柱状图
分组因子:对meta.data中的分类标签和【添加标签】中新添加的标签,选择一个来按组(颜色)对细胞进行分类,必填。可根据二级标签,只展示部分分组(颜色)。
拆分因子:对meta.data中的分类标签和【添加标签】中新添加的标签,选择一个将图形拆分为不同子图,非必填。可根据二级标签,只展示部分分组。
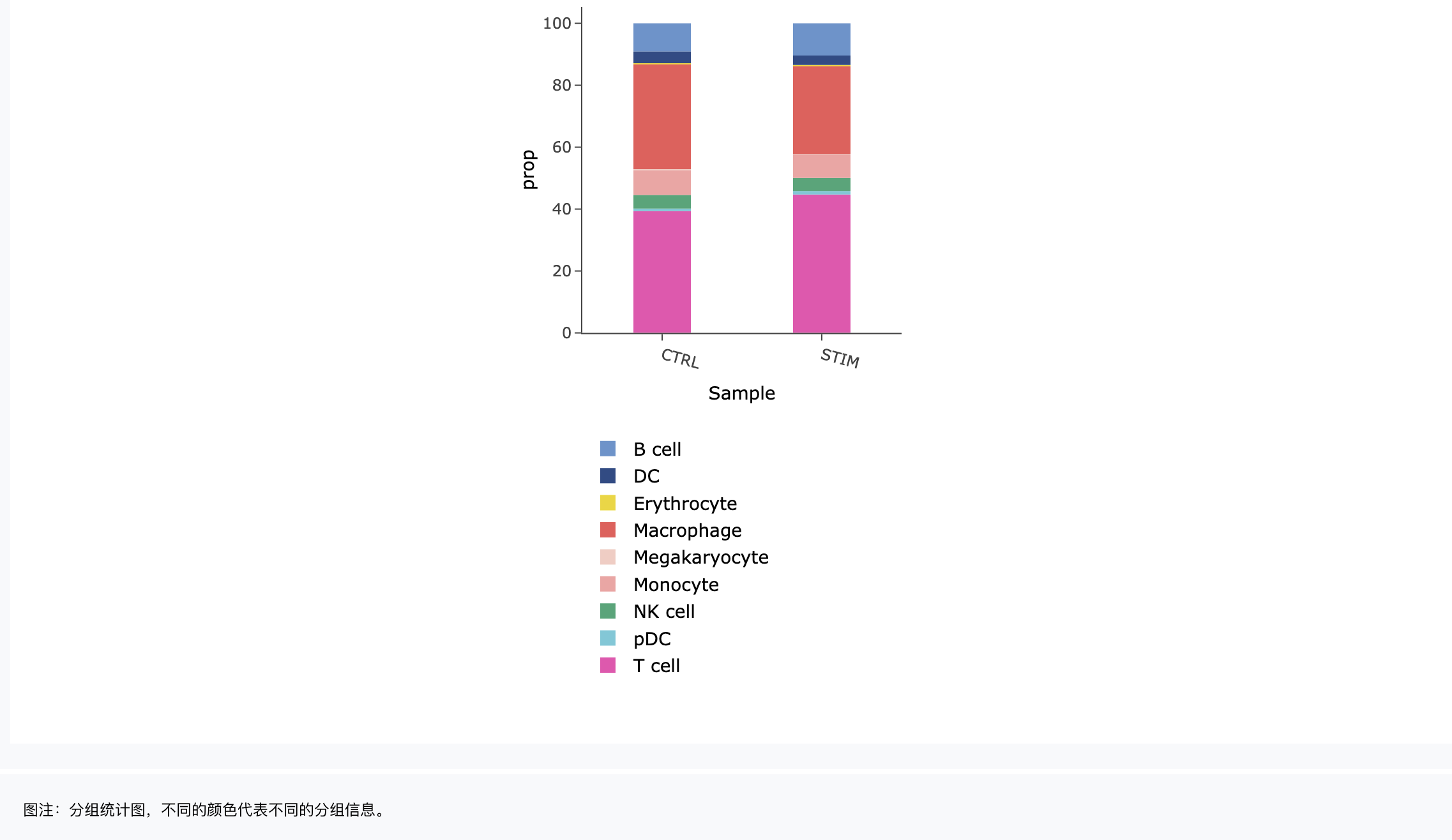
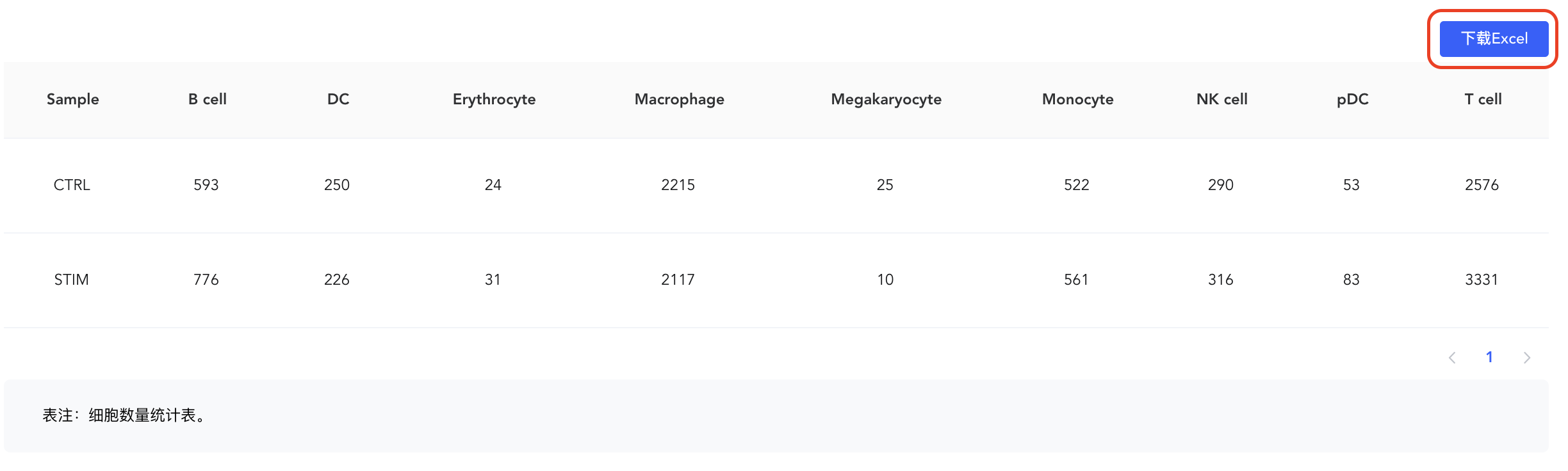
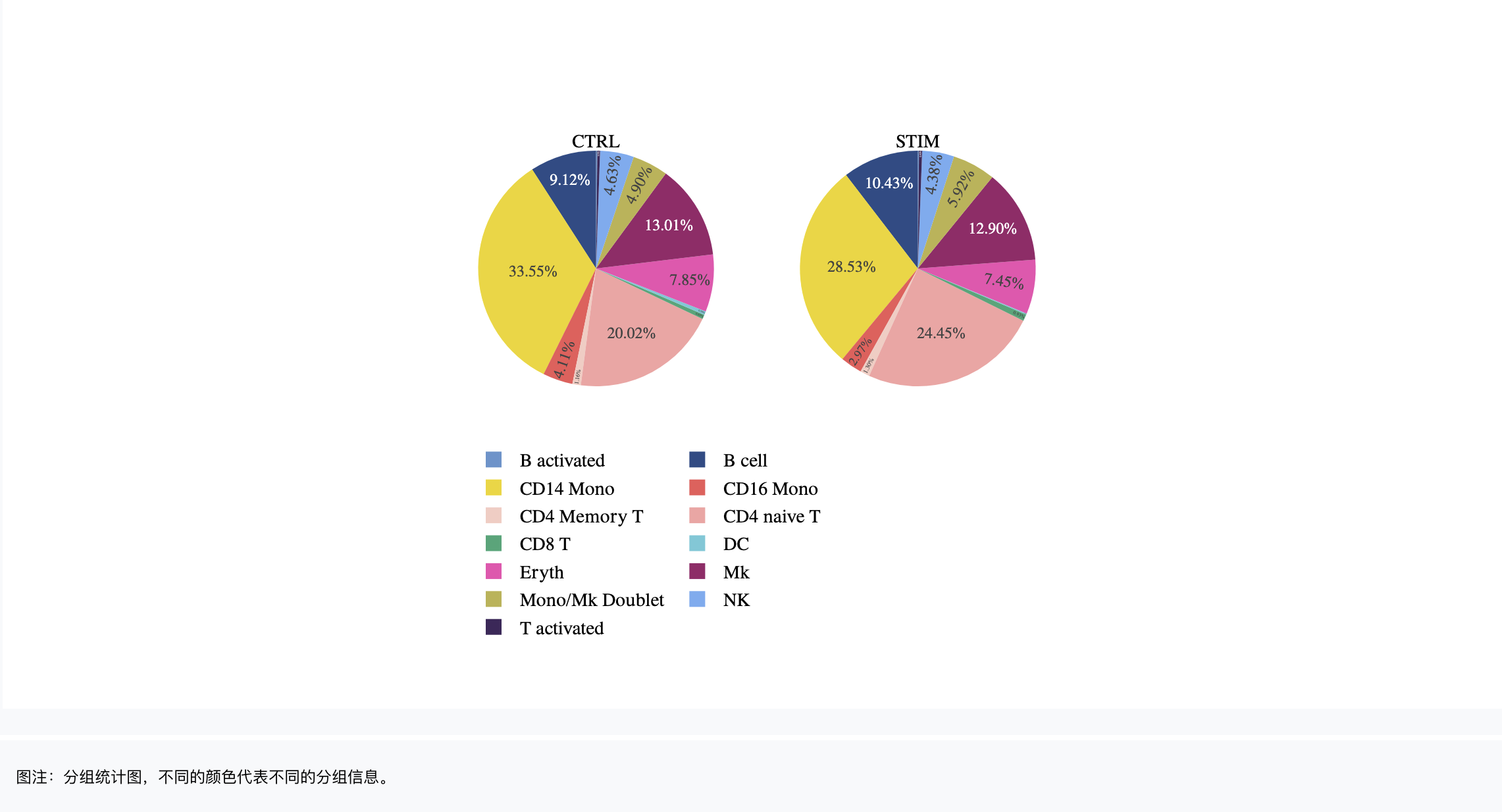
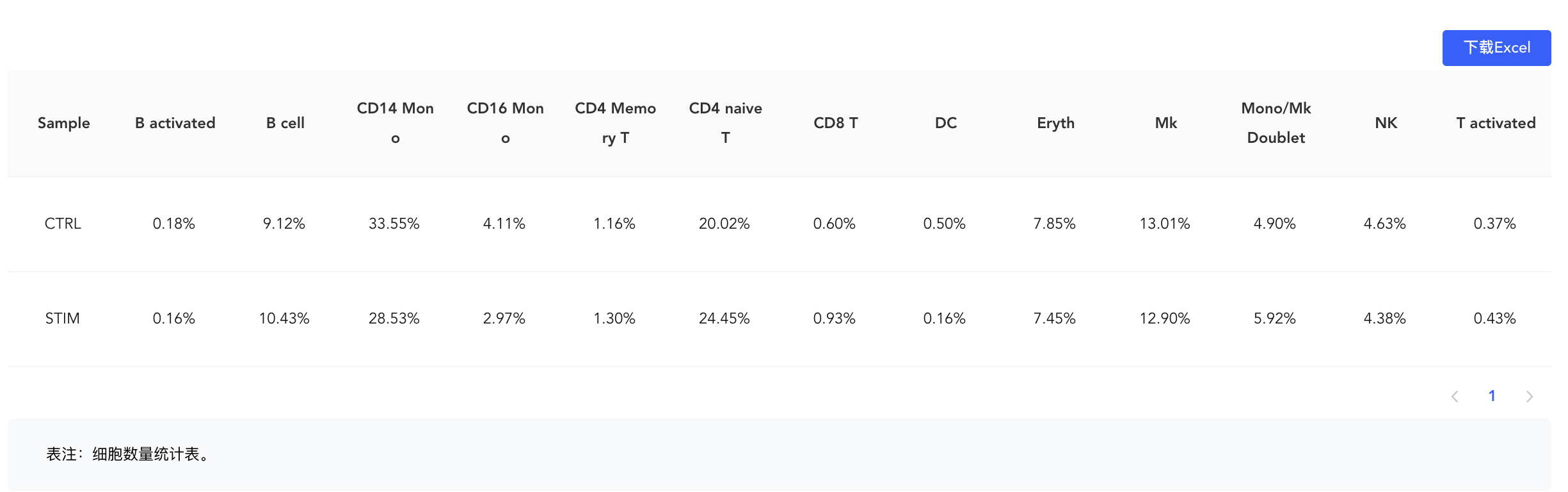
饼图
分组因子:对meta.data中的分类标签和【添加标签】中新添加的标签,选择一个来按组(颜色)对细胞进行分类,必填。可根据二级标签,只展示部分分组(颜色)。
拆分因子:对meta.data中的分类标签和【添加标签】中新添加的标签,选择一个将图形拆分为不同子图,非必填。可根据二级标签,只展示部分分组。
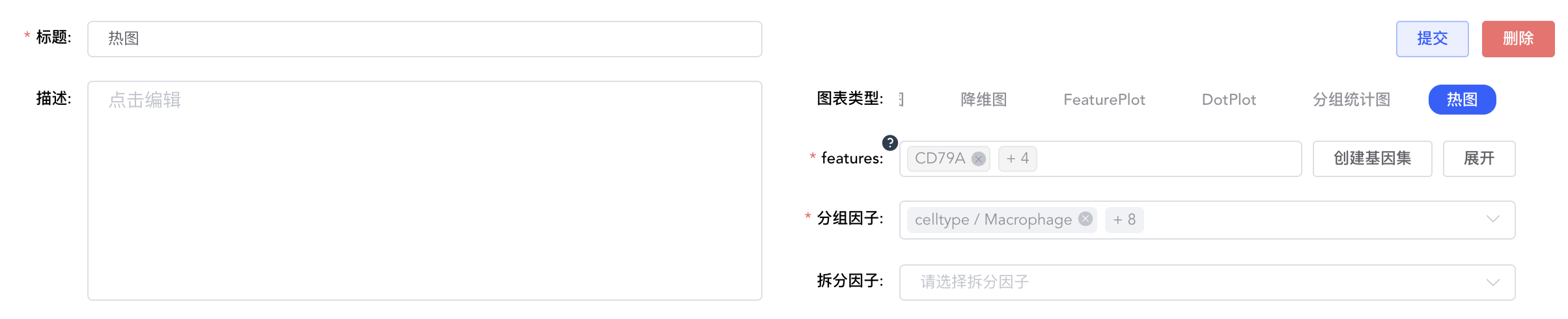
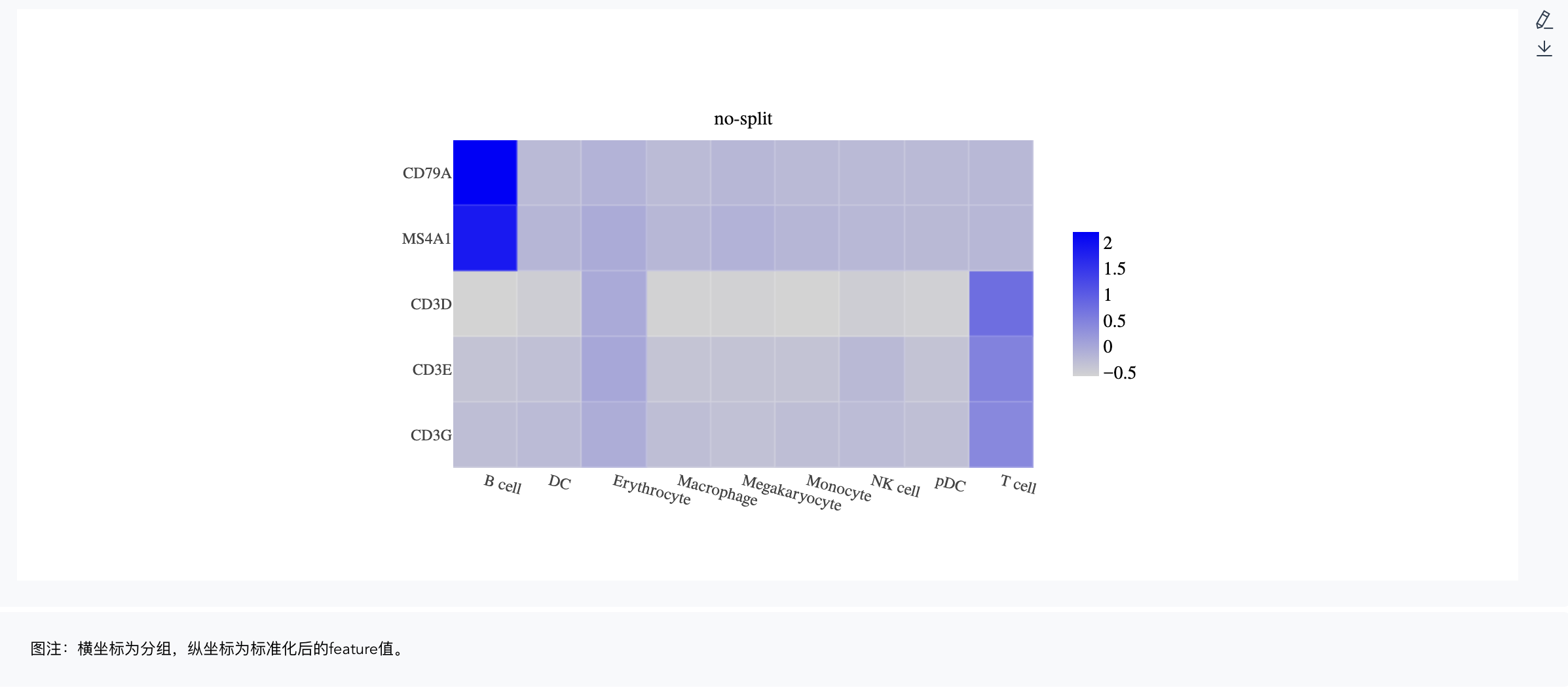
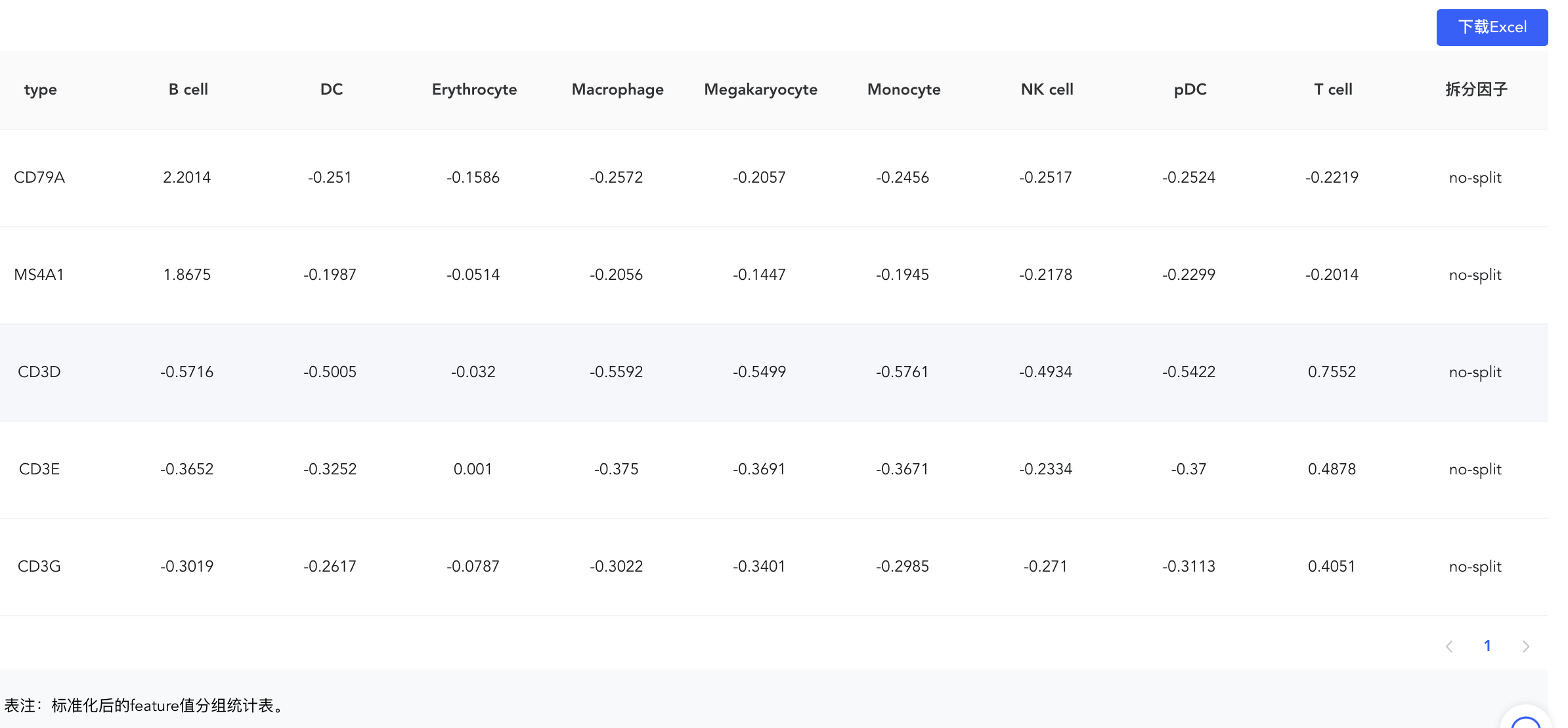
热图
features:输入要绘制的基因,下拉框模糊匹配搜索,选择要绘制的基因,可连续输入多个基因。点击【创建基因集】,弹出输入基因集名称窗口,输入基因集名称,点击【确定】完成基因集创建。输入要绘制的基因集名称,下拉框模糊匹配搜索,选择要绘制的基因集,快速对基因集进行绘图。点击【展开】查看全部基因名称,点击【折叠】折叠基因名称。
分组因子:对meta.data中的分类标签和【添加标签】中新添加的标签,选择一个来按组对细胞进行分类,必填。可根据二级标签,只展示部分分组。
拆分因子:对meta.data中的分类标签和【添加标签】中新添加的标签,选择一个将图形拆分为不同子图,非必填。可根据二级标签,只展示部分分组。
图片美化
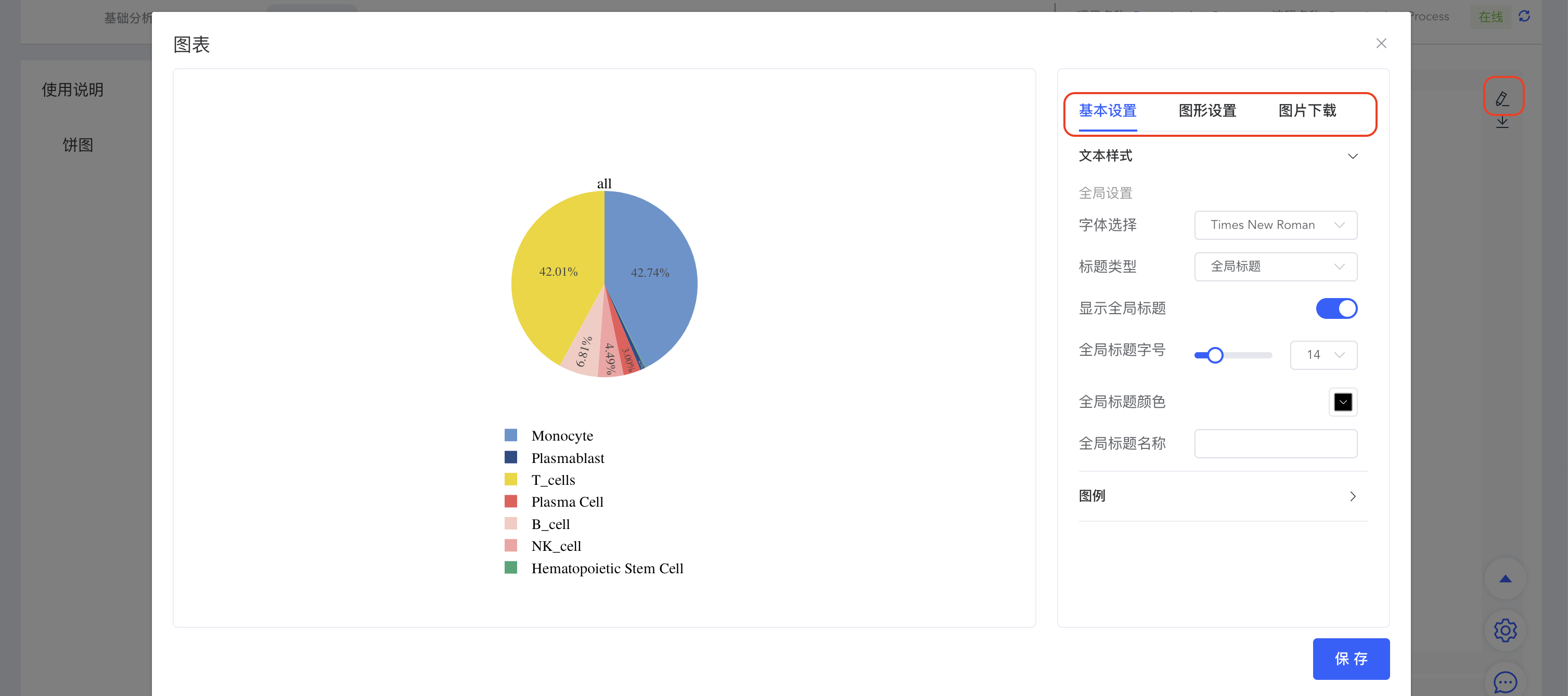
点击图片右侧的铅笔按钮,弹出图片美化界面,可以对标题、图例、图形大小及颜色进行修改,还可以下载多种格式的图片。
TIP
建议合理利用标签、分组和拆分因子,灵活组合可视化方式,提升数据解读效率。
