GSVA基因集变异分析
前言
TIP
GSVA (Gene Set Variation Analysis) 是一种功能强大的非参数、无监督的富集分析方法。它通过将基因表达谱转化为基因集(如信号通路)的活性得分,从而在单细胞或样本水平上评估生物学通路的变化,尤其适用于揭示不同细胞亚群间的功能异质性。
在单细胞转录组学研究中,我们不仅关心单个基因的表达变化,更希望从系统层面理解细胞的功能状态。传统的富集分析(如GO/KEGG)通常依赖于差异表达基因的筛选,这可能忽略那些表达变化虽不显著但协同发挥作用的基因。GSVA则弥补了这一不足,它将每个细胞视为一个独立的样本,基于所有基因的表达信息来计算每个功能基因集的富集分数,因此能够更全面、更灵敏地捕捉细胞状态的动态变化。
本篇文档旨在为单细胞研究者提供一份详尽的GSVA技术指南,内容涵盖其核心原理、在SeekSoulOnline云平台上的操作方法、结果解读、实战案例及常见问题,帮助您快速掌握并应用该工具进行深入的功能分析。
GSVA理论基础
核心原理
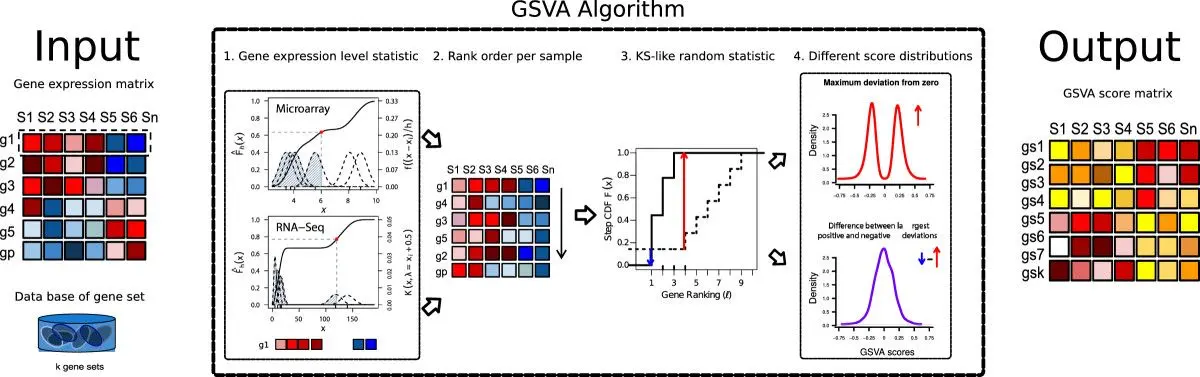
GSVA的核心思想是将一个“基因-细胞”表达矩阵,转换为一个“基因集-细胞”的GSVA得分矩阵。这个过程不依赖于预先的差异分析,而是直接对每个细胞进行打分。
其算法流程可以概括为:
- 基因表达排序:对于单个细胞,算法会根据基因的表达水平从高到低进行排序。
- 富集分数计算:对于一个给定的基因集(例如,某条KEGG通路),算法会评估该基因集内的基因在上述排序列表中的分布情况。如果基因集内的基因显著地聚集在排序列表的顶端或底端,则会得到一个较高的富集分数。这个过程类似于GSEA(Gene Set Enrichment Analysis),但GSVA是在单个样本(细胞)内完成的。
- 得分转化:通过非参数的核密度估计,将富集分数转化为最终的GSVA得分。这个得分反映了该基因集在该细胞中的相对活性。
TIP
与经典的GSEA(基于群体的富集分析)相比,GSVA最大的优势在于它是一种单样本(single-sample)富集分析方法。它为每个细胞都计算出一个通路活性得分,从而可以将功能分析提升到单细胞分辨率。这使得我们可以在UMAP降维图上直观地展示通路活性的分布,或者对不同细胞亚群的通路活性进行定量比较。
云平台操作指南
在云平台上,GSVA分析流程被设计得直观易用。您无需编写代码,只需通过参数配置界面即可完成分析。
分析前的准备
TIP
高质量的输入数据是获得可靠GSVA结果的前提。在开始分析前,请务必确认:
- 数据已完成预处理:您的单细胞数据已经过标准的质控、降维、聚类和细胞类型注释。
- 选择了合适的分组:GSVA分析旨在比较不同组别(如不同细胞类型、不同处理条件)的功能差异。请确保您的metadata中包含了用于分组的列。
参数详解
下表详细列出了云平台GSVA分析模块的主要参数及其说明。
| 界面参数 | 说明 |
|---|---|
| 任务名称 | 本次分析的任务名称,需以英文字母开头,可包含字母、数字、下划线和中文。 |
| 分组因子 | 选择用于定义细胞群体的标签,如celltype。 |
| 细胞类型 | 多选,选择要纳入分析的具体细胞类型或聚类。 |
| 物种 | 支持人和小鼠等多种物种。 |
| 基因集类型 | 多选,平台预置的MSigDB基因集。 |
| 自定义基因集 | 上传自定义的基因集文件(.gmt格式)。上传时,<物种>需选择“user-defined”。 |
| 拆分样本 | 绘图时的拆分标签,通常选择样本标签,如Sample。 |
| 样本类型 | 多选,选择要分析的样本。 |
| 拆分分组 | 绘图时的拆分标签,通常选择分组标签,如Group。 |
| 分组类型 | 多选,选择要分析的分组。 |
| 分组比较 | 多选,选择要进行差异通路分析的组别。 |
| qvalue | 差异通路筛选的q值(FDR)阈值。 |
| Downsample | 是否对大数据集进行随机下采样以加速分析。 |
| Downsample_num | 若开启下采样,每个细胞亚群保留的细胞数目。 |
| 备注 | 自定义备注信息。 |
| 基于历史结果分析 | 选择“基因集绘图”,可基于已完成的分析任务重新筛选基因集进行绘图。 |
| 历史任务名称 | 选择一个已成功的GSVA任务。 |
| 筛选基因集 | 上传一个只包含希望展示的基因集名称的列表文件。 |
重要注意事项
TIP
- 自定义基因集格式:上传的基因集文件(.gmt)必须使用制表符(Tab)作为分隔符,不能用空格。
- 热图限制:如果上传或筛选后的基因集只包含一个通路,流程将无法绘制热图。
操作流程
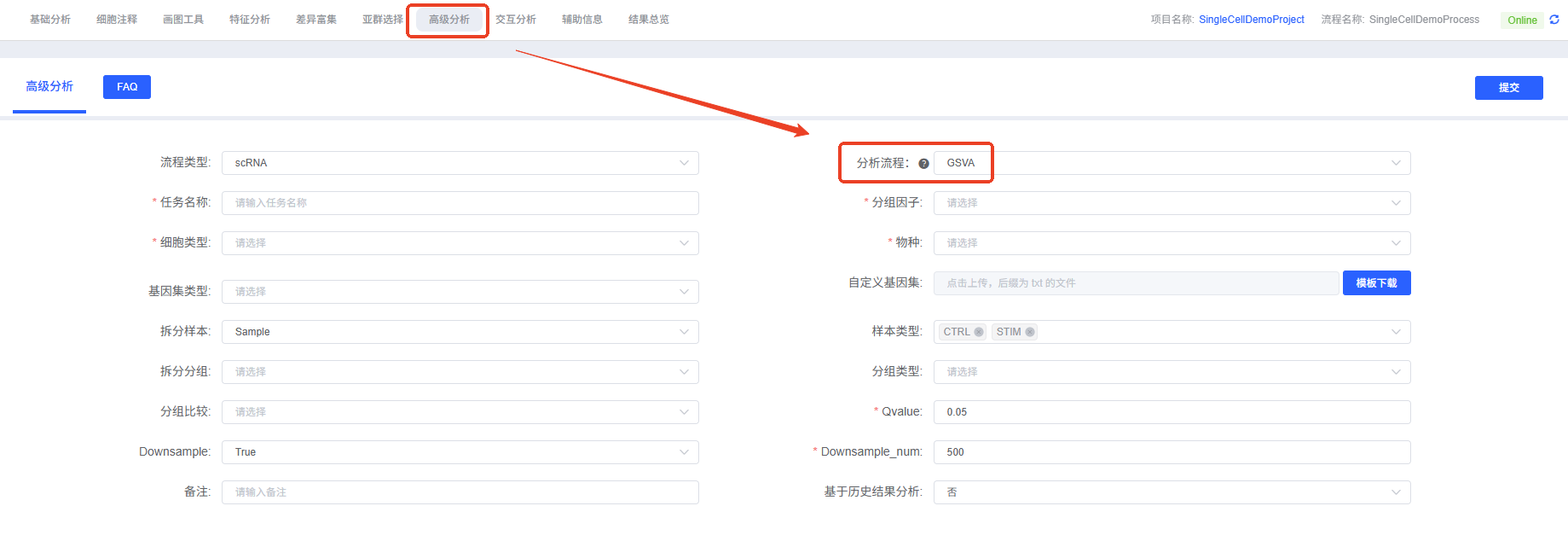
- 进入分析模块:在云平台导航至“高级分析”模块,选择“GSVA”。
- 创建新任务:为您的分析任务命名,并选择要分析的样本或项目。
- 配置参数:根据上述指南,选择分组因子、细胞类型、物种、基因集类型等参数。
- 提交任务:确认参数无误后,点击“提交”按钮,等待分析完成。
- 下载与查看:分析结束后,在任务列表中下载并查看生成的分析报告和结果文件。
结果解读
GSVA的分析报告提供了从多个维度审视通路活性的图表和文件。
基因集在不同细胞群间的GSVA得分
这是最核心的结果之一,通过热图展示不同细胞群在各个基因集上的平均GSVA得分,可以快速识别出特定通路在哪些细胞群中具有特异性的高活性或低活性。
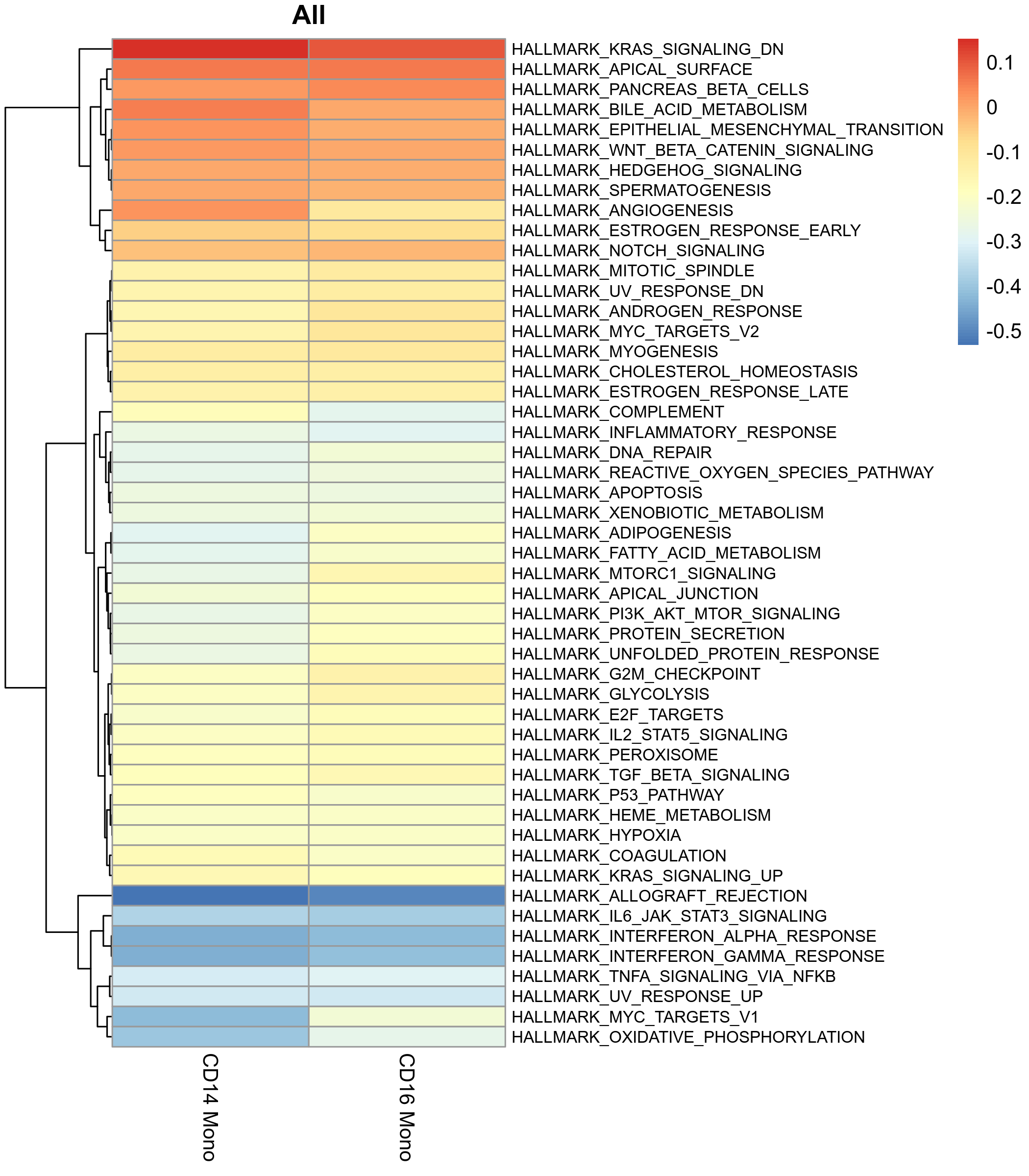
- 图表解读:
- 横坐标:细胞分组(如不同的细胞类型)。
- 纵坐标:基因集(通路)。
- 颜色:红色代表高GSVA得分(通路活性高),蓝色代表低GSVA得分(通路活性低)。
- 分析要点:
- 寻找在某一细胞群中呈现“亮红色”条带的通路,这些是该细胞群的特征性功能。
- 比较不同细胞群在同一通路上的得分差异,揭示功能异质性。
基因集在单个细胞中的GSVA得分
为了更细致地观察通路活性在细胞间的异质性,报告也提供了展示单个细胞GSVA得分的热图。
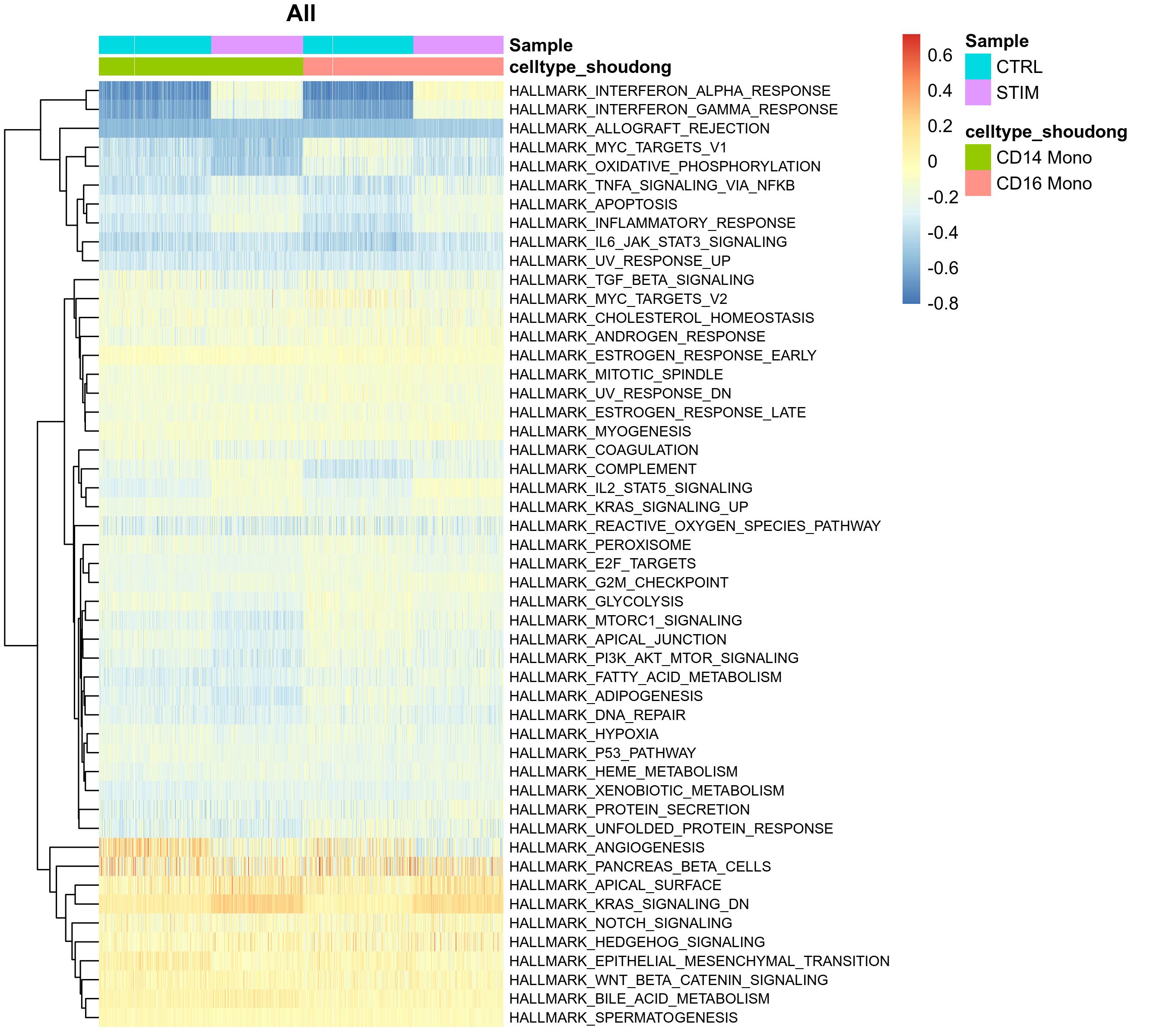
- 图表解读:
- 列:代表单个细胞,通常会按照细胞类型或分组进行排列。
- 行:代表基因集(通路)。
- 分析要点:
- 观察在同一细胞群内部,通路活性是否存在不均一性。
- 结合UMAP图,可以将高/低GSVA得分的细胞群对应到降维空间中的特定区域。
差异富集通路分析
如果设置了“分组比较”,平台会利用limma包对两组间的GSVA得分进行差异检验,并通过发散条形图(Diverging Bar Plot)等形式展示显著差异的通路。
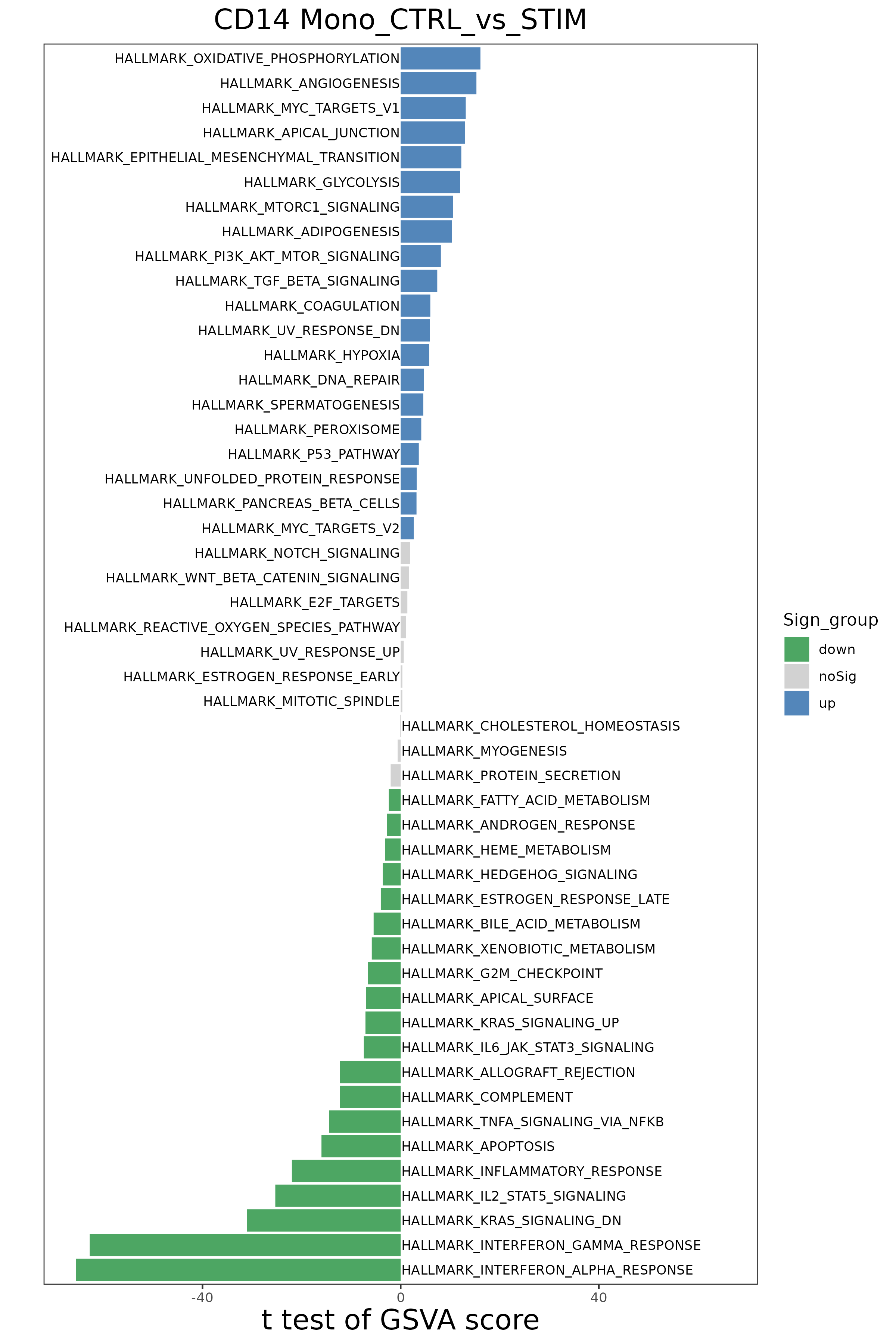
- 图表解读:
- 横坐标:t-value,反映了通路在两组间差异的强度和方向。t值为正,代表通路在比较组中活性上调;t值为负,则代表下调。
- 纵坐标:基因集(通路)名称。
- 颜色:通常,红色或蓝色等亮色代表差异具有统计学显著性(如q-value < 0.05),灰色代表不显著。
- 分析要点:
- 快速识别在不同条件下活性发生显著改变的关键生物学通路。例如,图中t值最大和最小的通路,分别是两组间差异最显著的上调和下调通路。
结果文件列表
| 文件名/目录 | 内容说明 |
|---|---|
*gsva_scores.csv | 核心数据:包含按细胞群或样本计算的GSVA得分。 |
*.png/pdf | 各种GSVA得分热图。 |
compare_Ttest/ | 差异通路:差异富集通路分析的统计结果及可视化图表。 |
split_cluster/ | 拆分样本:拆分样本后的GSVA分析结果。 |
应用案例
案例一:利用Hallmark基因集定义肿瘤亚型功能特征
- 文献:Wu SZ, et al. Nat Genet. 2021.
- 背景:研究者希望根据拷贝数变异(CNV)对乳腺癌中的恶性上皮细胞进行亚型划分,并探究不同亚型的功能特征。
- 分析策略:将细胞分为CNV-High, CNV-Low, Not detected, Control四组后,利用MSigDB的Hallmark基因集对它们进行GSVA打分。
- 核心发现:GSVA热图清晰地显示,CNV-High的细胞在“E2F信号”、“G2M检查点”和“MYC靶基因”等与细胞增殖和周期相关的通路上得分显著更高,揭示了高CNV水平与恶性增殖之间的功能关联。

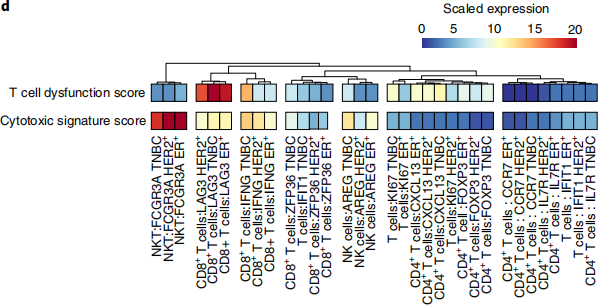
案例二:通过自定义基因集评估T细胞功能状态
- 文献:Kim, Ik Sun et al. Nature cell biology. 2019.
- 背景:在肿瘤免疫微环境中,T细胞的功能状态(如细胞毒性、功能障碍)至关重要。研究者希望定量评估不同T细胞亚群的功能。
- 分析策略:研究者首先根据经典Marker基因将T/NK细胞划分为多个亚群。随后,他们构建了两个自定义的基因集:“T细胞功能障碍评分(T cell dysfunction score)”和“细胞毒性特征评分(Cytotoxic signature score)”,并用GSVA计算每个亚群的得分。
- 核心发现:结果显示,耗竭性T细胞(Exhausted T cells)在“功能障碍评分”上得分最高,而在“细胞毒性评分”上得分较低,这与已知的生物学知识高度吻合,并为后续的免疫治疗研究提供了精准的评估指标。
常见问题解答 (FAQ)
Q1: GSVA和GSEA、GO/KEGG富集分析有什么区别?
A: 它们是三个层面的分析工具:
- GO/KEGG富集分析:基于差异基因列表,回答“这些差异基因主要富集在哪些功能/通路中?”的问题。它依赖于差异基因的阈值筛选。
- GSEA:基于所有基因在两个组间的表达变化排序,回答“某个基因集在A组是否比B组更富集?”的问题。它是一个组对组的比较。
- GSVA:同样基于所有基因,但它为每个样本/细胞都计算一个基因集得分,回答“这个细胞/样本中的某个通路活性有多高?”的问题。它将功能分析提升到了单样本/单细胞的分辨率。
Q2: 如何选择合适的基因集(.gmt文件)?
A: 基因集的选择取决于您的生物学问题:
- 探索性分析:推荐使用MSigDB的Hallmark (h.all) 基因集。它包含了50个定义明确、信息冗余度低的生物学状态或过程,是快速了解宏观功能变化的最佳选择。
- 代谢研究:可选择KEGG或Reactome通路基因集,它们包含了详尽的代谢通路信息。
- 免疫研究:可选择GO (Gene Ontology) 的BP (Biological Process) 部分,或专门的免疫基因集数据库(如ImmPort)。
- 验证性分析:如果您有特定的研究目标(如评估“铁死亡”、“上皮间质转换”),可以构建自定义基因集,将相关文献报道的核心基因列表制作成.gmt文件进行分析。
Q3: GSVA得分是正数好还是负数好?
A: GSVA得分是一个相对值,其绝对大小本身没有明确的生物学阈值。它的意义在于比较。例如,细胞群A在“凋亡”通路上的GSVA得分高于细胞群B,这说明相比于B,A中的凋亡通路可能更活跃。因此,分析的重点应放在比较不同组别、不同细胞间的得分差异,而不是纠结于得分的正负或绝对值。
参考文献
- Hänzelmann, S., Castelo, R. & Guinney, J. GSVA: gene set variation analysis for microarray and RNA-Seq data. BMC Bioinformatics 14, 7 (2013).
- Kim, Ik Sun et al. “Immuno-subtyping of breast cancer reveals distinct myeloid cell profiles and immunotherapy resistance mechanisms.” Nature cell biology vol. 21,9 (2019): 1113-1126.
- Chen Z, et al. Single-cell RNA sequencing highlights the role of inflammatory cancer-associated fibroblasts in bladder urothelial carcinoma. Nat Commun. 2020 Oct 8;11(1):5077.
- Wu SZ, et al. A single-cell and spatially resolved atlas of human breast cancers. Nat Genet. 2021 Sep;53(9):1334-1347.
- Lambrechts, et al. Phenotype molding of stromal cells in the lung tumor microenvironment. Nat Med 24, 1277–1289 (2018).
