CopyKAT拷贝数变异分析文档
前言
TIP
CopyKAT (Copynumber Karyotyping of Tumors) 是一款创新的单细胞CNV分析工具,其最大特点是能够自动、无监督地从单细胞转录组数据中区分肿瘤细胞(非整倍体)和正常细胞(二倍体),并进一步解析肿瘤内部的亚克隆结构。
与需要手动指定正常参考细胞的工具不同,CopyKAT通过集成的贝叶斯方法,自动在输入的细胞群体中识别出一组基因组最稳定的细胞作为内参。这一特性使其在以下场景中尤为强大:
- 缺乏明确的正常对照:当样本中没有可以确信为“正常”的细胞类型时。
- 探索未知肿瘤成分:在复杂的肿瘤微环境中,自动识别潜在的恶性细胞群。
- 简化分析流程:无需预先注释细胞类型,即可进行初步的肿瘤/正常细胞划分。
本篇文档将详细介绍CopyKAT的理论基础、云平台操作方法、结果解读及相关应用案例。
CopyKAT理论基础
核心原理
CopyKAT的核心假设是:在大多数肿瘤组织中,存在着基因组稳定的二倍体细胞(如免疫细胞、基质细胞)和基因组不稳定的非整倍体肿瘤细胞。这两类细胞的基因表达谱在全基因组尺度上存在系统性差异。
它通过一个多步骤的算法来识别这种差异:
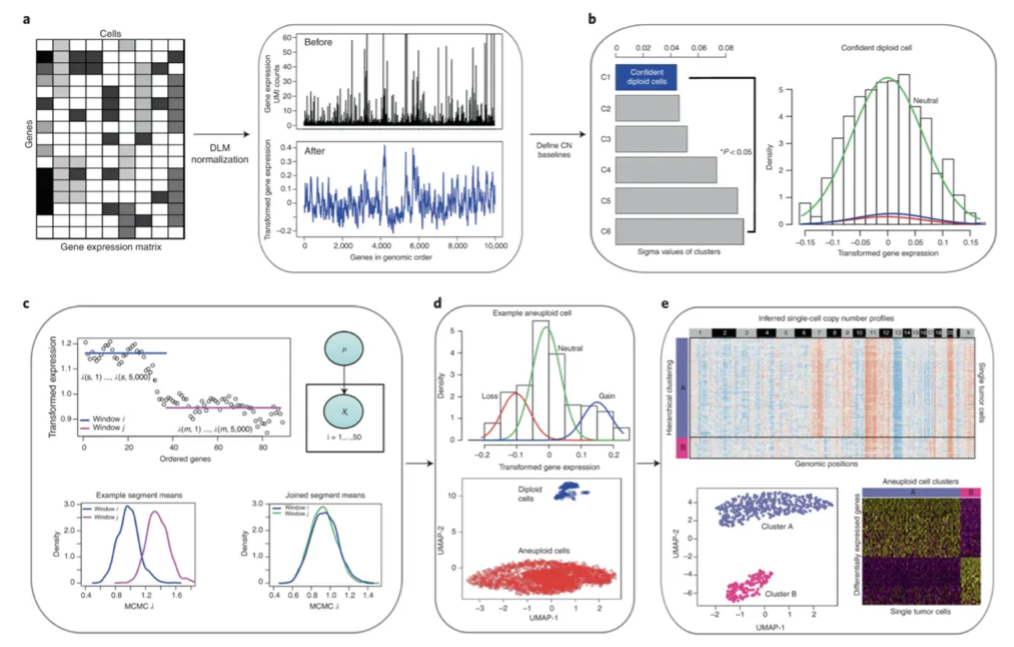
分析流程概览
- 数据标准化:对输入的基因表达矩阵进行标准化,稳定技术性方差。
- 无监督寻找正常细胞:通过高斯混合模型(Gaussian Mixture Model)分析所有细胞的基因表达分布,自动识别出一组最可能代表二倍体正常细胞的群体作为内参。
- 计算相对表达:以这组内参细胞为基准,计算其他每个细胞的基因相对表达水平。
- CNV片段识别:利用马尔可夫链蒙特卡洛(MCMC)方法,沿着染色体对基因表达信号进行分割,识别出CNV的断点(breakpoints),从而定义出具有一致拷贝数状态的染色体片段。
- 细胞分类:基于每个细胞的CNV谱,利用贝叶斯方法计算其属于“非整倍体”(aneuploid)或“二倍体”(diploid)的后验概率,从而对细胞进行分类。
- 亚克隆分析:在被分类为“非整倍体”的肿瘤细胞内部,通过对它们的CNV模式进行层次聚类,进一步划分出具有不同CNV特征的肿瘤亚克隆。
云平台操作指南
在云平台上,CopyKAT的分析流程非常简洁。
参数详解
| 界面参数 | 说明 |
|---|---|
| 任务名称 | 本次分析的任务名称,需以英文字母开头,可包含英文字母、数字、下划线和中文。 |
| 分组因子 | 选择要分析的细胞类型或聚类对应的标签(如CellAnnotation),与下方的“细胞类型”配合使用。 |
| 细胞类型 | 多选,选择要分析的具体细胞类型或聚类(如Epithelial cell等)。 |
| 物种 | 选择数据对应的物种,目前支持人和小鼠。 |
| 筛选因子 | 选择用于筛选样本或分组的标签(如Sample)。 |
| 筛选对象 | 多选,选择在筛选因子中要保留的样本或分组(如Sample_A)。 |
| Downsample | 是否对细胞进行降采样(抽取部分细胞)进行分析。 |
| Downsample_num | 若启用降采样,此处设置抽取的细胞数量。 |
| 备注 | 自定义备注信息。 |
TIP
CopyKAT非常适合用于对整个细胞群体进行初步的恶性/非恶性筛选。您可以选择所有的细胞类型作为输入,让算法自动完成分类。
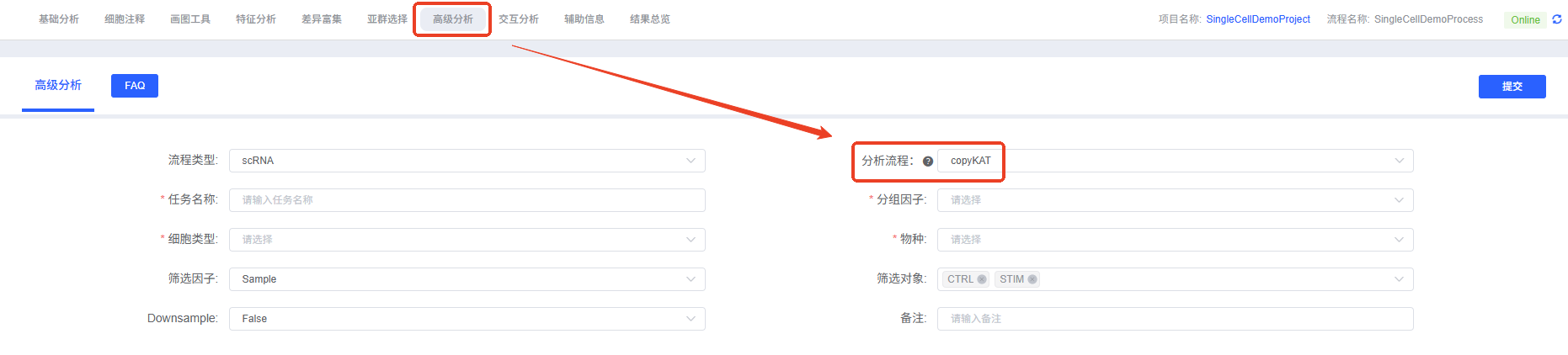
操作流程
- 进入分析模块:在云平台导航至“高级分析”模块,选择“copyKAT”。
- 创建新任务:为您的分析任务命名,并选择要分析的样本或项目。
- 配置参数:根据上述指南,选择要分析的细胞类型、分组信息等。
- 提交任务:确认参数无误后,点击“提交”按钮,等待分析完成。
- 下载与查看:分析结束后,在任务列表中下载并查看生成的分析报告和结果文件。
结果解读
CopyKAT的报告直观地展示了细胞分类和CNV的结果。
细胞类型推断热图
这是CopyKAT的核心结果图,展示了所有细胞的CNV谱以及对其“肿瘤/正常”身份的预测。
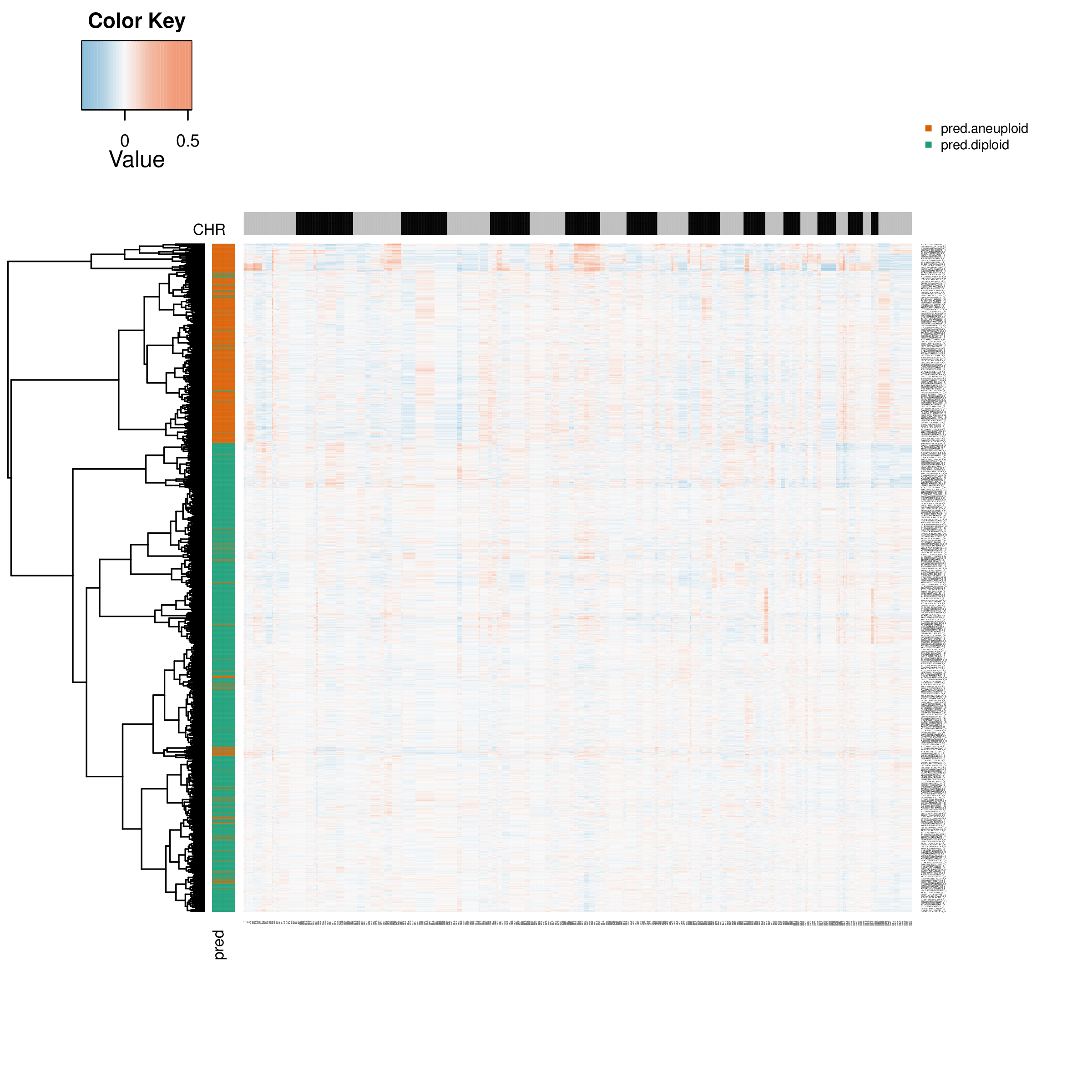
- 图表解读:
- 行:代表单个细胞。
- 列:代表按染色体位置排序的基因。
- 颜色(热图主体):红色表示拷贝数增加,蓝色表示拷贝数减少。
- 颜色(行注释):左侧的颜色条带是CopyKAT对细胞的分类结果。通常,绿色代表预测的“二倍体”(正常)细胞,橙色或其他暖色调代表预测的“非整倍体”(肿瘤)细胞。
- 分析要点:
- 观察是否存在一个明显的细胞群被预测为“非整倍体”,并且该群组显示出清晰的CNV模式(大片红色/蓝色区域)。
- 检查被预测为“二倍体”的细胞群是否CNV信号较弱(颜色接近中性)。
细胞分类结果降维图
为了直观地查看预测结果在细胞空间上的分布,报告会将分类结果投影到UMAP图上。
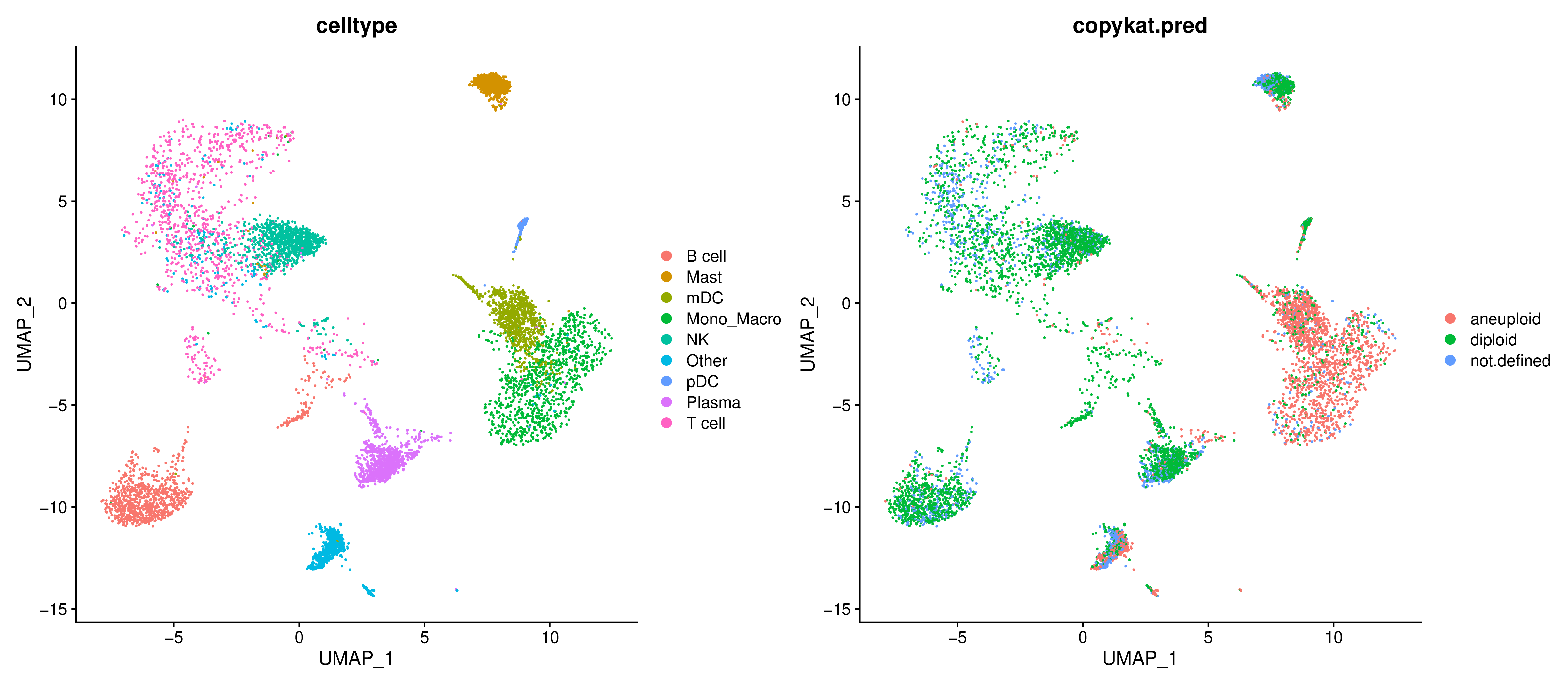
- 图表解读:图中每个点是一个细胞,颜色代表CopyKAT的预测结果(例如,正常 vs. 肿瘤)。
- 分析要点:检查预测的肿瘤细胞是否与您预先注释的肿瘤细胞聚类重合度高。
肿瘤亚克隆分析
在识别出肿瘤细胞后,CopyKAT会进一步对它们进行亚克隆分析。
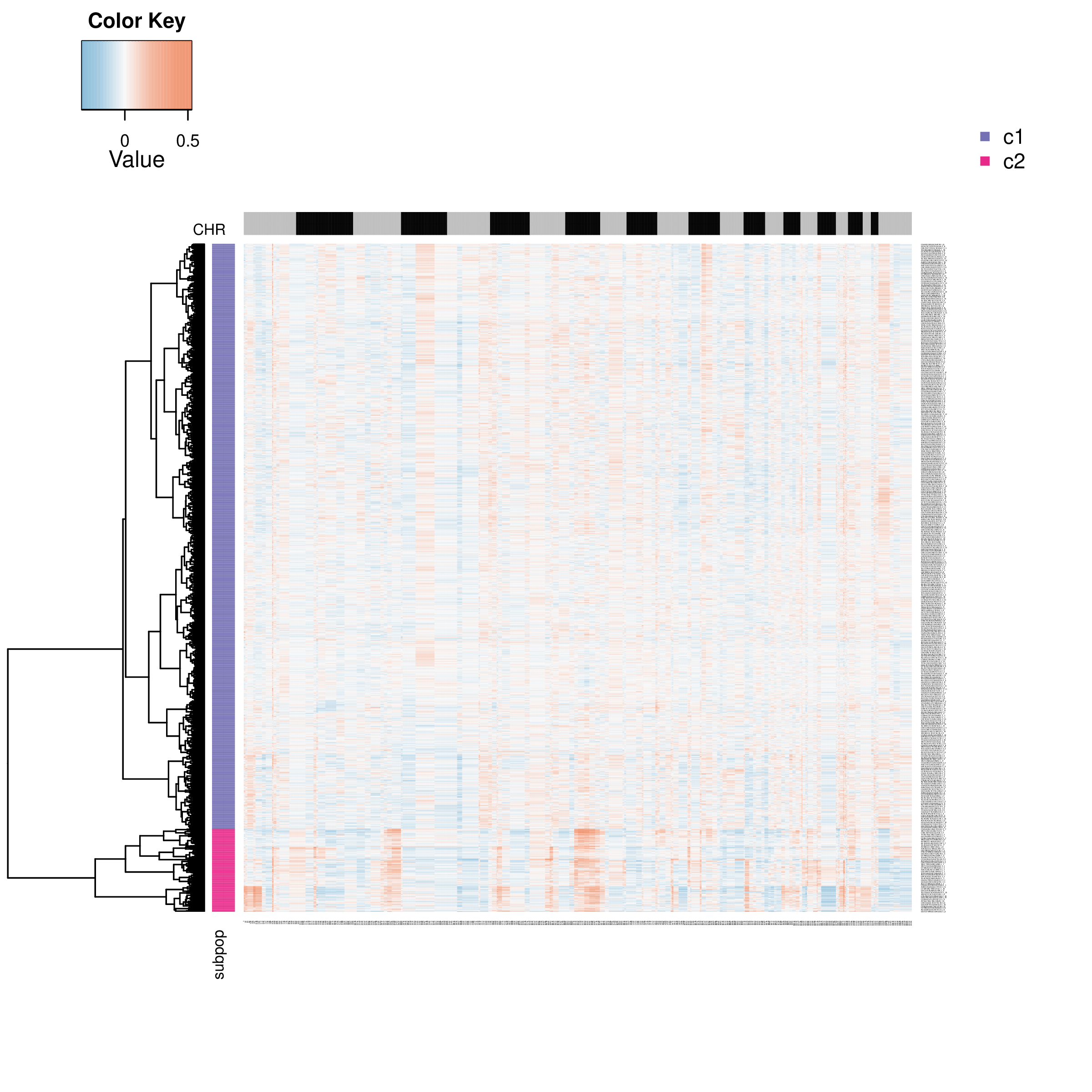
- 图表解读:该热图只展示被预测为“非整倍体”的肿瘤细胞。通过对它们的CNV模式进行聚类,可以将肿瘤细胞划分为不同的亚克隆(如热图行注释中的c1, c2等)。
- 分析要点:比较不同亚克隆之间的CNV差异。例如,亚克隆c1可能携带了亚克隆c2没有的特定染色体扩增,这可能代表了肿瘤的演进方向。
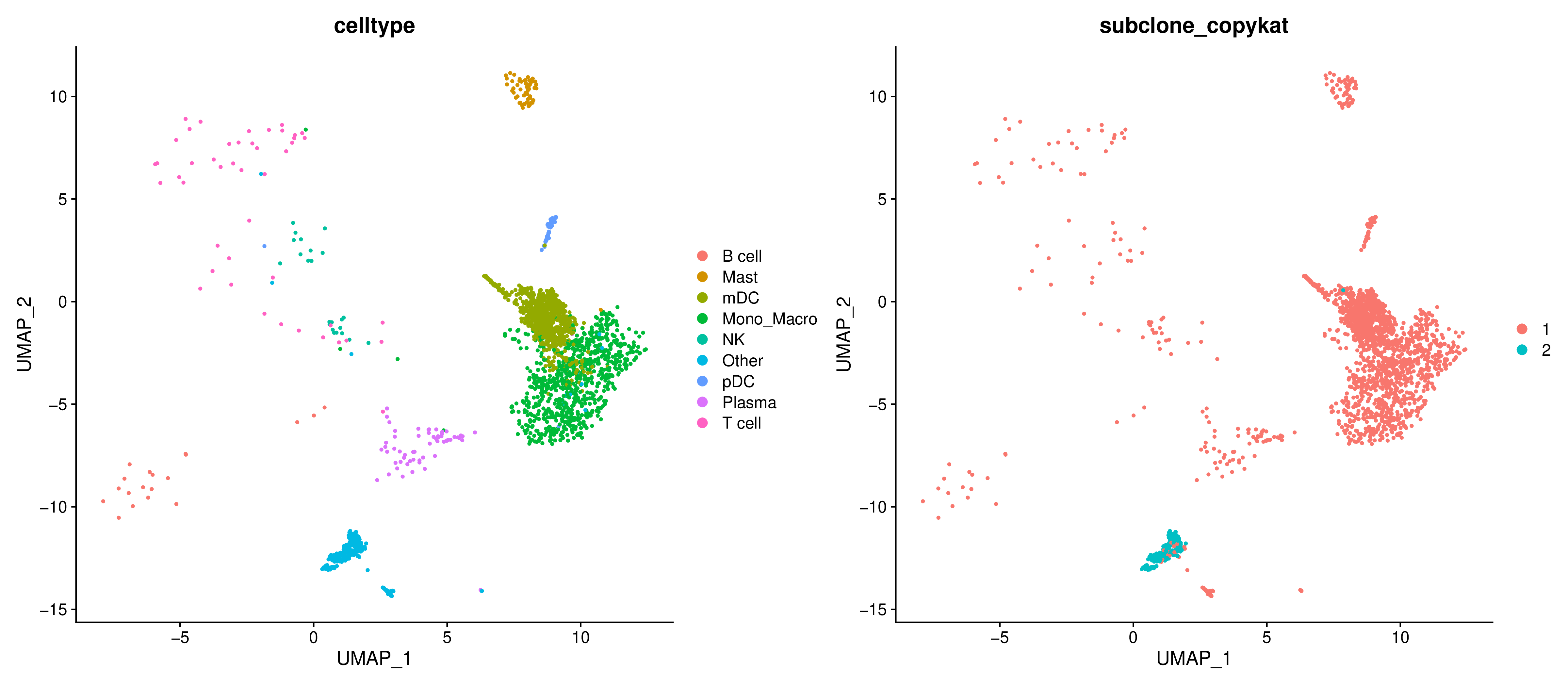
- 图表解读:将识别出的肿瘤亚克隆(c1, c2等)投影到UMAP图上,可以观察它们在细胞空间的分布关系。
应用案例
案例一:骨肉瘤中的肿瘤微环境
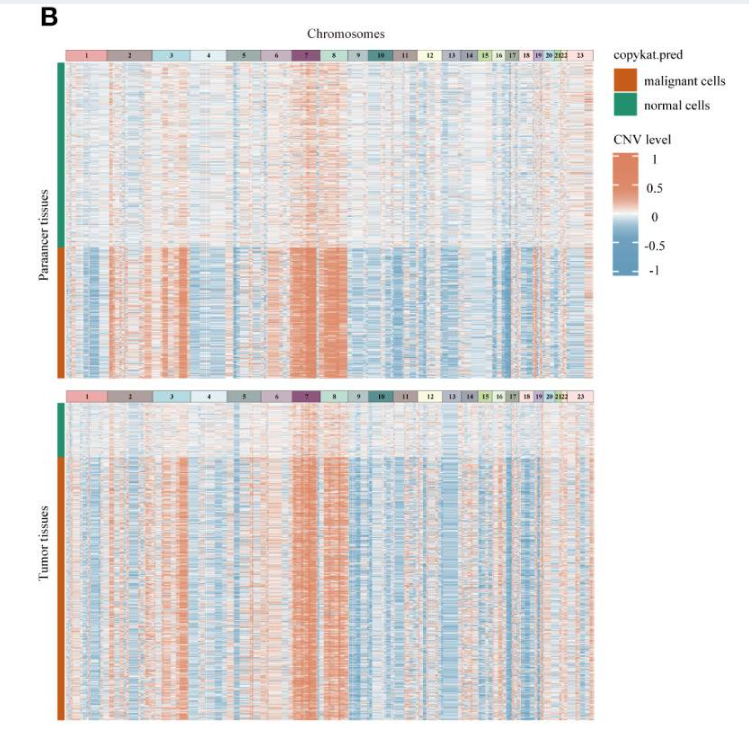
在一项研究未分化多形性肉瘤(UPS)的工作中,研究人员使用CopyKAT来区分恶性成纤维细胞和正常成纤维细胞。结果显示,CopyKAT成功地将成纤维细胞分为了两群,一群具有明显的CNV事件(恶性),另一群则基因组稳定(正常)。
案例二:鼻咽癌中的肿瘤演进
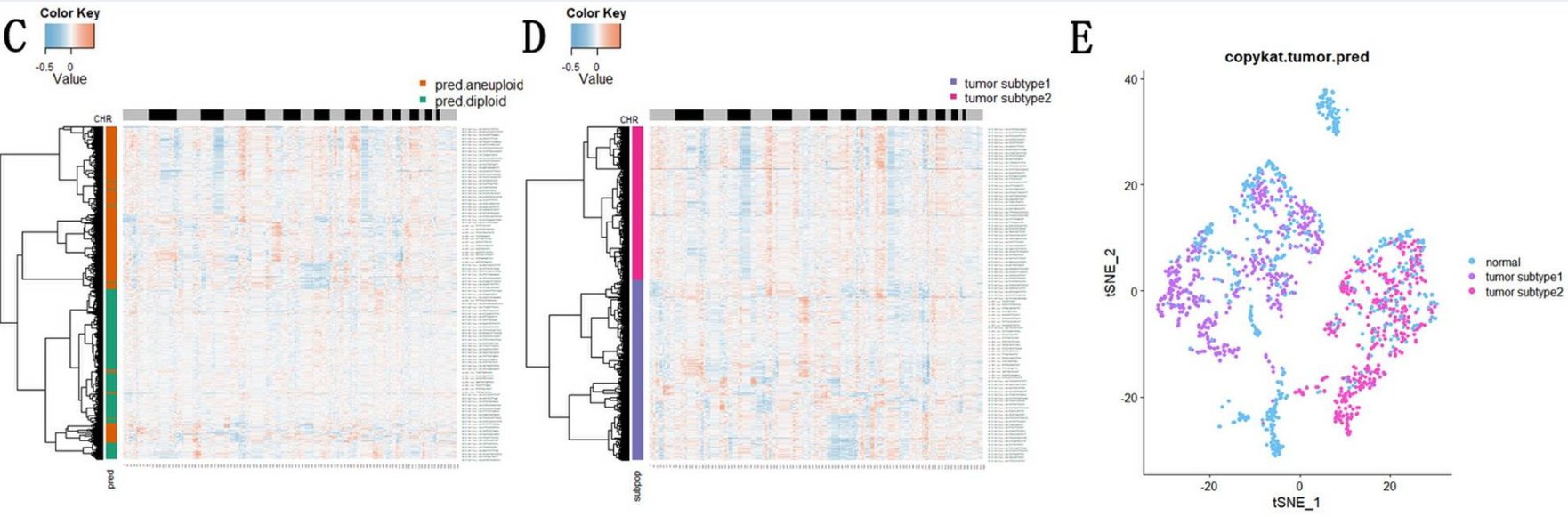
在对非角化性鼻咽癌(NPC)的研究中,CopyKAT被用来识别肿瘤细胞,并将其分成了两个主要的亚克隆。后续分析发现,这两个亚克隆在代谢和免疫逃逸相关的通路上存在显著差异,揭示了肿瘤内部的功能异质性。
注意事项
1. 结果并非金标准:CopyKAT是基于转录组数据的生物信息学推断,其对“肿瘤”或“正常”细胞的分类结果需要结合其他生物学证据(如关键基因标记、其他组学数据)进行综合判断,尤其是在肿瘤纯度较低或CNV信号不强的样本中。
2. 内参细胞的质量:CopyKAT自动寻找正常二倍体细胞作为内参是其核心优势,但也可能成为潜在的风险点。如果待分析的细胞群体中不包含真正的正常细胞,或者正常细胞比例极低,算法可能会错误地将一批CNV程度最轻的肿瘤细胞当作“正常”参照,导致后续所有细胞的CNV评估出现偏差。
3. 结合inferCNV进行验证:CopyKAT和inferCNV的原理不同,可以互为补充。当您对结果不确定时,可以尝试使用inferCNV(需要提供明确的正常细胞参照)进行交叉验证,观察两者结果的一致性。
常见问题解答 (FAQ)
Q1: copyKAT和inferCNV有什么区别?我应该选择哪一个?
A1: 主要区别在于是否需要手动指定正常参考细胞。
- inferCNV:需要用户明确提供一组“正常”细胞作为参考。它的优势在于,如果参考细胞非常可靠,结果会非常精确。适用于有明确正常对照组(如来自健康样本的细胞,或样本中确定为正常的非恶性细胞类型)的情况。
- copyKAT:自动从所有待分析细胞中识别最可能为正常的细胞作为内参。适用于无法确定哪些细胞是正常细胞,或者希望对整个样本进行无偏见的恶性/非恶性探索性分析的场景。
- 选择建议:如果有可靠的正常参照,两者都可以使用,inferCNV可能更经典;如果没有可靠的正常参照,强烈推荐使用copyKAT。
Q2: 为什么我的结果中有很多细胞被标记为“not defined”(未定义)?
A2: 这通常意味着这些细胞的CNV信号不够明确,算法无法以高置信度将其划分为“非整倍体”或“二倍体”。可能的原因包括:细胞本身的CNV水平较低、测序深度不足导致信号弱、或者是处于某种中间状态的细胞。
Q3: copyKAT可以用于非肿瘤研究吗?
A3: 理论上可以。任何导致大规模染色体拷贝数变化的生物学场景,如某些遗传病或细胞培养过程中的基因组不稳定性,都可以尝试使用copyKAT进行分析。但其算法是为肿瘤场景优化的,在其他场景下的解释需要更加谨慎。
参考文献
- Gao, R., et al. (2021). Delineating copy number and clonal substructure in human tumors from single-cell transcriptomes. Nature Biotechnology, 39(5), 599-608.
- Yuan, L. L., et al. (2022). Single-cell sequencing reveals the landscape of the tumor microenvironment in a skeletal undifferentiated pleomorphic sarcoma patient. Frontiers in Immunology, 13, 1019870.
- Chen, H., et al. (2023). Comprehensive single-cell transcriptomic and proteomic analysis reveals NK cell exhaustion and unique tumor cell evolutionary trajectory in non-keratinizing nasopharyngeal carcinoma. Journal of Translational Medicine, 21(1), 1-18.
