利用单细胞多组学数据发现新的基因调控网络
目录
基因调控网络(GRN)简介
什么是GRN?
基因调控网络(Gene Regulatory Network, GRN)描述了细胞内转录因子(Transcription Factors, TFs)与其他分子如何协同作用,精确控制基因表达时空的复杂互作体系。其核心是解答“哪个调控因子在何时何地控制哪个基因”的问题。
GRN的核心要素
- 转录因子 (TF): 识别并结合特定DNA序列,启动或抑制基因转录的蛋白质。
- 顺式调控元件 (CRE): DNA上的功能序列,如启动子和增强子,是TF的结合位点。
- 靶基因: 受一个或多个TF调控的基因。
GRN的研究意义
- 揭示生命机制:阐明细胞分化、组织发育等基本生命过程的分子基础。
- 解析疾病机理: 识别疾病(如癌症、神经退行性疾病)发生发展的关键调控失常。
- 发现药物靶点: 为精准医疗提供新的治疗靶点和干预策略。
传统单组学研究GRN的局限
scRNA-seq:只见“表达结果”,难寻“调控原因”
- 优势: 精确测量单个细胞的基因表达水平。
- 局限: 无法直接获取染色质状态信息,难以区分直接调控与间接效应,调控网络依赖于推断。
scATAC-seq:只见“调控潜力”,难判“实际功能”
- 优势: 识别全基因组范围内的开放染色质区域(潜在的CRE)。
- 局限: 无法直接测量基因表达,难以确认CRE的实际功能及其靶基因,同样依赖于推断。
核心困境:数据割裂,关联靠“猜”
传统单组学方法如同盲人摸象,scRNA-seq和scATAC-seq提供了关于GRN的不同维度的信息,但由于数据来源不同细胞,二者之间的关联是推测而非实测的,限制了调控关系认定的准确性。
单细胞多组学如何突破局限
核心突破:同一细胞内同步测量,实现直接关联
单细胞多组学(如10x Genomics Multiome)技术革命性地实现了在同一个细胞内同时捕获**转录组(scRNA-seq)和染色质可及性(scATAC-seq)**信息。
技术优势
1. 直接关联CRE与基因表达
- 告别推测: 直接利用同一细胞的数据,将开放的CRE与靶基因的表达水平进行关联分析,显著提高准确性。
2. 精准解析细胞类型特异性调控
- 消除平均效应: 在高度异质的组织中,能够构建每个细胞亚群特有的GRN,避免关键信息被掩盖。
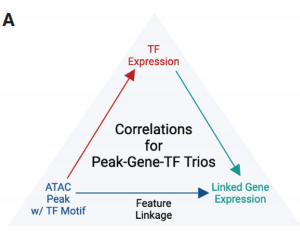
3. 构建高可信度的“TF-CRE-Gene”三元调控轴
- 整合分析: 结合TF motif富集分析、CRE可及性及靶基因表达,构建从上游调控因子到下游靶基因的完整调控链路。
应用案例解析
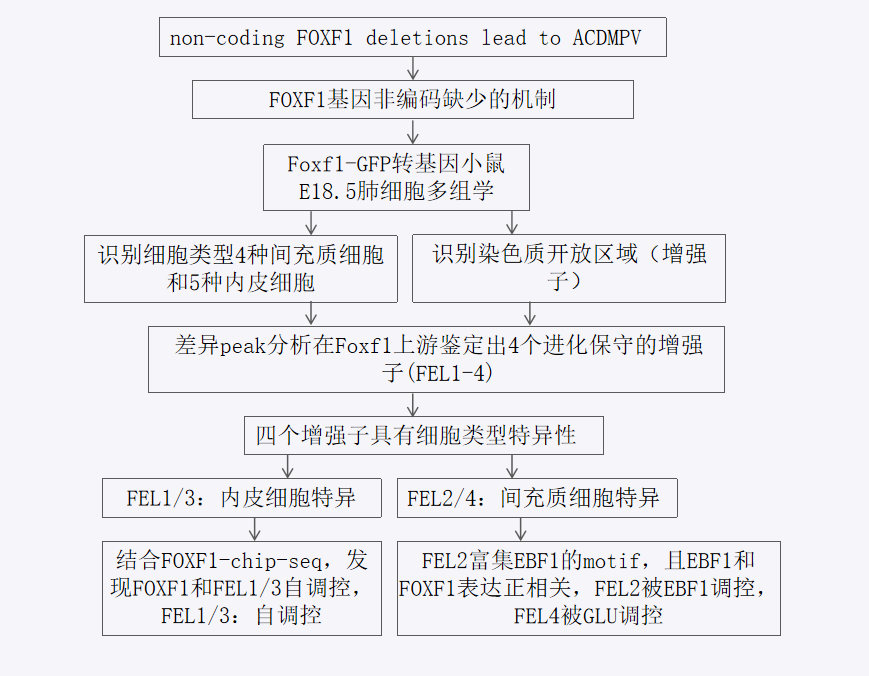
案例1:解析FOXF1基因在肺发育中的复杂调控网络
研究背景:关键发育基因的调控“黑盒”
FOXF1是肺泡发育的关键转录因子,其非编码区的突变可导致严重的先天性疾病(ACDMPV)。然而,这些非编码元件如何精确调控FOXF1在不同细胞类型中的表达,一直是个未解之谜。传统方法因无法同时获取同一细胞的基因表达和染色质状态,难以破解这一难题。
核心突破:单细胞多组学实现“一核双查”
研究人员利用单核多组学测序技术,在同一个细胞核内同时获取了snRNA-seq和snATAC-seq数据,实现了对FOXF1表达与其调控元件活性的直接关联分析。
研究路线图:破解FOXF1调控密码
- 多组学数据捕获: 对
Foxf1基因报告小鼠的肺组织进行单核多组学测序,获得数千个细胞核的配对snRNA-seq和snATAC-seq数据。 - 细胞特异性CRE识别: 结合两组学数据,在精确定义的细胞类型(如内皮细胞、间充质细胞)中,识别出
Foxf1基因座上游的四个关键增强子(FEL1-4),并发现它们的开放具有高度的细胞类型特异性。 - 调控关系直接关联: 通过多组学数据的内在联系,直接证实了这些特异性开放的增强子与
FOXF1在相应细胞中的表达高度相关。 - 上游TF预测与验证: 基于增强子序列的motif分析,预测了调控这些增强子的上游转录因子(如EBF1, GLI1),并通过后续实验(如ChIP-seq、报告基因实验)验证了这些TF的直接调控作用。
- 构建完整调控网络: 整合所有信息,构建了一个包含“上游TF → 细胞类型特异性增强子 → FOXF1”以及FOXF1自调控环路在内的完整GRN。
- 临床转化验证: 将在小鼠模型中的发现与ACDMPV患者的基因组缺失数据进行比对,证实了这些增强子的缺失是导致疾病的关键原因,完成了从基础研究到临床意义的转化。
单细胞多组学的核心贡献
- 实现“因果”关联: 突破了传统方法只能分别观测表达和染色质状态的局限,在单细胞内建立了调控元件(因)与基因表达(果)的直接联系。
- 赋予调控“细胞身份”: 在复杂的组织环境中,精确地为每一个调控关系打上了“细胞类型”的标签,揭示了
FOXF1在不同细胞谱系中被不同机制调控的精密图景。 - 指导功能实验: 多组学分析预测的关键TF和CRE,为后续的分子生物学验证提供了精确的“导航”,极大提高了实验效率和成功率。

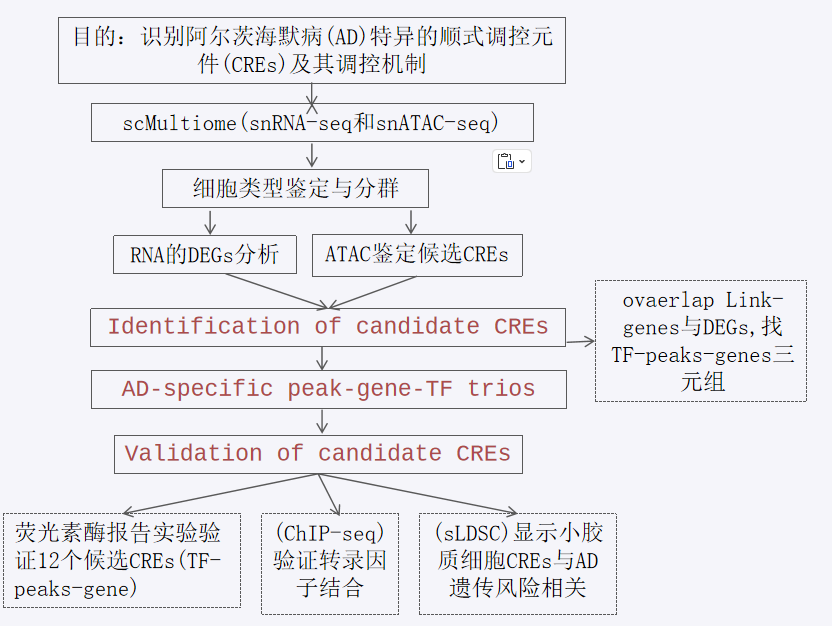
案例2:解构阿尔茨海默病(AD)的细胞特异性调控网络
研究背景:AD研究的“细胞异质性”与“非编码”双重挑战
阿尔茨海默病(AD)的病理机制极为复杂。一方面,大脑由多种细胞(神经元、小胶质细胞、星形胶质细胞等)构成,它们在AD进程中扮演不同角色,传统bulk测序无法区分其特异性变化。另一方面,大量AD遗传风险位点位于非编码区,其功能未知。如何将这些风险位点与特定细胞类型的基因调控异常联系起来,是AD研究的关键瓶颈。
核心突破:多组学技术精准绘制AD大脑的“细胞-调控”图谱
通过对AD患者和健康对照的脑组织进行大规模单核多组学测序,研究者得以在单细胞分辨率下,同时解析基因表达和染色质可及性的变化,为破解AD的细胞特异性调控失常提供了前所未有的机会。
研究路线图:解构AD的复杂调控网络
- 大规模多组学数据生成: 对超过10万个来自AD患者与健康对照脑组织的细胞核进行多组学测序。
- 整合数据定义细胞亚型: 利用加权最近邻(WNN)算法整合RNA和ATAC数据,构建了包含36个精细亚群的脑细胞图谱,并发现AD患者中特定神经元和小胶质细胞亚群的比例失调。
- 识别AD特异性调控变化: 在细胞类型特异的水平上,系统识别了AD相关的差异表达基因(DEGs)和差异可及性染色质区域(DARs)。
- 构建“CRE-Gene”关联: 通过计算将数万个差异CRE与它们的潜在靶基因直接关联起来,形成了AD特异性的调控网络。
- 鉴定核心调控因子: 在此网络基础上,进一步推断出驱动不同细胞类型病理变化的核心转录因子,如神经元中的
ZEB1和小胶质细胞中的MAFB。 - 关联遗传风险: 最关键的是,研究者发现AD的遗传风险位点(GWAS hits)在小胶质细胞的特异性CREs中显著富集,首次在全基因组层面、以细胞类型特异的方式,为AD的遗传易感性提供了功能性解释。
单细胞多组学的核心贡献
- 破解“细胞异质性”难题: 高分辨率揭示了AD病理在不同脑细胞类型中的独特分子特征,将研究从“组织水平”推进到“细胞亚群水平”。
- 连接“遗传”与“功能”: 成功地将抽象的GWAS遗传风险位点与具体的、在特定细胞中发挥作用的调控元件联系起来,填补了AD遗传学和神经生物学之间的巨大鸿沟。
- 提供精准靶点: 发现的如
ZEB1和MAFB等细胞类型特异性的核心调控因子,为开发针对特定细胞病理过程的AD治疗策略提供了极具潜力的新靶点。

案例3:描绘CD8+ T细胞分化的增强子调控蓝图
研究背景:T细胞命运决定的“增强子密码”与“TCR噪音”
在抗肿瘤免疫中,CD8+ T细胞如何分化为具有持久杀伤力的组织驻留记忆T细胞(Trm),是决定免疫治疗成败的关键。这一过程受到复杂的GRN调控,但研究面临两大挑战:1)增强子未知:决定T细胞命运的关键增强子及其调控的TF尚不明确。2)TCR异质性:肿瘤浸润的T细胞具有海量的不同TCR克隆,其异质性带来的“噪音”掩盖了共通的分化调控规律。
核心突破:TCR标准化结合多组学,消除噪音,聚焦调控
研究者设计了精巧的实验策略:首先通过采用TCR转基因小鼠模型,将T细胞的TCR“标准化”,从而排除了TCR异质性的干扰。在此基础上,对这些TCR均一的T细胞进行单细胞多组学测序。
研究路线图:绘制Trm分化调控图
- TCR均一化与多组学测序: 在多种肿瘤模型中,采用TCR转基因的P14 CD8+ T细胞,进行配对scRNA-seq和scATAC-seq,获得“干净”的多组学数据。
- 构建增强子调控网络(eRegulons): 应用SCENIC+算法,整合RNA和ATAC数据,首次构建了T细胞分化过程中的“TF-增强子-靶基因”三维调控网络。
- 发现关键拮抗调控轴: 通过分析eRegulon活性,发现
KLF2是抑制Trm分化的关键因子,而BATF是促进Trm分化的核心因子,二者形成拮抗关系,共同决定T细胞命运。 - 功能验证与临床关联: 通过基因过表达和敲低实验,在动物模型中成功验证了
KLF2和BATF的调控功能,并发现BATF的表达水平与人类胰腺癌患者的良好预后显著相关。
单细胞多组学的核心贡献
- 实现“增强子级别”的GRN解析: 将T细胞分化的调控研究,从传统的“TF-基因”二维关系,提升到了包含增强子在内的“TF-增强子-基因”三维层面,极大提升了网络的精细度和准确性。
- 从“相关性”到“因果性”的跨越: 通过TCR标准化排除了混杂因素,使得多组学数据揭示的调控关系更接近“因果”,为后续功能验证提供了高可信度的候选靶点。
- 发现可干预的治疗靶点: 鉴定出的
KLF2/BATF调控轴,不仅深刻揭示了T细胞分化的内在机制,也为开发新型免疫疗法(如通过抑制KLF2或激活BATF来增强抗肿瘤免疫)提供了明确的分子靶点。
附录:核心技术简介
SCENIC+:从多组学数据到增强子调控网络
在众多单细胞多组学GRN推断工具中(如Pando、Linger、CellOracle、FigR、RENIN等),SCENIC+是具代表性的计算框架之一。它专门整合scRNA-seq和scATAC-seq数据,通过先进算法构建"TF-增强子-靶基因"三维调控网络(eRegulons),是实现上述案例分析的核心工具。
Peaks-to-Gene (P2G) 关联分析
P2G关联是连接染色质开放区域(Peaks)与其靶基因的过程,是构建GRN的基础。常用策略包括:
- 距离法:简单地将Peak关联到基因组上最近的基因。
- 相关性法:计算Peak开放度与基因表达值的相关性,高相关则认为存在调控关系。
- 回归模型法:构建预测模型,筛选对基因表达有显著影响的Peaks。
技术优势总结
1. 数据完整性
| 技术 | 基因表达 | 染色质状态 | 细胞类型 | 直接关联 |
|---|---|---|---|---|
| scRNA-seq | ✅ | ❌ | ✅ | ❌ |
| scATAC-seq | ❌ | ✅ | ✅ | ❌ |
| 多组学 | ✅ | ✅ | ✅ | ✅ |
2. 分析精度提升
- 减少推测: 直接观察关联关系
- 提高准确性: 避免假阳性关联
- 增强特异性: 细胞类型特异的调控网络
3. 生物学发现能力
- 新调控因子发现: 如KLF2在TIL中的作用
- 疾病机制解析: 如AD相关的调控元件
- 发育过程理解: 如T细胞命运决定
4. 实验验证效率
- 候选基因筛选: 通过多组学数据缩小范围
- 功能验证: 基于预测结果设计实验
- 机制解析: 从分子水平理解调控过程
总结:单细胞多组学——GRN研究的范式革命
单细胞多组学技术,特别是scRNA-seq与scATAC-seq的结合,通过在单个细胞内同步解析“基因表达”和“染色质开放状态”,为基因调控网络(GRN)的研究带来了革命性的突破。它克服了传统组学“只见森林,不见树木”的局限,使我们能够:
- 直击细胞异质性:精准区分不同细胞亚群的特异性调控模式。
- 构建三维调控网络:从“TF-基因”的二维关系,升级到“TF-增强子-基因”的三维视图。
- 增强因果推断:通过直接关联调控元件的“开闭”与基因的“表达”,使调控关系的推断更接近因果。
本文通过解析肺发育、阿尔茨海默病和肿瘤免疫三个前沿案例,清晰地展示了单细胞多组学如何帮助科学家发现新的疾病驱动基因、识别细胞特异性的治疗靶点,并描绘出复杂的细胞分化蓝图。这项技术不仅在推动基础生命科学的边界,更在为精准医疗和药物开发铺设坚实的道路。
参考文献
- FOXF1调控机制: Dong, et al. Single-cell multiomics reveals the orchestrated regulation of lung development. Nature Cell Biology, 2023. (案例1)
- 阿尔茨海默病研究: Morabito, et al. Single-cell multi-omic analysis identifies cell-type-specific cis-regulatory elements and transcription factors in Alzheimer’s disease. Nature Genetics, 2021. (案例2)
- CD8+ T细胞命运决定: Ekiz, et al. CD8+ T cell differentiation in cancer is regulated by an enhancer network orchestrated by the transcription factors KLF2 and BATF. Immunity, 2024. (案例3)
- SCENIC+方法学: Granja, et al. SCENIC+: a framework for single-cell multi-omics gene regulatory network analysis and cell-state identification. Nature Methods, 2021.
注:本文档基于实际研究案例编写,旨在帮助理解单细胞多组学在GRN推断中的优势和应用。所有图片均来自原始研究文献,版权归原作者所有。
