参数模板
时长: 9 分钟
字数: 2.2k 字
更新: 2026-01-21
阅读: 0 次
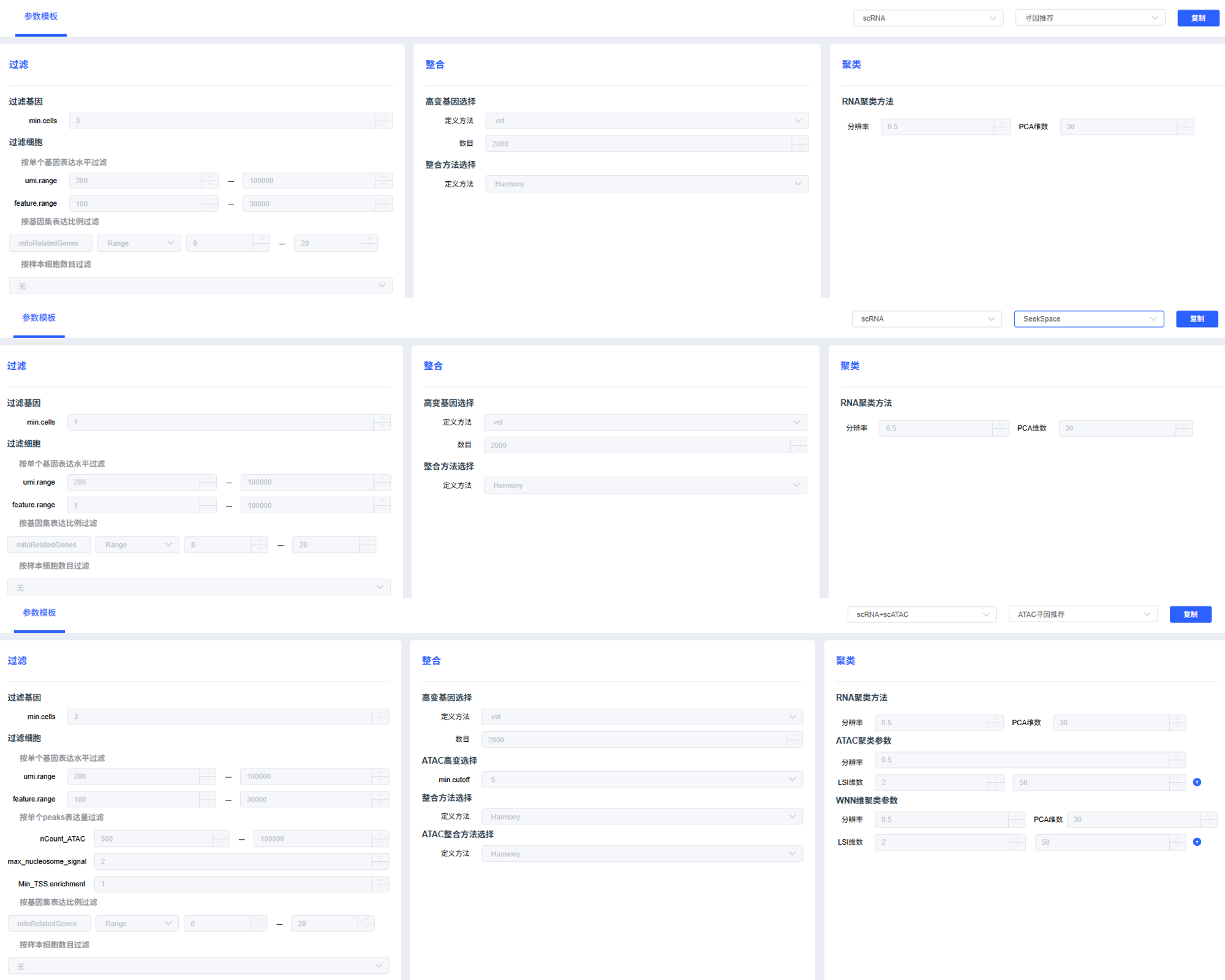
默认参数模板
云平台配置默认模板,包含所有分析的相关参数和颜色配置,共六个板块(过滤、整合、聚类、差异分析、富集分析和配色方案)。该模板不可修改。
NOTE
默认参数模板为平台推荐配置,建议在复制模板基础上根据实际需求进行个性化调整。
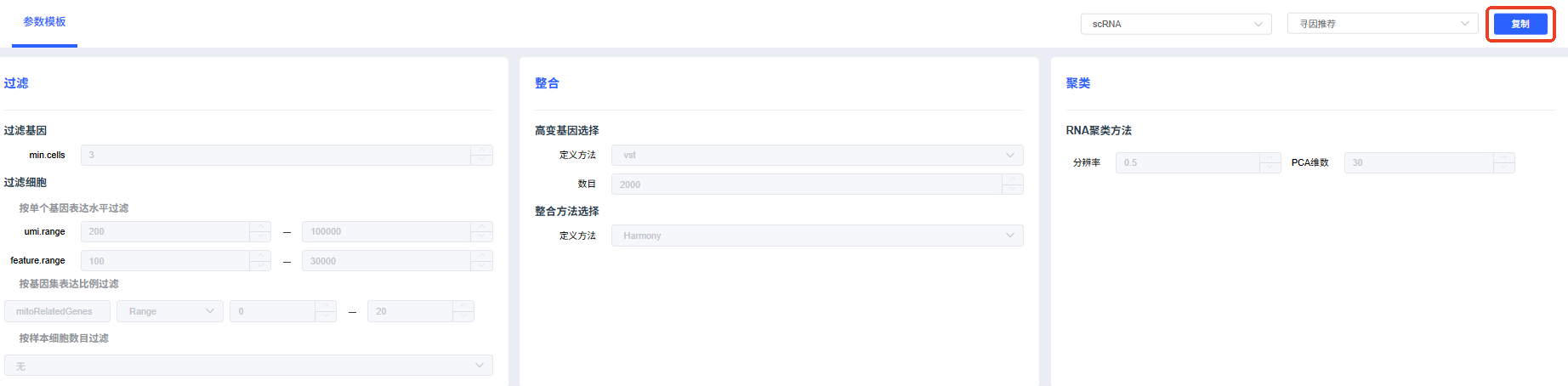
复制参数模板
如需修改模板,可点击页面右上角复制按钮,设置模板名称,并按需设置参数。
过滤参数
scRNA
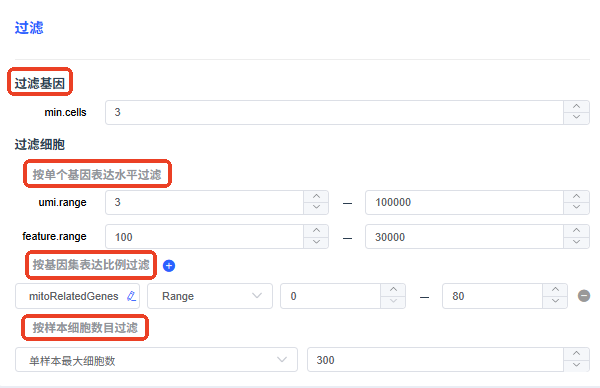
- min.cells:设置每个基因至少在多少个细胞中表达,低于该阈值的基因会被过滤。
- umi.range:设置每个细胞检测到的 UMI 范围阈值,UMI 值不在此范围内的细胞会被过滤。
- feature.range:设置每个细胞表达的基因数范围阈值,表达基因数不在此范围内的细胞会被过滤。
- 按基因集表达比例过滤:
- a. 基因集设置:点击 🖌,可从“我的基因集”添加/增加基因集进行过滤,默认选择线粒体基因集。点击【+】新增基因集进行过滤。
- b. 过滤方式:
- MAD:根据细胞整体基因集表达比例计算阈值,过滤掉基因集占比高于此值的所有细胞。
- range:根据设置的最大最小值,过滤掉线粒体基因占比不在此区间内的所有细胞。
- 按样本细胞数目过滤:多样本分析时,不同样本检测到的细胞数量可能差异较大,可通过细胞抽样方法将各样本细胞数量维持在相同水平。
scRNA + scATAC
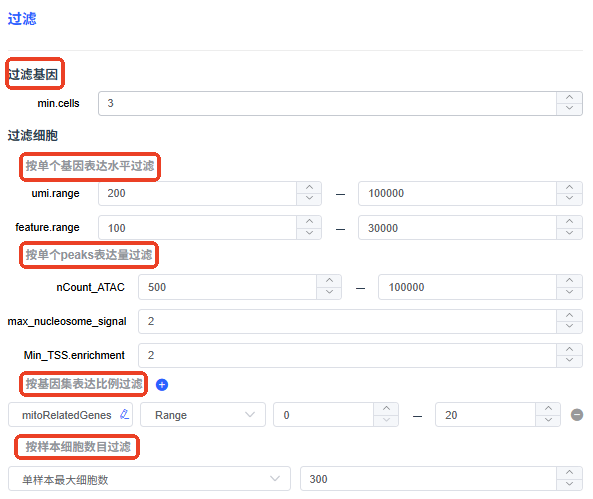
- min.cells:设置每个基因至少在多少个细胞中表达,低于该阈值的基因会被过滤。
- umi.range:设置每个细胞检测到的 UMI 范围阈值,UMI 值不在此范围内的细胞会被过滤。
- feature.range:设置每个细胞表达的基因数范围阈值,表达基因数不在此范围内的细胞会被过滤。
- nCount_ATAC:每个细胞中检测到 ATAC 转座酶事件的总 count 数。
- max_nucleosome_signal:每个细胞中核小体信号,过滤染色状态不好的细胞(如核小体过度降解、细胞坏死等)。
过滤大于 2 的细胞
- Min_TSS.enrichment:每个细胞中 TSS 富集分数,过滤染色质状态不好的细胞(如死细胞)。
过滤掉 TSS 小于 2 的细胞
- 按基因集表达比例过滤:
- a. 基因集设置:点击 🖌,可从“我的基因集”添加/增加基因集进行过滤,默认选择线粒体基因集。点击【+】新增基因集进行过滤。
- b. 过滤方式:
- MAD:根据细胞整体基因集表达比例计算阈值,过滤掉基因集占比高于此值的所有细胞。
- range:根据设置的最大最小值,过滤掉线粒体基因占比不在此区间内的所有细胞。
- 按样本细胞数目过滤:多样本分析时,不同样本检测到的细胞数量可能差异较大,可通过细胞抽样方法将各样本细胞数量维持在相同水平。
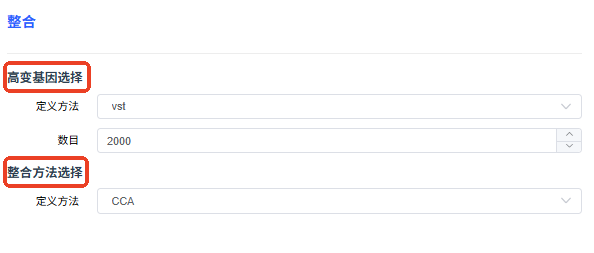
整合参数
scRNA
- 高变基因方法:选择降维过程中查找高变基因的方法,常用 vst 方法。
- 高变基因数量:设置高变基因 / bins 的数量。
- 整合方法:多样本整合分析时,若样本间批次效应较小可用 merge 方法,批次效应较大可用 Harmony(默认)/ CCA / RPCA 算法去除批次效应。
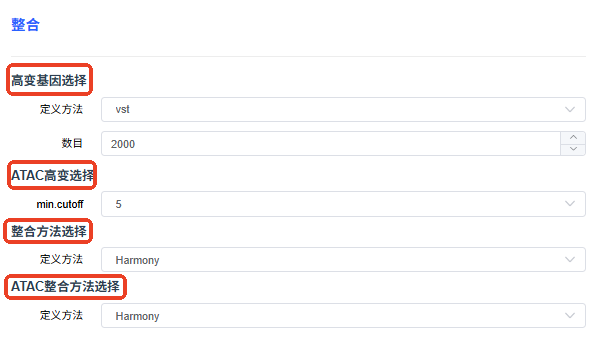
scRNA + scATAC
- 高变基因方法:选择降维过程中查找高变基因的方法,常用 vst 方法。
- 高变基因数量:设置高变基因 / bins 的数量。
- ATAC 高变选择:计算高变 peaks 时用于过滤低开放 peaks 的阈值,如 min.cutoff=5 表示至少在 5 个细胞中开放的 peaks 才纳入计算。
- 整合方法选择:同 scRNA,批次效应较大可用 Harmony / CCA / RPCA。
- ATAC 整合方法选择:根据 RNA 整合方法自动填充,ATAC 和 RNA 分开独立进行批次矫正和整合。
多样本整合完成后,仍为一个 ATAC assay 和 RNA assays(如用 CCA / RPCA 还会有 integrated assay,存放 RNA 矫正后表达矩阵)。
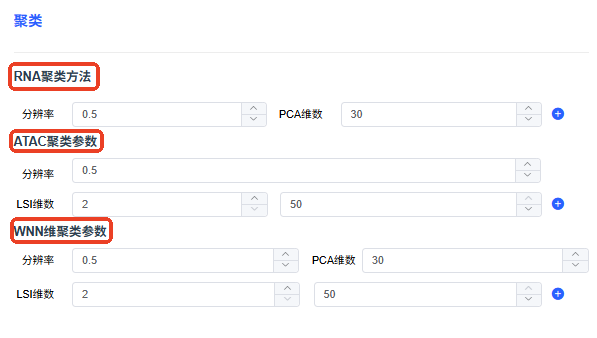
聚类参数
scRNA
- 分辨率:设定细胞聚类分群时的分辨率(resolution,默认 0.5),可调整 cluster 数量,resolution 越高 cluster 越多。
- PCA 维数:根据 PCA 轴突拐点所对应的 PCA 数量,设定聚类时使用降维数量(默认 30),一般在 10~30 之间,细胞数越多数量越大。
scRNA + scATAC
- ATAC 聚类参数:基于 ATAC 开放性,以 LSI 降维聚类,只考虑 ATAC 异质性。
- WNN 维聚类参数:综合 PCA 和 LSI 降维聚类,综合考虑 ATAC 和 RNA 异质性。
基于 Seurat 的 Signac 工具,LSI 维数默认值 dims = 2:50,维数太低可能丢失重要生物学变异,太高可能导致过度聚类。
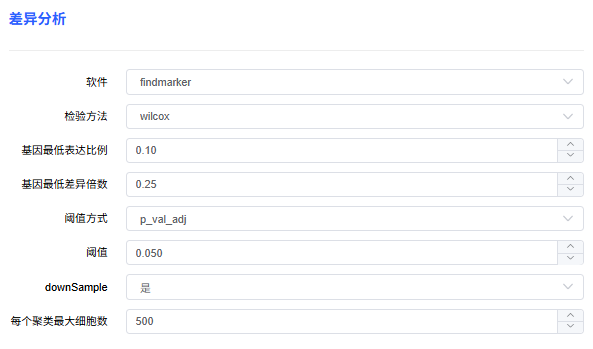
差异分析参数
- 软件:presto 或 findmarker。
- 检验方法:选择寻找差异基因的分析方法,默认 wilcox 方法。
- 基因最低表达比例:设置基因表达细胞的最小比例阈值(默认 0.1),低于该阈值的基因不列入差异基因列表。
- 基因最低差异倍数:设置上下调差异倍数的最小绝对值(average Log2 fold change,默认 0.25)。
- p_val_adj 阈值:设定判定 cluster 间基因表达显著差异的筛选阈值,默认 Padj < 0.05。
- downSample:是否进行细胞抽样,默认是。
- 每个聚类最大细胞数:设置每个细胞群中最大细胞数,针对细胞抽样情况。
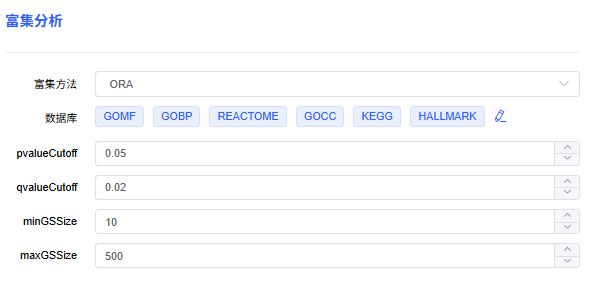
富集分析参数
- 富集方法:Over-Representation Analysis (ORA) 或 Gene Set Enrichment Analysis (GSEA)。
- 数据库:选择富集分析数据库,点击 🖌 可从“我的数据库”添加/增加基因集。
- pvalueCutoff:设置富集结果的 pvalue 阈值,高于该值的结果会被过滤。
- qvalueCutoff:设置富集结果的 qvalue 阈值,高于该值的结果会被过滤。
- minGSSize:设置基因集的最小基因数量,低于该值的结果会被过滤。
- maxGSSize:设置基因集的最大基因数量,高于该值的结果会被过滤。
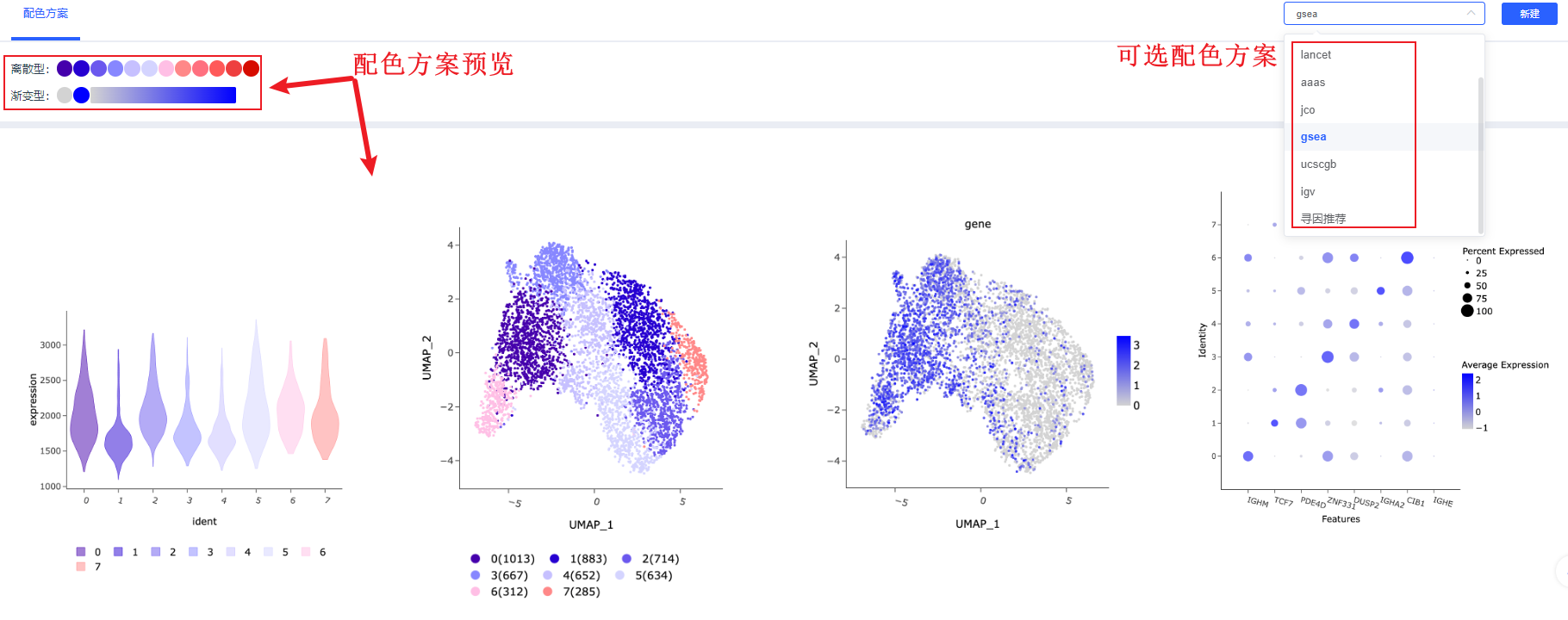
配色方案设置
- 选择配色方案:在配色方案板块右上角选择所需配色方案,离散型颜色用于分类/分组图,渐变型颜色用于连续数值图,下方预览区展示示例。
- 自定义配色方案:
- a. 新建配色方案:点击“配色方案预览”右侧新建按钮,弹出编辑颜色窗口。点击配色方案名称行展示颜色设置,每次只能展开一种配色方案。点击【➤】新建颜色配色,填写方案名称(不能重复),编辑颜色,点击【重置】可清空离散型颜色。
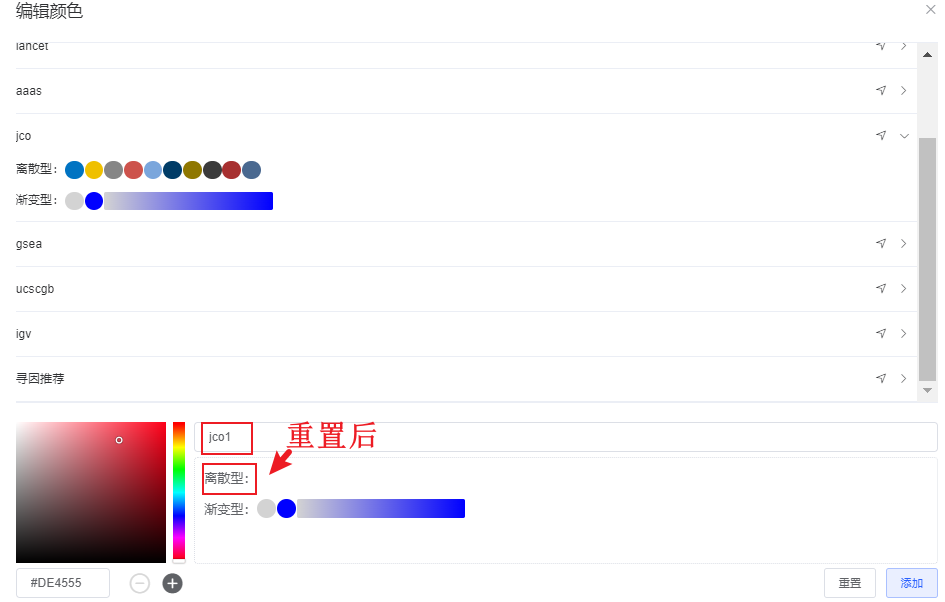
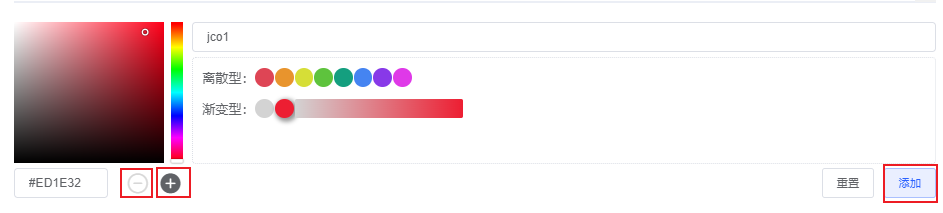
- 添加颜色:在颜色盘选择颜色,点击下方【+】添加到离散型配色。
- 删除颜色:点击离散型颜色出现阴影,再点击下方【-】删除。
- 修改颜色:确定颜色点后,点击颜色盘其他颜色即可修改,支持离散型和连续型。
- 点击【添加】按钮新建配色方案。
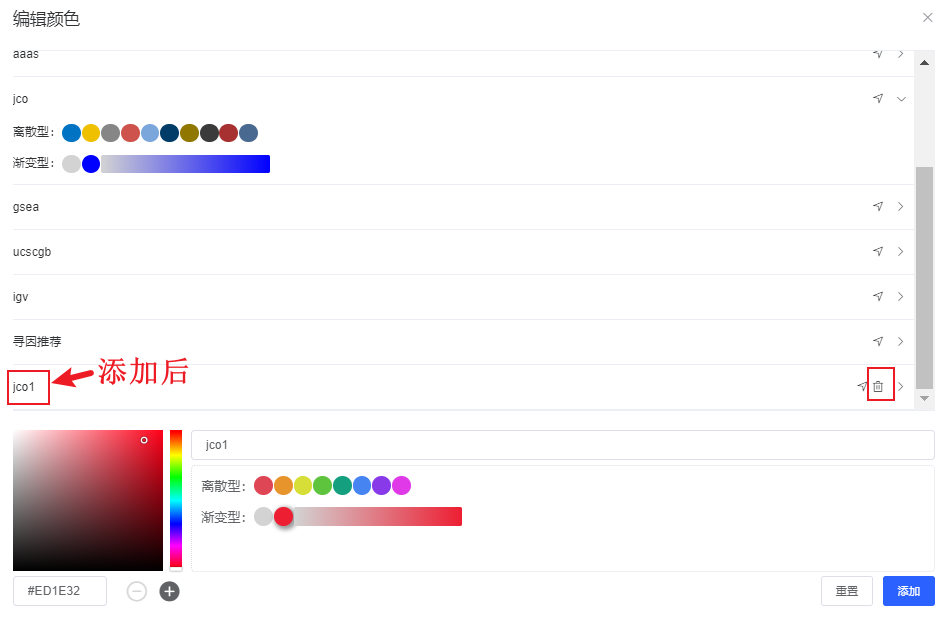
- b. 删除配色方案:自定义配色方案支持删除,点击方案列表中该方案的【删除】按钮即可。
- 保存和删除模板:点击参数模板页面右上角【保存】按钮保存修改,点击【删除】按钮删除弃用模板。
