基础分析 - scRNA+scATAC
时长: 6 分钟
字数: 1.5k 字
更新: 2026-01-21
阅读: 0 次
“基础分析”是分析流程的初始模块,可进行基因、线粒体过滤、去批次整合聚类,保留质量合格的细胞用于后续分析。分析流程创建及过滤整合聚类流程如下:
初始阶段
- 【新建流程】创建单细胞分析流程。项目开始一般选择样本进行大群分析,后续可选择注释好的细胞类型进行亚群分析。
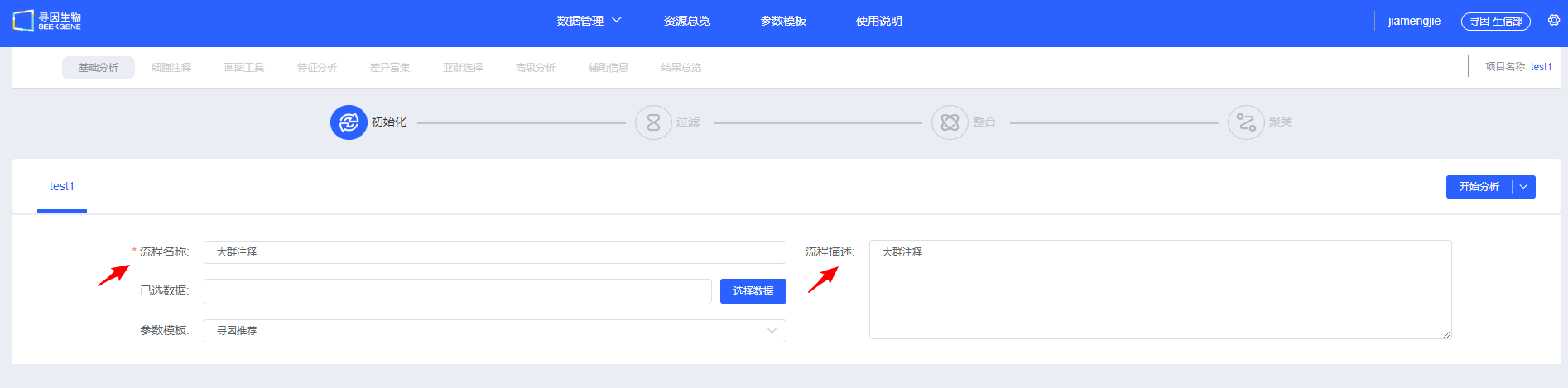
- 填写“流程名称”及“流程描述”,用于后续查找和了解流程信息。
- 【选择数据】选择待分析的样本,可选择已经整合好的多样本数据,也可选多个单样本进行过滤整合,无论是整合好的还是单样本的,都需要选择 ATAC 标准分析结果的数据类型。

TIP
数据详细来源可查看《我的数据》。
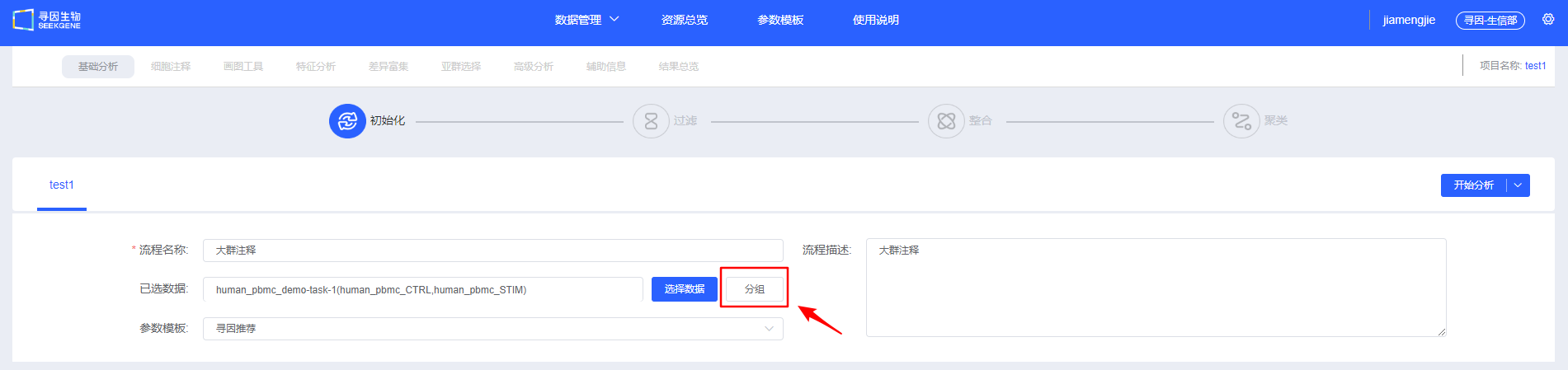
- 填写信息,选择待分析的数据后,选择流程类型为 scRNA+scATAC,可以选择【开始分析】手动进行过滤整合和聚类,也可以点击下拉框选择【基础分析】、【基础分析+细胞注释】、【基础分析+细胞注释+差异富集】或【基础分析+细胞注释+差异富集+画图工具】快速进行自动分析。

过滤
- 【开始分析】后的“图表数据”会统计样本 RNA 表达数据的 UMI、线粒体占比及基因表达情况,也会统计 ATAC 数据的核小体信号、TSS 富集分数和细胞中 peaks 等指标,简要展示各样本各个质量指标信息。

- 展开按钮可查看默认参数,用户可参考已发表单细胞文章方法中的过滤参数进行【过滤】。
TIP
如果选择的是已经整合的数据,点击【过滤】会提示是否需要跳过过滤整合步骤。如果不需要调整参数可跳过进行后续分析,如需调整过滤阈值则取消跳过,重新进行过滤整合。

- 【过滤】后可查看各样本过滤后质量信息,同时可对单样本进行个性化调整,保证整体样本质量一致。

WARNING
避免机械性依赖“拉高细胞数”的操作(如仅凭经验值强行放宽下限)。若瀑布图拐点不清、背景高或低 UMI 群占比异常,强行回收会降低下游稳定性。
详细 scRNA-seq+scATAC-seq 双组学数据质控相关的知识讲解可查看《单细胞 ATAC_RNA 多组学的质控》。
整合
- 质控合格的数据进行【整合】,目前提供四种整合方法,其中 CCA、Harmony 和 RPCA 会对多个样本进行批次矫正。在整合的时候,样本间的 scRNA-seq 数据和 scRNA-seq 数据进行整合,scATAC-seq 数据和 scATAC-seq 数据进行整合。选择 RNA 数据的整合方法后,ATAC 数据的整合方法会自动根据 RNA 的整合方法进行自匹配。

TIP
ATAC 的 min.cutoff 参数默认值是 5,该参数建议直接选择默认值

- 【整合】后可分别查看 RNA 数据和 ATAC 数据的整合情况,可调整整合参数重新整合。建议用户尝试多种整合方法,选择更合适的方法进行后续分析。

TIP
整合方法选择:
- 批次效应较弱:merge 直接合并,避免过度校正。
- 中等批次:Harmony,通常同平台产出的数据间的批次用 Harmony 就可有效矫正,CCA/RPCA 可能会过度矫正。
- 批次显著或异构明显:CCA/RPCA 更稳健。
TIP
效果评估三准则:
- 批次混合度:同一细胞类型在 UMAP/t-SNE (
检查 RNA 整合效果)、ATAC UMAP/ATAC t-SNE (检查 ATAC 整合效果) 中跨样本均匀混合。 - 生物信号保留:经典 marker 梯度与分群边界清晰,差异/富集结果符合预期。
- 过度/欠校正告警:过度校正会抹平差异,欠校正会出现“按批次聚类”。
聚类
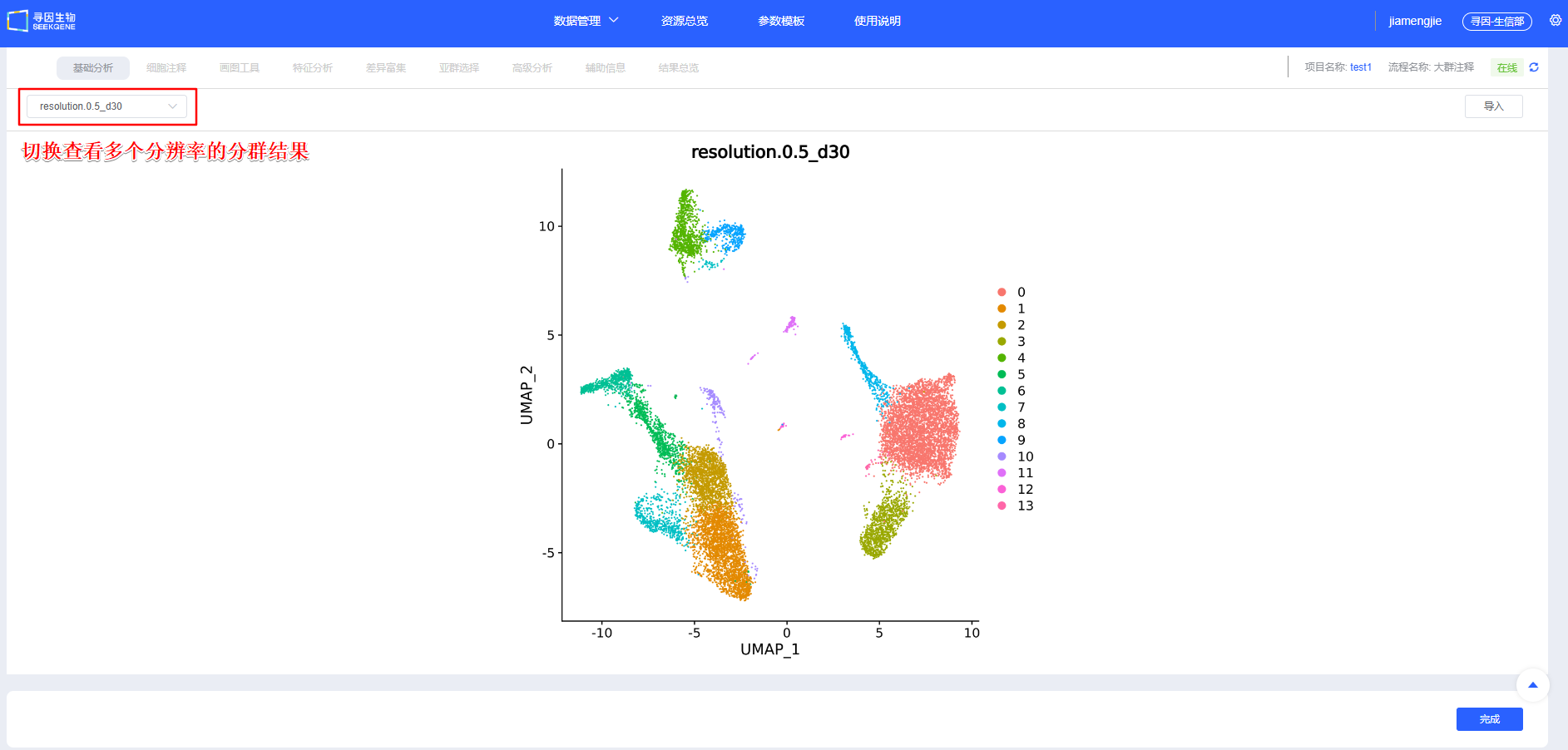
【整合】确认后进行【聚类】,可新建选择多个分辨率进行聚类,scRNA-seq 和 scATAC-seq 双组学数据会进行三种聚类:基于 ATAC 数据进行聚类、基于 RNA 数据进行聚类、联合 RNA 和 ATAC 数据进行 wnn 聚类。后续模块也可新增分辨率进行聚类。

TIP
聚类调参
- RNA 数据的拐点法:以 PCA 肘部拐点作为 dims 起点,细胞量越大适当增加。
- ATAC 数据的 lsi 的第一个 dim 通常是技术噪音,需要从第 2 个 lsi 开始选择。 dims 过低会遗漏关键异质性,过高易过聚类与放大噪音。请结合拐点与重现性优选。
详细 scRNA-seq+scATAC-seq 双组学数据降维聚类相关的知识讲解可查看《单细胞 ATAC_RNA 多组学的降维聚类》。
分析完成
【聚类】后无需调整则点击【完成】,跳转“细胞注释”模块,正式开始进行单细胞相关分析。该步会耗费一定时间,请用户耐心等待。