Banksy 空间聚类分析
前言
IMPORTANT
Banksy 是一种专门用于空间转录组学数据的聚类分析方法,通过融合细胞表达特征与空间邻域信息,显著提升空间域识别和细胞类型判别的准确性。该方法能够:
- 有效处理空间转录组数据中的噪声和稀疏性
- 识别具有相似空间微环境的细胞亚群
- 发现组织中的空间功能域和细胞间相互作用模式
- 适用于多种空间转录组技术平台(如 10x Visium、Slide-seq、MERFISH、CosMX 等)
在空间转录组学研究中,传统的聚类方法往往忽略了细胞的空间位置信息,导致无法准确识别空间相关的细胞亚群。Banksy 通过构建空间邻域特征矩阵,将每个细胞的表达特征与其周围细胞的表达模式相结合,从而更好地捕获空间组织结构。
Banksy 的核心功能
- 空间邻域特征计算:基于空间坐标构建邻域网络,计算邻域均值和梯度特征
- 多尺度聚类分析:支持多种聚类算法(Leiden、Louvain、K-means、Mclust)
- 参数优化:通过λ参数平衡表达特征与空间信息的重要性
- 可视化展示:提供空间聚类分布图和 UMAP 降维可视化
本篇文档旨在为空间转录组学研究者提供一份详尽的 Banksy 技术指南,内容涵盖其基本原理、在 SeekSoul Online 云平台上的操作方法、结果解读、实战案例及常见问题,帮助您快速掌握并应用该工具。
Banksy 理论基础
核心原理
Banksy 的核心思想是:通过融合细胞表达特征与空间邻域信息,构建增强的特征矩阵用于聚类分析。这一过程可以概括为以下几个主要步骤:
- 空间邻域构建:根据细胞的空间坐标确定空间邻居关系
- 邻域特征计算:计算每个细胞邻域的均值和梯度特征
- 特征矩阵融合:将原始表达特征与空间邻域特征按权重融合
- 聚类分析:基于融合后的特征矩阵进行聚类分析
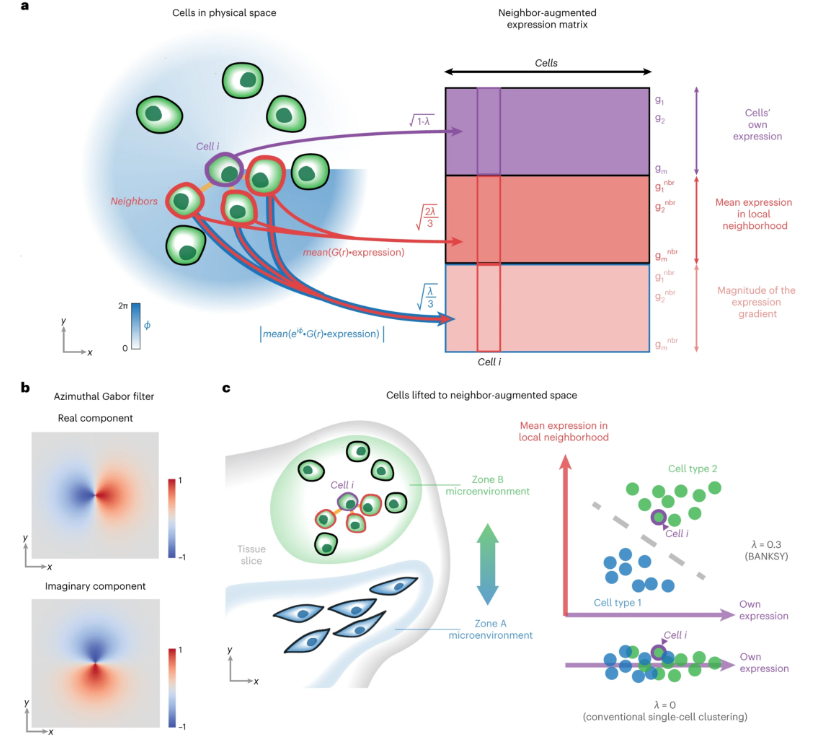
关键算法详解
空间邻域构建
- 原理:基于细胞或斑点的空间坐标,使用 k 近邻方法构建空间邻域网络
- 方法:对每个细胞,找到其 k 个最近的空间邻居
- 参数:k_geom 控制邻域大小,通常设置为 15 和 30
- 优势:能够准确反映细胞在组织中的真实空间关系
邻域特征计算
- 邻域均值(Neighborhood Mean):计算每个细胞邻域内基因的平均表达水平
- 邻域梯度(Neighborhood Gradient):计算每个细胞邻域内基因表达的空间梯度
- 特征融合:将原始表达特征与邻域特征按λ权重进行线性组合
Banksy 矩阵构建
- 原始特征矩阵(M):细胞的基因表达矩阵
- 邻域均值矩阵(G_mean):邻域内基因平均表达矩阵
- 邻域梯度矩阵(G_grad):邻域内基因表达梯度矩阵
- 融合矩阵:B = M + λ × (G_mean + G_grad)
聚类分析
- 降维处理:对融合后的特征矩阵进行 PCA 降维
- 聚类算法:支持多种聚类方法,包括 Leiden、Louvain、K-means、Mclust
- 参数优化:通过调整λ和分辨率参数优化聚类效果
生物学意义
空间域识别
- 功能:识别组织中具有相似空间微环境的功能区域
- 应用:理解组织发育、疾病进展等生物学过程
细胞亚型发现
- 功能:发现受空间微环境影响的细胞亚型
- 应用:研究细胞分化和功能特化过程
空间相互作用研究
- 功能:揭示细胞间通过空间邻近性进行的相互作用
- 应用:研究细胞通讯和调控网络
云平台操作指南
在云平台上,Banksy 分析流程被设计得直观易用。您无需编写代码,只需通过参数配置界面即可完成分析。
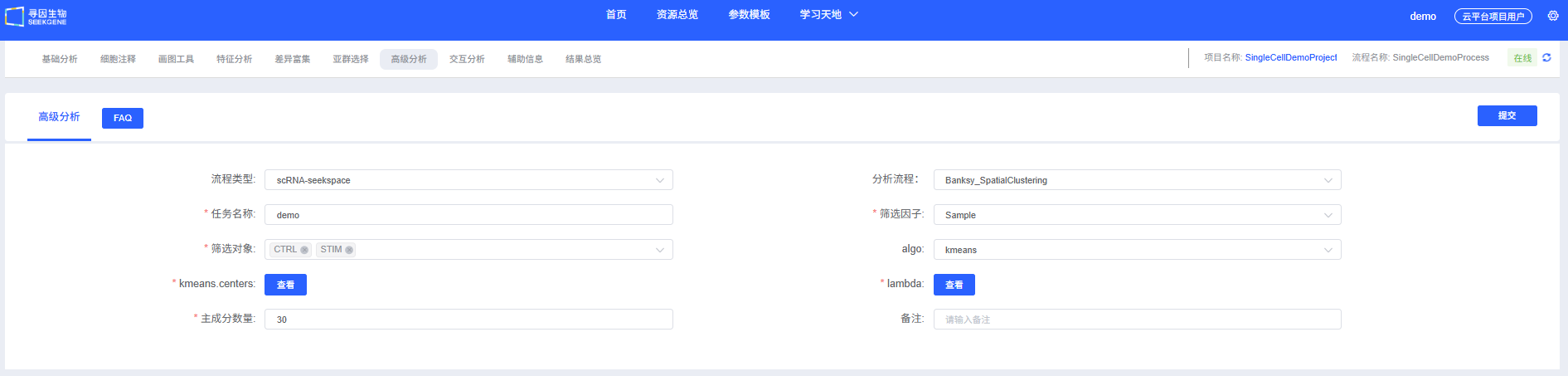
分析前的准备
IMPORTANT
Banksy 分析的成功与否,很大程度上取决于输入数据的质量和空间信息的准确性。在开始分析前,请务必确认:
- 数据已完成预处理:您的空间转录组数据已经过标准的质控、降维、聚类和细胞类型注释。
- 空间坐标信息完整:确保每个细胞或斑点都有准确的空间坐标信息。
- 数据格式正确:确保输入数据为标准的 Seurat 对象或 SpatialExperiment 对象。
参数详解
下表详细列出了云平台 Banksy 分析模块的主要参数及其说明。
| 界面参数 | 说明 | 推荐值 | 注意事项 |
|---|---|---|---|
| 任务名称 | 本次分析的任务名称,需以英文字母开头,可包含英文字母、数字、下划线和中文。 | 如:Banksy_analysis_001 | 建议使用有意义的命名便于后续管理 |
| 筛选因子 | 需要进行 Banksy 空间聚类分析在 rds 的 meta 列存储列名,必填。 | orig.ident | 确保该列包含样本信息 |
| 筛选对象 | 需要进行 Banksy 空间聚类分析的在 rds 的 meta 列样本名,必填。 | 如:sample_001 | 确保样本名与筛选因子列中的值一致 |
| 聚类算法 | Banksy 空间聚类分析的聚类方法选择,leiden、louvain、kmeans 和 mclust 可选,必填。 | leiden | 推荐首选 leiden 算法 |
| 分辨率 | Banksy 空间聚类分析的 cluster 数,当 algo 选择 leiden 或 louvain 时使用。resolution 越大,cluster 数越多。 | 0.4,0.8 | 建议尝试多个值,选择生物学意义最合理的结果 |
| K-means 聚类数 | 指定 Banksy 空间聚类分析的 cluster 数,当 algo 选择 kmeans 时使用。填写指定聚类的 cluster 数。 | 5,10,15 | 需要预先估计聚类数目 |
| Mclust 聚类数 | 指定 Banksy 空间聚类分析的 cluster 数,当 algo 选择 mclust 时使用。填写指定聚类的 cluster 数。 | 3,5,7 | 适用于数据分布复杂的情况 |
| λ权重 | Banksy 空间聚类分析表达和空间位置对聚类效果影响的权重,多个值用逗号分隔。 | 0.6,0.8 | 建议从 0.2 开始尝试,根据数据特点调整 |
| 主成分数量 | 默认是 30,用于 PCA 降维。 | 30 | 可根据数据规模调整,建议 20-50 之间 |
| 备注 | 自定义备注信息。 | - | 记录分析目的和特殊要求 |
重要注意事项
CAUTION
- 空间坐标要求:确保空间坐标信息准确且完整,缺失或错误的坐标信息会导致分析失败。
- λ参数选择:λ参数控制空间信息的重要性,建议从 0.2 开始尝试,根据数据特点调整。
- 聚类算法选择:不同聚类算法适用于不同的数据特点,建议先尝试 Leiden 算法。
操作流程
- 进入分析模块:在云平台导航至"高级分析"模块,选择"Banksy_SpatialClustering"。
- 创建新任务:为您的分析任务命名,并选择要分析的样本或项目。
- 配置参数:根据上述指南,选择合适的聚类算法、λ权重等参数。
- 提交任务:确认参数无误后,点击"提交"按钮,等待分析完成。
- 下载与查看:分析结束后,在任务列表中下载并查看生成的分析报告和结果文件。
结果解读
Banksy 的分析报告包含丰富的图表和数据文件,以下是对核心结果的详细解读。
结果文件列表
| 文件名 | 内容说明 | 文件格式 |
|---|---|---|
*_banksy_colData.csv | 包含所有细胞聚类标签的元数据文件,包括 barcode、sizeFactor、不同参数组合的聚类标签和空间坐标 | CSV |
*_clust_M1_lam*_k50_res*_spatial_cluster_plot.png/pdf | 不同λ和分辨率下的空间聚类分布图,展示细胞在空间中的聚类结果 | PNG/PDF |
*_clust_M1_lam*_k50_res*_umap_plot.png/pdf | 不同λ和分辨率下的 UMAP 降维可视化图,展示细胞在降维空间中的分布 | PNG/PDF |
NOTE
文件名中的参数含义:
M1:表示使用邻域均值特征lam*:λ参数值(如 lam0.6 表示λ=0.6)k50:表示使用 50 个空间邻居res*:分辨率参数值(如 res0.4 表示分辨率=0.4)
空间聚类分布图解读
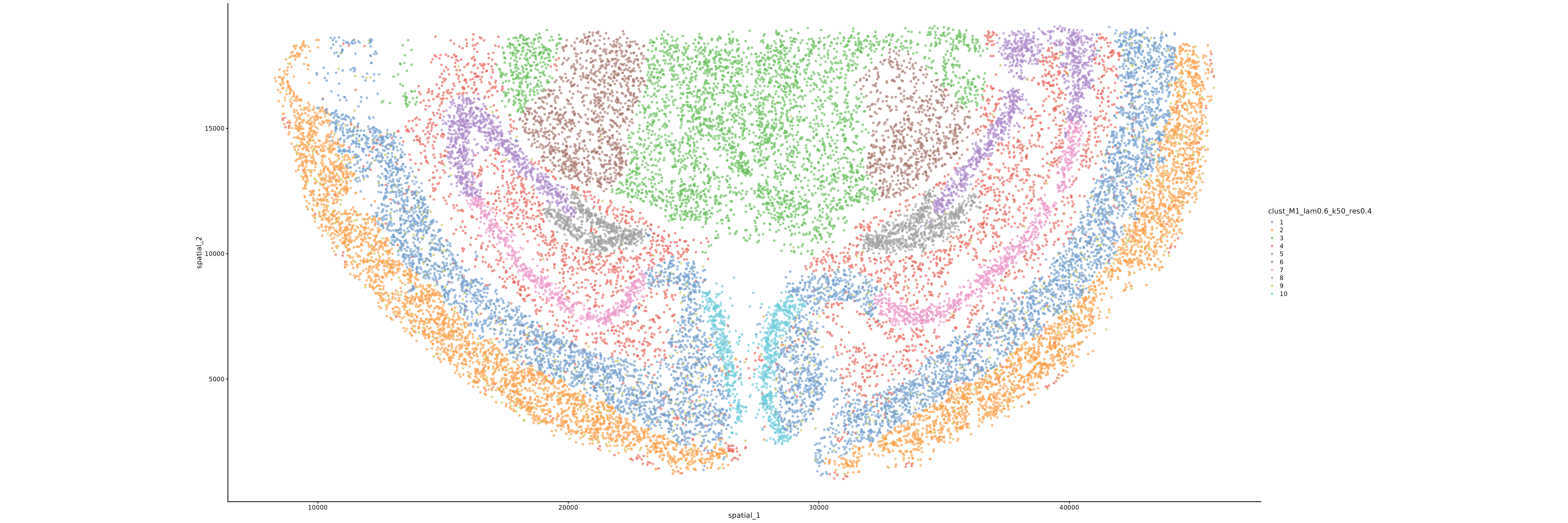
图表结构
- 横轴:细胞的空间 X 坐标
- 纵轴:细胞的空间 Y 坐标
- 颜色:不同的聚类标签,每个颜色代表一个空间域
解读要点
- 空间连续性:良好的聚类结果应该显示出空间上的连续性,相邻的细胞倾向于属于同一聚类
- 边界清晰:不同聚类之间应该有相对清晰的边界
- 生物学意义:聚类结果应该与已知的组织结构或功能区域相对应
UMAP 降维可视化解读
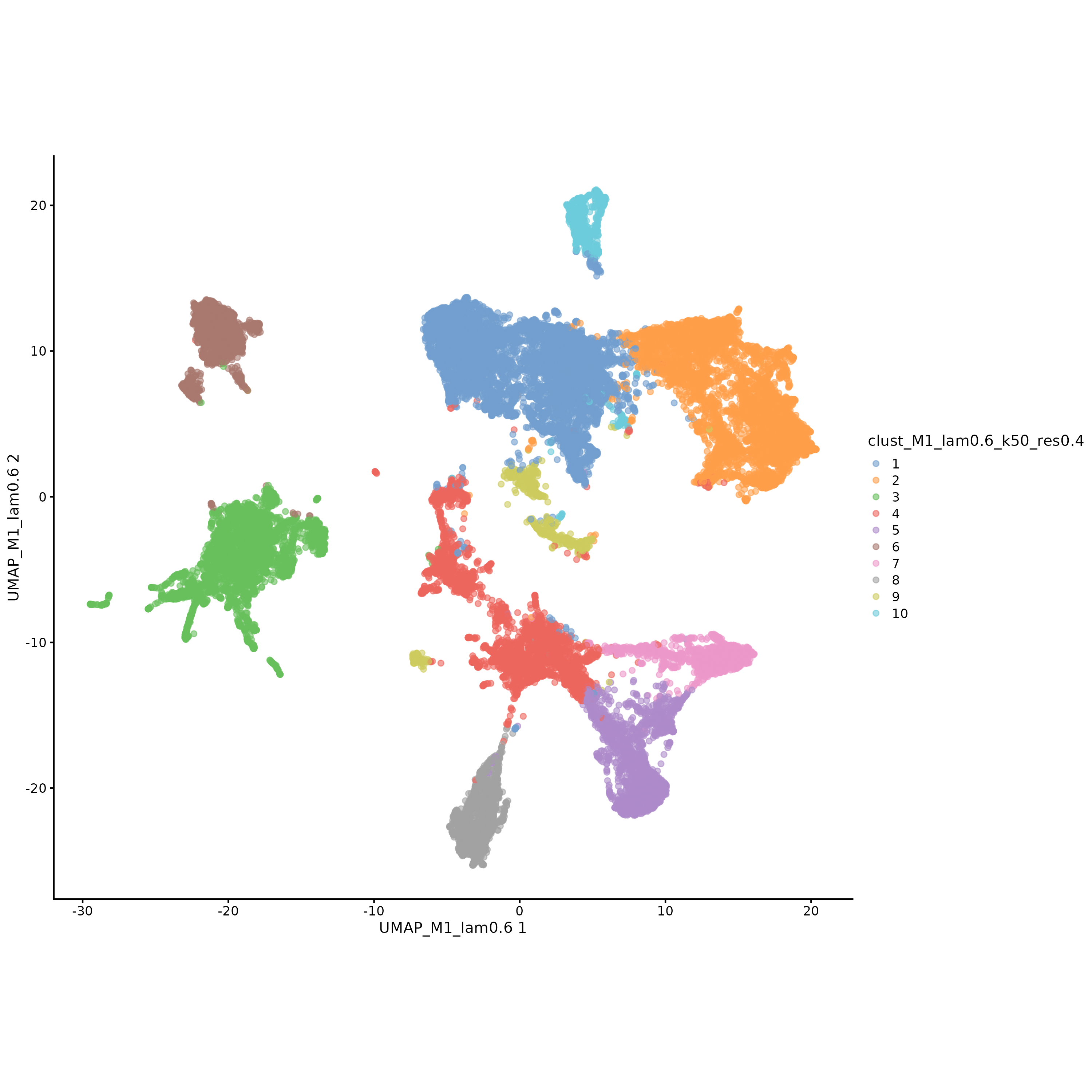
图表结构
- 横轴:UMAP 第一主成分
- 纵轴:UMAP 第二主成分
- 颜色:不同的聚类标签
解读要点
- 聚类分离度:不同聚类的细胞在 UMAP 空间中应该相对分离
- 聚类紧密度:同一聚类的细胞在 UMAP 空间中应该相对聚集
- 异常值识别:可以识别出可能的异常细胞或边界细胞
元数据文件解读
元数据文件(*_banksy_colData.csv)包含以下信息:
| 列名 | 数据类型 | 说明 | 示例值 |
|---|---|---|---|
| barcode | 字符串 | 细胞条形码,用于唯一标识每个细胞 | "AAGGAATGCTGATTCGTTTCTGCGCTC" |
| sizeFactor | 数值 | 细胞大小因子,用于归一化处理 | 0.168491435461364 |
| clust_M1_lam_k50_res** | 字符串 | 不同参数组合下的聚类标签,数字表示聚类 ID | "2", "11", "4", "12" |
| spatial_1 | 数值 | 细胞的空间 X 坐标 | 42357 |
| spatial_2 | 数值 | 细胞的空间 Y 坐标 | 10507 |
聚类标签解读
- 聚类 ID:数字表示不同的空间域,相同数字的细胞属于同一聚类
- 参数组合:不同λ和分辨率参数会产生不同的聚类结果
- 空间坐标:用于可视化细胞在组织中的空间位置
TIP
可以通过比较不同参数组合的聚类结果,选择生物学意义最合理的参数设置。
应用案例
案例一:Banksy 算法原理演示
- 数据来源:Banksy 官方 GitHub README 示例
- 背景:使用 Banksy 官方提供的小鼠海马体空间转录组数据,演示 Banksy 算法的基本工作原理。
- 分析策略:比较不同λ值(0, 0.2)对聚类结果的影响,展示空间信息的重要性。
- 核心发现:
- λ=0 时(非空间聚类):聚类结果主要基于基因表达相似性,空间连续性较差。
- λ=0.2 时(Banksy 聚类):结合空间邻域信息后,聚类结果在空间上更加连续和一致。
- Banksy 能够有效平衡基因表达特征与空间位置信息,提高空间聚类的生物学意义。
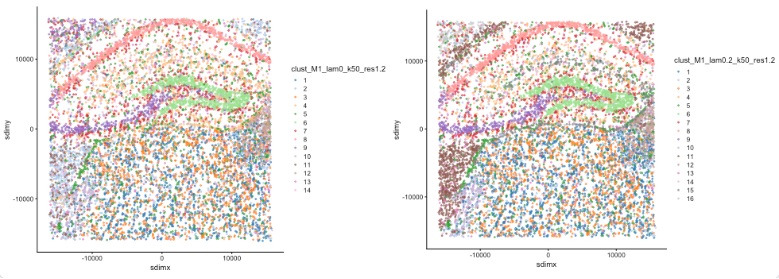
图:Banksy 算法原理演示。左图显示非空间聚类(λ=0),右图显示 Banksy 空间聚类(λ=0.2)。通过对比可以看出,Banksy 能够更好地识别具有空间连续性的功能区域。
案例二:Banksy 聚类分离效果展示
- 数据来源:Banksy 官方 GitHub README 示例
- 背景:使用 Banksy 官方提供的小鼠海马体空间转录组数据,展示不同λ参数对聚类分离效果的影响。
- 分析策略:比较λ=0 和λ=0.2 两种参数设置下的聚类结果,展示每个聚类的空间分布模式。
- 核心发现:
- λ=0 时:识别出 14 个聚类,部分聚类空间分布较为分散,边界不够清晰。
- λ=0.2 时:识别出 16 个聚类,空间分布更加集中,边界更加清晰。
- 通过增加空间权重,Banksy 能够识别出更精细的空间结构,提高聚类的生物学意义。
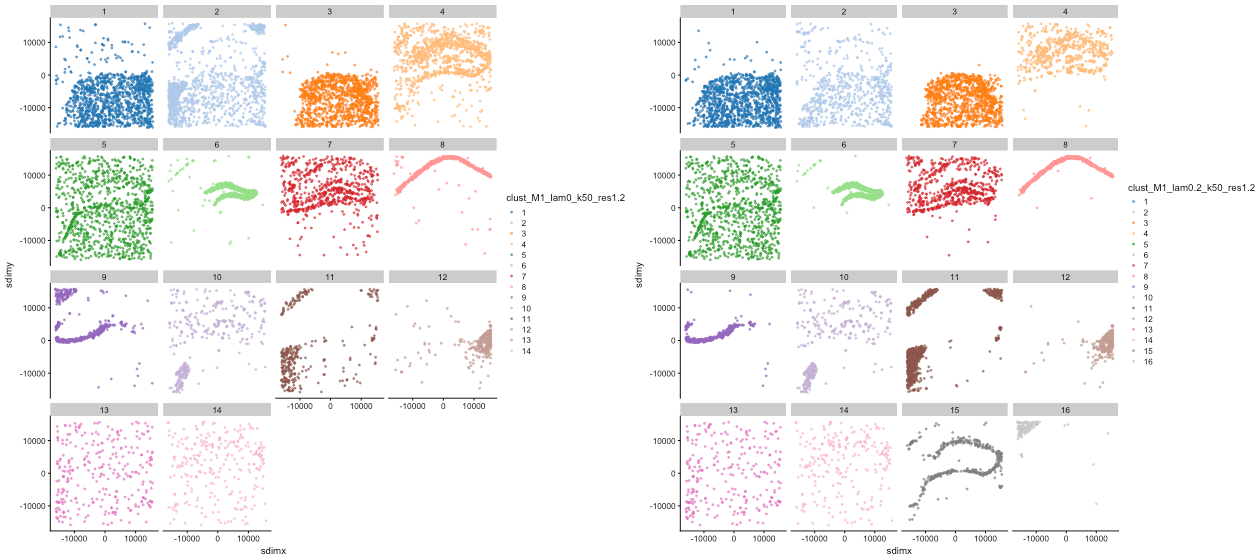
图:Banksy 聚类分离效果展示。左图显示λ=0 的聚类结果(14 个聚类),右图显示λ=0.2 的聚类结果(16 个聚类)。通过对比可以看出,增加空间权重后能够识别出更精细的空间结构。
注意事项与最佳实践
WARNING
避免过度解读:Banksy 结果是基于空间邻域特征的计算推断,不等于真实的生物学相互作用。任何关键发现都需要后续的生物学实验来证实。
CAUTION
数据质量至关重要:Banksy 分析对空间坐标信息的准确性要求很高,低质量的空间数据可能导致假阳性结果。务必确保空间坐标信息准确且完整。
TIP
参数优化建议:
- λ参数:建议从 0.2 开始尝试,根据数据特点调整
- 分辨率参数:建议尝试多个值,选择生物学意义最合理的结果
- 聚类算法:建议先尝试 Leiden 算法,效果不佳时再尝试其他算法
NOTE
结果受参数影响:Banksy 分析结果会受到λ参数、分辨率参数以及聚类算法选择的影响。如果初步结果不理想,可以尝试调整这些参数重新进行分析。
常见问题解答 (FAQ)
Q1: Banksy 分析需要多长时间?
A: 分析时间取决于数据规模和计算资源配置。一般来说:
- 小数据集(1,000-5,000 细胞):10-30 分钟
- 中等数据集(5,000-20,000 细胞):30 分钟-2 小时
- 大数据集(>20,000 细胞):2-8 小时或更长
Q2: λ参数如何选择?
A: λ参数控制空间信息的重要性:
- λ = 0:仅使用表达特征,相当于传统聚类
- λ = 0.2:推荐起始值,平衡表达与空间信息
- λ = 0.5-1.0:更重视空间信息,适用于空间结构明显的数据
- λ > 1.0:过度重视空间信息,可能导致过度平滑
Q3: 如何选择合适的聚类算法?
A: 不同聚类算法适用于不同场景:
- Leiden:推荐首选,适用于大多数数据
- Louvain:与 Leiden 类似,但计算速度可能更快
- K-means:适用于已知聚类数目的情况
- Mclust:适用于数据分布复杂的情况
Q4: 如何验证 Banksy 分析结果的可靠性?
A: 可通过以下方式验证结果可靠性:
- 生物学验证:结合已知的组织结构和功能区域验证聚类结果
- 参数敏感性:测试不同参数组合的稳定性
- 交叉验证:使用不同的聚类算法验证结果一致性
- 功能富集:对每个聚类进行功能富集分析验证生物学意义
Q5: Banksy 适用于哪些空间转录组技术?
A: Banksy 适用于多种空间转录组技术:
- 10x Visium:推荐λ = 0.2-0.5
- Slide-seq:推荐λ = 0.1-0.3
- MERFISH:推荐λ = 0.3-0.6
- CosMX:推荐λ = 0.2-0.4
- SeekSpace:推荐λ = 0.6-0.8
- 其他技术:根据空间分辨率调整λ参数
Q6: 如何选择合适的λ参数?
A: λ参数的选择需要考虑以下因素:
- 空间分辨率:高分辨率数据可以使用较小的λ值
- 组织类型:结构复杂的组织可以使用较大的λ值
- 分析目标:如果更关注空间连续性,使用较大的λ值
- 建议策略:从 0.2 开始尝试,逐步增加到 1.0,选择生物学意义最合理的结果
Q7: 聚类结果不理想怎么办?
A: 可以尝试以下优化策略:
- 调整λ参数:尝试不同的λ值组合
- 调整分辨率:尝试不同的分辨率参数
- 更换聚类算法:尝试 Leiden、Louvain、K-means 等不同算法
- 检查数据质量:确保空间坐标信息准确完整
- 预处理优化:检查数据预处理步骤是否合适
参考资料
[1] CHUNG J, et al. Banksy: spatial clustering with spatial omics data[J]. Nature Genetics, 2024, 56: 74–84.
[4] CHUNG J, et al. Spatial transcriptomics: technologies, applications and experimental considerations[J]. Genomics, 2021, 113: 1-15.
[5] MOSES L, PACHTER L. Museum of spatial transcriptomics[J]. Nature Methods, 2022, 19: 534–546.
