COMMOT 分析
前言
IMPORTANT
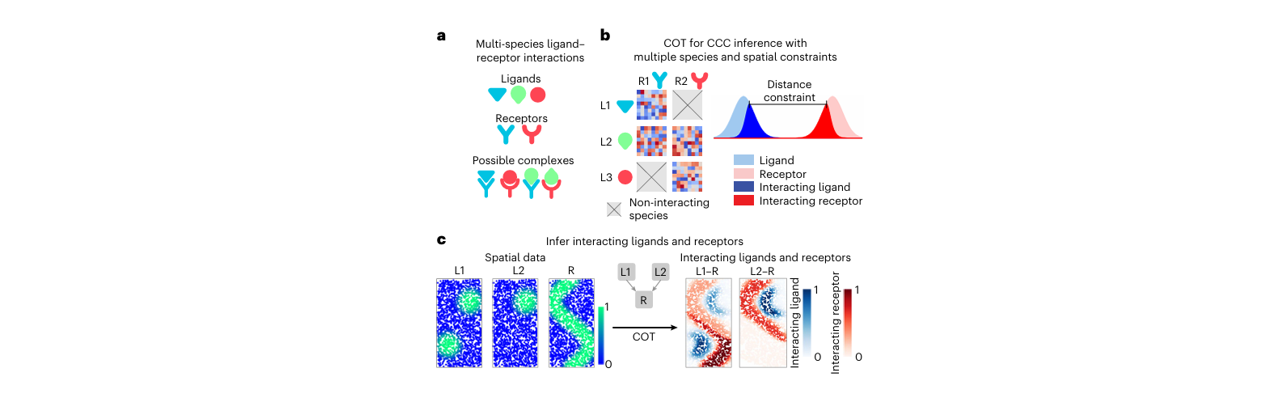
COMMOT (COMMunication analysis by Optimal Transport) 是一个基于最优传输理论的空间细胞通讯推断工具,专门用于分析空间转录组数据中的细胞间通讯。COMMOT 通过集合最优传输 (Collective Optimal Transport) 算法,在考虑配体-受体竞争关系和空间距离约束的前提下,准确推断空间邻近细胞间的通讯模式。
在空间转录组学研究中,细胞通讯不仅依赖于配体和受体的表达,空间位置关系和分子扩散过程也对细胞间相互作用产生重要影响。COMMOT 通过整合基因表达数据、配体-受体数据库和空间坐标信息,利用最优传输理论对配体分子从发送细胞到接收细胞的"传输"过程进行建模,从而识别具有生物学意义的空间细胞通讯网络。
COMMOT 的核心功能
- 集合最优传输算法:同时考虑多个配体-受体对之间的竞争关系
- 空间距离约束:基于细胞间的实际空间距离建模分子扩散
- 信号方向推断:识别信号从发送细胞到接收细胞的传递方向
- 下游基因预测:推断受特定信号通路调控的靶基因
- 丰富的可视化:提供空间图、流向图、和弦图等多种可视化方式
- 多数据库支持:支持 CellChat 和 CellPhoneDB 等主流配体-受体数据库
本篇文档旨在为空间转录组学研究者提供一份详尽的 COMMOT 技术指南,内容涵盖其基本原理、在 SeekSoul Online 云平台上的操作方法、结果解读、实战案例及常见问题,帮助您快速掌握并应用该工具。
COMMOT 理论基础
COMMOT 的核心思想是:将细胞间通讯建模为一个最优传输问题,配体分子从发送细胞 (source) 传输到接收细胞 (target),传输成本取决于空间距离,传输量取决于配体和受体的表达量以及它们之间的竞争关系。这一过程可以概括为以下几个主要步骤:
- 数据预处理:提取表达矩阵和空间坐标信息
- 配体-受体对筛选:从数据库中筛选在数据中表达的配体-受体对
- 空间距离矩阵计算:计算所有细胞对之间的欧氏距离
- 集合最优传输:利用集合最优传输算法求解配体-受体通讯网络
- 信号方向推断:基于传输流向识别信号传递方向
- 下游分析:使用机器学习模型预测信号调控的靶基因
云平台操作指南
在云平台上,COMMOT 分析流程被设计得直观易用。您无需编写代码,只需通过参数配置界面即可完成分析。
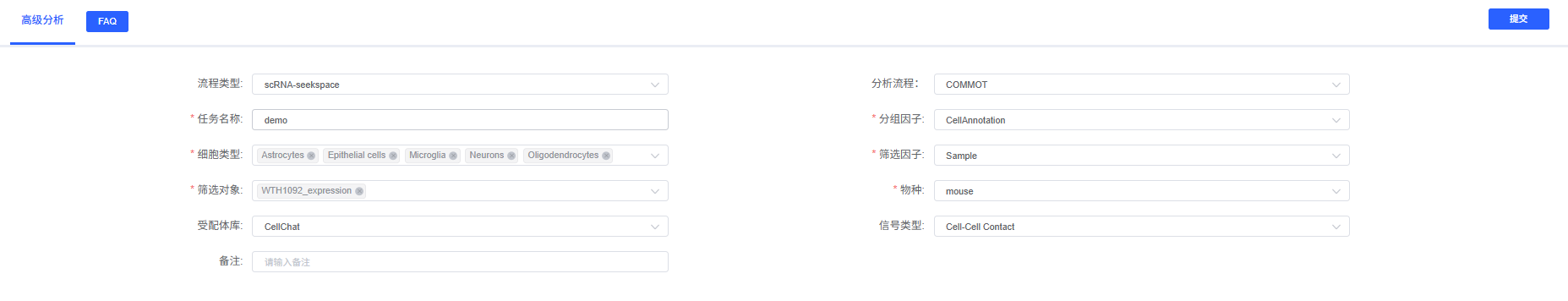
参数设置说明
在 SeekSoul Online 云平台的"高级分析"模块中选择"COMMOT",需要配置以下参数:
基础参数
任务名称
- 说明:本次分析的任务名称
- 格式要求:需以英文字母开头,可包含英文字母、数字、下划线和中文
- 示例:
COMMOT_tumor_analysis
分组因子
- 说明:meta 数据中的细胞类型列名
- 示例:
CellAnnotation、celltype - 用途:用于定义细胞类型,分析不同细胞类型间的通讯
细胞类型
- 说明:基于分组因子列选择要分析的细胞类型
- 格式:可多选,用逗号分隔
- 示例:
pPCM,dPCM或T_cells,B_cells,Macrophage
样本筛选参数
筛选因子
- 说明:meta 数据中的样本列名
- 示例:
Sample、GD.N_A
筛选对象
- 说明:基于筛选因子列选择要分析的样本名称
- 示例:
A15、sample1 - 注意:COMMOT 一次分析一个样本,如需比较多个样本需分别提交任务
配体-受体数据库参数
物种
- 选项:
human(人)或mouse(小鼠) - 说明:选择样本对应的物种
受配体库
- 选项:
CellChat:来自 CellChat 数据库的配体-受体对(~2000 对)CellPhoneDB_v4.0:来自 CellPhoneDB v4.0 数据库的配体-受体对(~3000 对)
信号类型
- 选项:
Secreted Signaling:分泌型信号(可扩散的分泌蛋白)Cell-Cell Contact:细胞-细胞接触信号(需直接接触)ECM-Receptor:细胞外基质-受体信号(仅 CellChat 支持)None:使用所有类型的配体-受体对
备注
- 说明:自定义备注信息(选填)
报告结果解读
COMMOT 分析完成后,会生成一份包含多种可视化结果和数据文件的 HTML 报告。以下详细解读各部分结果的含义和解读要点。
配体-受体通讯网络
总体通讯强度矩阵
图片示例:total-total_cluster.png
这是一个热图,展示不同细胞类型对之间的总体通讯强度。
解读要点:
- 行:信号发送细胞类型 (Sender)
- 列:信号接收细胞类型 (Receiver)
- 颜色深度:通讯强度(值越大,颜色越深)
- 对角线:同类型细胞间的自分泌信号
- 非对角线:不同类型细胞间的旁分泌信号
信号发送-接收特征图
发送细胞与接收细胞的空间分布
图片示例:sender_receiver_signature.png
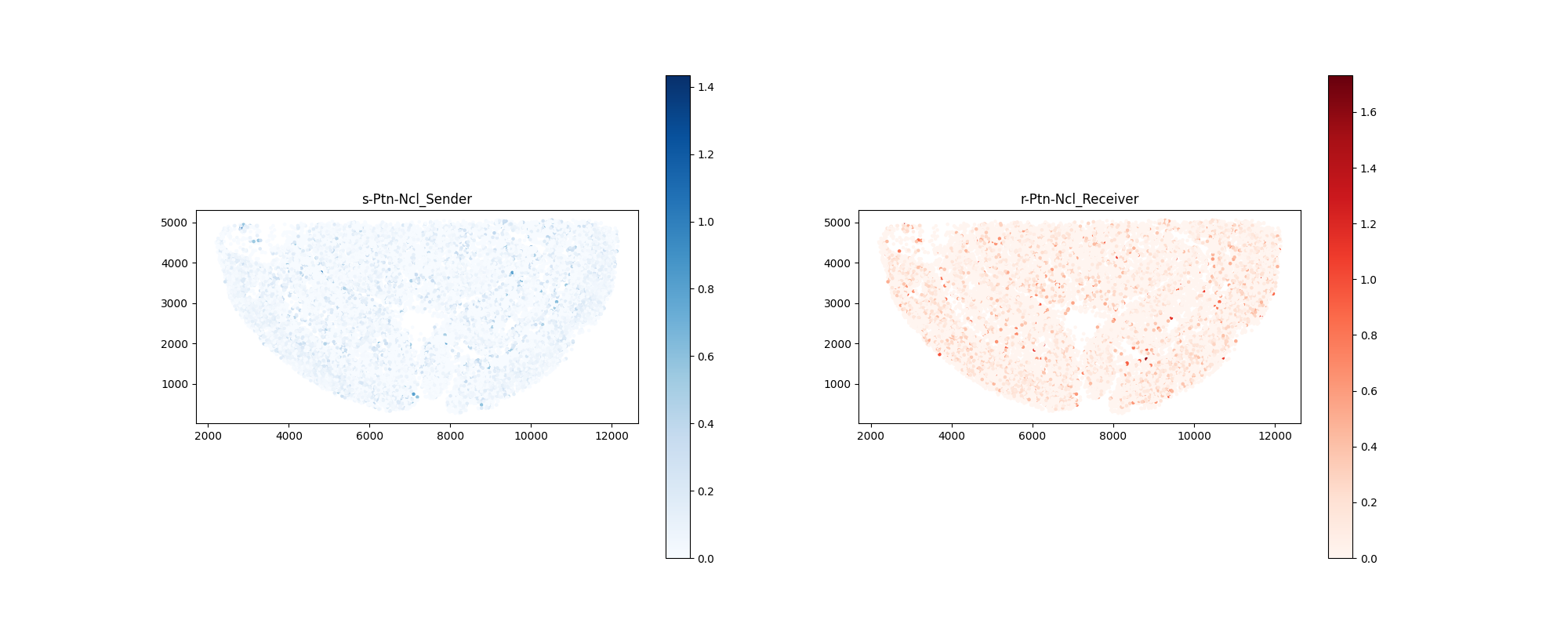
解读要点:
- 左图:发送细胞的空间分布和信号发送强度(蓝色为高强度)
- 右图:接收细胞的空间分布和信号接收强度(红色为高强度)
生物学意义:
- 可视化信号从哪里发出、到哪里接收
- 识别信号梯度和空间模式
- 发现信号传播的空间规律
信号流向图 (Signal Flow)
总体信号流向
图片示例:total_signature_stream.png

解读要点:
- 点:细胞的空间位置
- 箭头:信号传播方向
- 箭头长度/粗细:信号强度
关键观察点:
- 信号源:箭头起始密集的区域
- 信号汇:箭头指向密集的区域
- 信号路径:箭头连续传递的路线
细胞类型间通讯网络
和弦图 (Chord Diagram)
图片示例:total_chord_plot.png
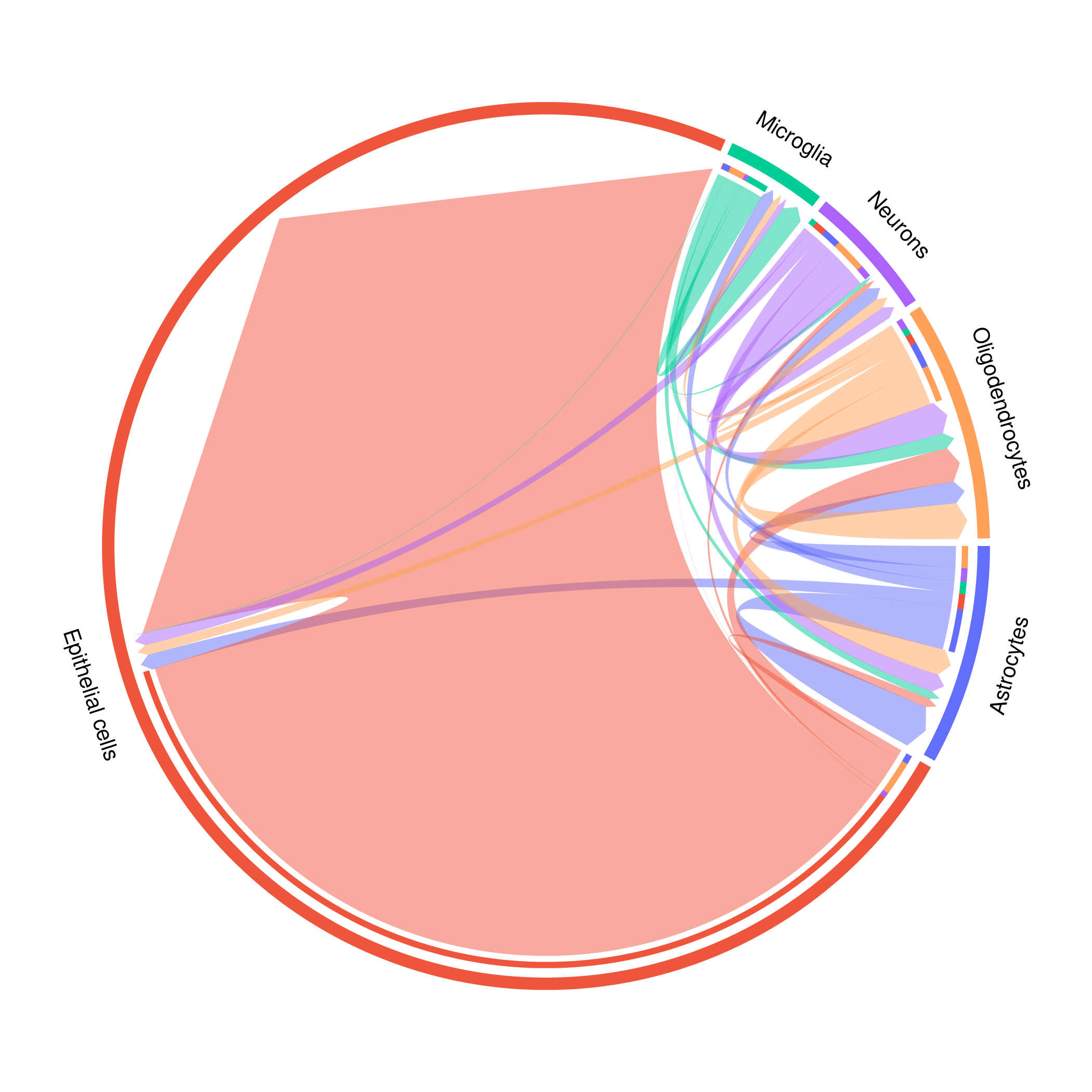
解读要点:
- 圆环扇形:不同的细胞类型
- 扇形大小:该细胞类型参与通讯的总强度
- 连线:细胞类型对之间的通讯
- 连线宽度:通讯强度
分析策略:
- 识别枢纽细胞:扇形大、连线多的细胞类型
- 关注强通讯:宽连线代表的细胞类型对
- 发现通讯模式:如单向通讯、双向通讯、多对一等
定量数据文件
COMMOT 输出的 h5ad 文件包含:
commot-sum:总通讯强度矩阵commot-sum-sender:各细胞作为发送者的强度commot-sum-receiver:各细胞作为接收者的强度- 特定配体-受体对的通讯矩阵
结果解读的最佳实践
TIP
结果解读建议:
- 从整体到局部:先看总体通讯模式,再看具体配体-受体对
- 结合生物学知识:将发现的通讯模式与已知的生物学功能联系
- 空间验证:关注空间上邻近的细胞是否确实存在强通讯
- 多角度分析:结合不同可视化方式综合判断
应用案例
新生儿人类表皮发育中的细胞通讯分析
- 文献:Cang Z., Zhao Y., Almet A.A., et al. Screening cell–cell communication in spatial transcriptomics via collective optimal transport. Nature Methods 20, 218–228 (2023). DOI: 10.1038/s41592-022-01728-4
- 研究背景:人类表皮稳态维持需要基底层干细胞的增殖和向上层的分化之间的精确平衡,这一过程受到细胞间通讯的调控。该研究使用 COMMOT 分析新生儿人类表皮的空间转录组数据,解析表皮稳态维持过程中的细胞通讯网络。
- 数据类型:scRNA-seq + 空间转录组数据(通过 SpaOTsc 算法整合 scRNA-seq 数据与免疫荧光染色图像数字化的空间信息)
- 样本:新生儿人类表皮 (neonatal human epidermis)
- 参考文献:PMC9911355
COMMOT 分析结果
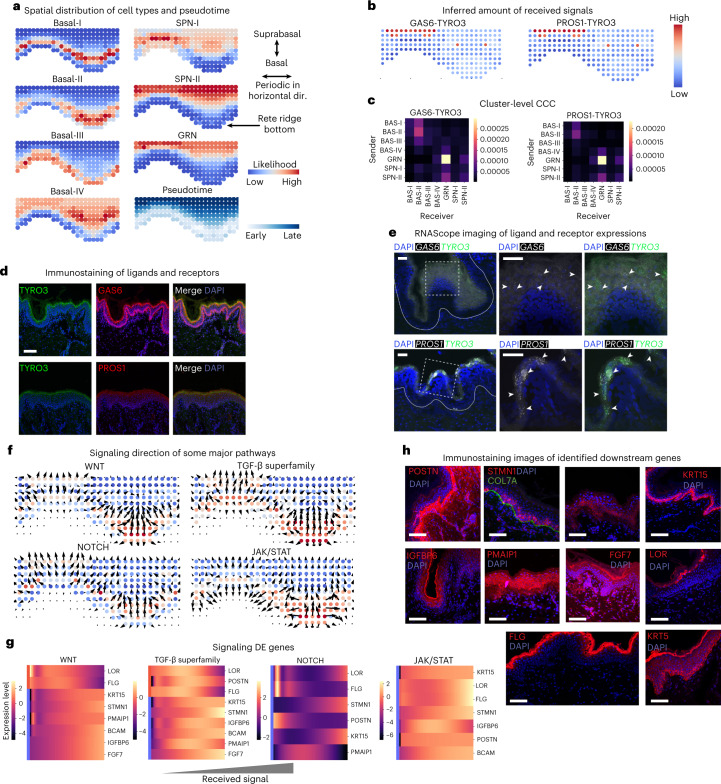
图 2:COMMOT 分析人类表皮发育中的细胞通讯。a. 细胞类型的空间预测分布和伪时间在空间中的投影,展示从基底层到上层表皮的发育路径(GRN:颗粒层细胞;SPN:棘层细胞)。b. 细胞水平上两个配体-受体对(GAS6-TYRO3 和 PROS1-TYRO3)的接收信号量推断。c. 细胞簇水平上的推断信号量。d. GAS6、TYRO3 和 PROS1 蛋白的免疫荧光染色验证。e. 荧光原位杂交 (FISH) 验证 GAS6-TYRO3(上排)和 PROS1-TYRO3(下排)的 RNA 表达模式,白色箭头标示颗粒层的共定位信号。f. 四个主要信号通路(WNT、TGF-β、NOTCH、JAK/STAT)的信号传播方向。g. 四个信号通路相关的差异表达基因热图。h. 免疫荧光染色验证 WNT 信号与预测基因表达的相关性。比例尺:d, e, h 为 100 μm。
核心发现
GAS6/PROS1-TYRO3 配体-受体对的验证:COMMOT 预测并通过免疫荧光和 RNA 原位杂交验证了 GAS6/PROS1-TYRO3 相互作用在颗粒层细胞中最强,参与调控细胞存活和终末分化。
四个主要信号通路的空间传播方向:COMMOT 揭示所有四个通路(WNT、TGF-β、NOTCH、JAK/STAT)都主要表现为向上传播,即从上层分化细胞向基底层干细胞传递信号,形成反馈调控机制维持表皮稳态。
信号通路的下游基因:基于 COMMOT 推断的信号强度,识别了与各通路相关的差异表达基因。WNT 信号促进基底标志物(KRT15、KRT5)上调、抑制分化标志物(LOR、FLG),验证了 WNT 促进干细胞增殖、抑制终末分化的功能。
WNT 信号下游基因的验证:通过免疫荧光验证了 COMMOT 预测的 WNT 响应基因(BCAM、POSTN、STMN1)在基底层的高表达,进一步证实了 COMMOT 对信号通路下游效应的预测能力。
注意事项与最佳实践
WARNING
避免过度解读:COMMOT 结果是基于配体-受体表达和空间距离的计算推断,不等于真实的细胞间相互作用。任何关键发现都需要后续的实验验证。
CAUTION
数据质量至关重要:
- 确保空间坐标信息准确
- 细胞类型注释需可靠
- 基因表达数据需经过适当的质控和归一化
分析前的准备
数据质控:
- 移除低质量的 spots/cells
- 进行适当的归一化(推荐使用 log-normalization)
- 确保空间坐标与表达矩阵对应
细胞类型注释:
- 使用可靠的方法进行细胞类型注释
- 避免过度细分导致某些类型细胞数过少
- 建议每种细胞类型至少有 20-30 个 cells/spots
参数选择:
- 根据研究问题选择合适的配体-受体数据库
- 根据空间分辨率选择合适的信号类型
- 对于 Visium 数据,建议使用 Secreted Signaling
结果验证建议
文献验证:查阅已有文献,确认发现的通讯是否有生物学依据
实验验证:
- 免疫荧光:验证关键配体和受体的空间表达
- 功能实验:使用抑制剂或敲除验证信号功能
跨样本验证:在多个样本中重复分析,确认发现的稳定性
常见问题解答
问题 1:COMMOT 分析失败,报错 "No valid ligand-receptor pairs found"
- 原因:数据中表达的配体-受体对太少
- 解决:
- 检查物种选择是否正确
- 尝试更换配体-受体数据库
- 检查基因表达数据的归一化是否合适
问题 2:所有细胞类型间的通讯强度都很弱
- 原因:可能是数据质量问题或细胞类型距离较远
- 解决:
- 检查数据质控步骤
- 尝试使用 "None" 信号类型包含所有配体-受体对
- 检查空间坐标是否正确
问题 3:和弦图显示某些细胞类型没有通讯
- 原因:该细胞类型可能不表达任何配体或受体,或与其他细胞距离太远
- 解决:
- 检查该细胞类型的配体-受体表达情况
- 考虑该细胞类型的生物学特性是否确实不参与通讯
问题 4:如何选择分析的细胞类型
- 建议:
- 如果细胞类型较少(<10 种),建议分析所有类型
- 如果细胞类型较多,建议根据研究目的选择感兴趣的类型
- 避免选择细胞数量过少的稀有细胞类型
参考资料
- GitHub:https://github.com/zcang/COMMOT
- [1] CANG Z, et al. Screening cell-cell communication in spatial transcriptomics via collective optimal transport[J]. Nature Methods, 2023, 20: 418–428.
总结
COMMOT 是一个强大的空间细胞通讯分析工具,通过集合最优传输算法,在考虑空间距离和配体-受体竞争的前提下,准确推断细胞间通讯网络。SeekSoul Online 云平台集成了 COMMOT 分析流程,使得研究者无需复杂的编程即可完成分析。
