scVelo 拟时序分析
前言
TIP
scVelo 是基于 RNA 速度(RNA Velocity)概念的单细胞轨迹推断工具,它通过分析新生(未剪切)和成熟(已剪切)的 mRNA 比例,来预测单个细胞未来的转录状态,从而揭示细胞动态过程的方向性和速率。与传统的拟时序分析相比,scVelo 提供了更具生物学意义的矢量信息,能够直接推断细胞的命运走向。
在单细胞研究中,理解细胞状态转变的动态调控是核心挑战之一。传统的轨迹推断方法通常基于细胞间的转录相似性来排序,但无法明确指明转变的方向。scVelo 通过引入“RNA 速度”这一概念,巧妙地利用了基因转录过程中的时间信息,为我们描绘了一幅细胞状态变化的“矢量场图”,使得我们可以预测细胞在不久的将来会向哪个方向分化或转变。
scVelo 的核心功能
- RNA 速度计算:基于剪切和未剪切的 reads 计数,估计每个基因在每个细胞中的转录动态速率。
- 细胞轨迹推断:将 RNA 速度嵌入到低维空间(如 UMAP)中,以流式图(Streamline Plot)的形式可视化细胞状态转变的矢量场。
- 驱动基因鉴定:识别在特定细胞谱系转变过程中起关键调控作用的速度基因(Velocity Genes)。
- 潜伏时间推断:估计细胞在转录事件发生后达到特定状态所需的时间(Latent Time)。
本篇文档旨在为研究者提供一份详尽的 scVelo 技术指南,内容涵盖其核心原理、在 SeekSoul Online 云平台上的操作方法、结果解读、实战案例及常见问题,帮助您深入理解并高效利用 scVelo 进行细胞动态过程的研究。
RNA 速度概述
什么是 RNA 速度(RNA Velocity)
RNA 速度(RNA Velocity)是 scVelo 分析的核心概念,它指的是在某个时间点,单个细胞中单个基因 mRNA 丰度的变化速率。这个概念的生物学基础是基因的转录过程:
- 转录:基因被转录成带有内含子(Introns)的前体 mRNA(pre-mRNA),对应测序数据中的未剪切(Unspliced) reads。
- 剪切:内含子被切除,外显子(Exons)连接在一起,形成成熟 mRNA(mature mRNA),对应测序数据中的已剪切(Spliced) reads。
- 降解:成熟 mRNA 最终被降解。
通过定量一个细胞中某个基因的未剪切和已剪切 mRNA 的比例,scVelo 可以推断该基因是处于上调(未剪切 > 预期稳态比例)、下调(未剪切 < 预期稳态比例)还是稳定状态。将所有基因的速度信息整合起来,就可以得到该细胞在转录组水平上的整体变化方向,即“RNA 速度矢量”。
scVelo 的优势
TIP
相较于传统的拟时序分析,scVelo 具有以下显著优势:
- 方向性:能够直接推断出细胞状态转变的方向,解决了传统轨迹分析中“起点”和“终点”难以确定的问题。
- 瞬时性:捕捉的是细胞在当前时刻的动态变化趋势,而非基于过去状态的排序。
- 无监督:无需预设起始细胞或分化路径,完全由数据驱动。
- 循环过程解析:能够更好地识别和分析细胞周期等循环性的生物学过程。
scVelo 理论基础
scVelo 的理论核心是**RNA 速度(RNA Velocity)**的动态模型。该模型基于一个简单而强大的生物学原理:基因表达是一个动态过程,包括转录、剪切和降解三个主要步骤。通过量化这一过程中间产物(未剪切 mRNA)和最终产物(已剪切 mRNA)的丰度,我们可以推断出基因表达的变化方向和速率。
动态转录模型
scVelo 将每个基因的转录动力学过程数学化,其基本方程描述了未剪切(
- 转录(Transcription):
- 剪切(Splicing):
其中:
是基因的转录速率。 是从未剪切到已剪切的剪切速率。 是已剪切 mRNA 的降解速率。
在 scVelo 中,转录速率
速度的计算
RNA 速度(
这个公式是 scVelo 计算的核心。它告诉我们,一个基因的速度取决于其**新合成的 mRNA 量(
- 当
时,速度为正,表示基因正在被上调。 - 当
时,速度为负,表示基因正在被下调。 - 当两者相等时,系统处于稳态(Steady State)。
模型模式
scVelo 提供了几种模式来求解上述方程和推断速度:
稳态模型(Steady-State Model): 这是最初 RNA Velocity 方法的基础。它假设细胞处于转录稳态,即转录速率
和降解速率 为常数。此时,未剪切和已剪切 mRNA 之间存在一个线性的稳态比例( )。速度被计算为观测值与这条稳态线的残差。该模型简单快速,但无法准确描述基因上调或下调的完整动态过程。 动态模型(Dynamical Model): 这是 scVelo 的标志性改进。该模型放宽了稳态假设,允许转录速率
随时间变化,从而能够对基因的上调和下调阶段分别建模。它通过期望最大化(EM)算法为每个基因拟合转录速率( )、剪切速率( )和降解速率( ),并推断出每个细胞所处的具体转录状态(诱导、抑制或稳态)。这使得速度估计更为准确,尤其适用于描述复杂的、非稳态的生物学过程。
速度可视化
计算出每个细胞中每个基因的速度后,scVelo 会将这些高维的速度矢量投影到我们熟悉的低维空间(如 UMAP 或 t-SNE)中进行可视化。
- 速度图(Velocity Graph): 首先,scVelo 会构建一个“速度图”,这是一个
(N 为细胞数)的转换矩阵。矩阵中的每个元素代表细胞 i 转向细胞 j 的可能性,这个可能性是基于细胞 i 的速度矢量与细胞 i 到细胞 j 的方向矢量之间的余弦相似度来计算的。 - 流式图(Streamline Plot): 最后,通过在低维空间中对速度矢量进行平均或插值,scVelo 生成了直观的流式图。图中的箭头清晰地展示了细胞群体在转录空间中的主要流动方向和轨迹,从而揭示了细胞分化的路径和命运决定点。
云平台操作指南
在 SeekSoul Online 云平台上进行 scVelo 分析,操作流程直观便捷。
分析前的准备
TIP
scVelo 分析对输入文件有特定要求。在开始分析前,请务必确认:
- 输入文件为
loom格式:您需要提供包含剪切(spliced)和未剪切(unspliced) reads 计数的loom文件。这是计算 RNA 速度的根本依据。 - 数据已完成预处理:数据应已完成标准的质控、降维(PCA、UMAP)、聚类和细胞类型注释。
- 选择了合适的细胞亚群:RNA 速度分析应在具有潜在动态转变关系的细胞亚群中进行,以确保结果的生物学意义。
参数详解
| 界面参数 | 说明 |
|---|---|
| 任务名称 | 本次分析的任务名称,需以英文字母开头,可包含英文字母、数字、下划线和中文。 |
| 数据信息 | - 样本列:样本标签。 - 样本名称:样本的名称。 - loom 文件:上传对应样本的 loom文件。 |
| 分组因子 | 选择要分析的细胞类型或者聚类对应的标签。例如CellAnnotation,与“细胞类型”配合使用。 |
| 细胞类型 | 多选,选择要分析的细胞类型或者聚类结果,如Monocyte和Macrophage等。 |
| 备注 | 自定义备注信息。 |
操作流程
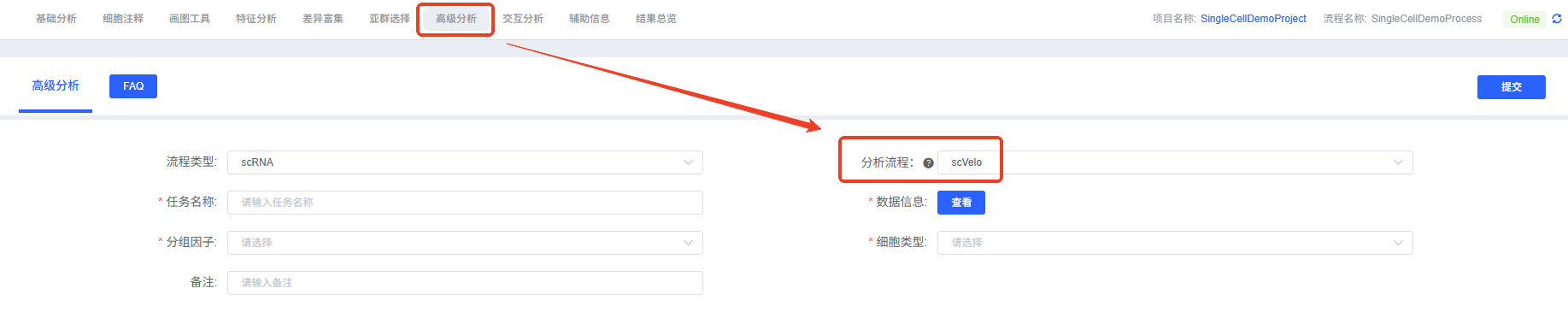
- 进入分析模块:在云平台导航至“高级分析”模块,分析流程选择“scVelo”。
- 创建新任务:为您的分析任务命名,并上传
loom文件。 - 配置参数:根据上述指南,选择要分析的分组和细胞类型。
- 提交任务:确认参数无误后,点击“提交”按钮,等待分析完成。
- 下载与查看:分析结束后,在任务列表中下载并查看生成的分析报告和结果文件。
结果解读
scVelo 分析报告提供了一系列图表,从不同维度揭示了细胞状态转变的动态过程。以下是对核心结果的详细解读。
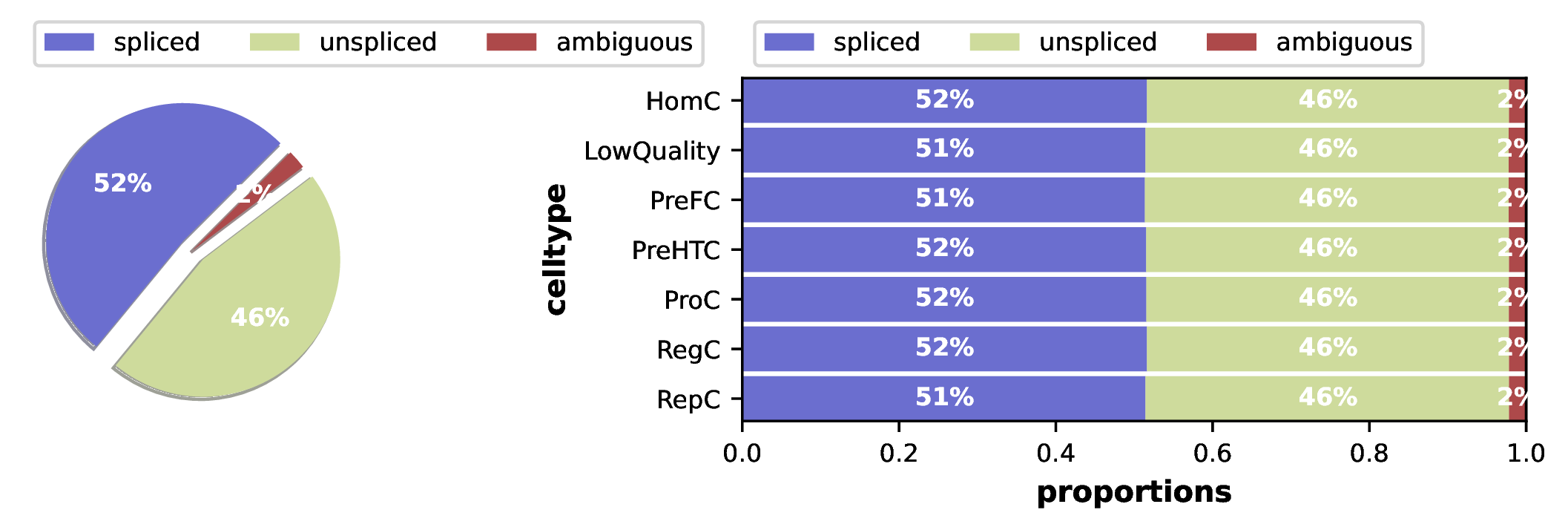
Spliced/Unspliced RNA 比例图
该图展示了在不同细胞类型中,已剪切(spliced)和未剪切(unspliced)RNA 的相对丰度。
- 解读要点:
- 左图(总体比例):显示了所有细胞中 spliced 与 unspliced reads 的总计数比例。通常,unspliced reads 占 10%-25%是比较典型的情况,这反映了整体的转录和剪切效率。
- 右图(按细胞类型):更精细地展示了每个细胞亚群的剪切效率差异。一般来说,未分化或处于快速增殖/分化状态的细胞(如祖细胞、干细胞)通常具有更高的转录活性,其 unspliced RNA 的比例也相对更高。而分化成熟、功能稳定的细胞,其转录活动趋于稳态,unspliced 比例可能较低。
- 质量控制:通过此图可以检查不同细胞类型的剪切效率是否一致。如果某个亚群的比例异常高或低,可能需要检查数据质量或考虑其独特的生物学特性。
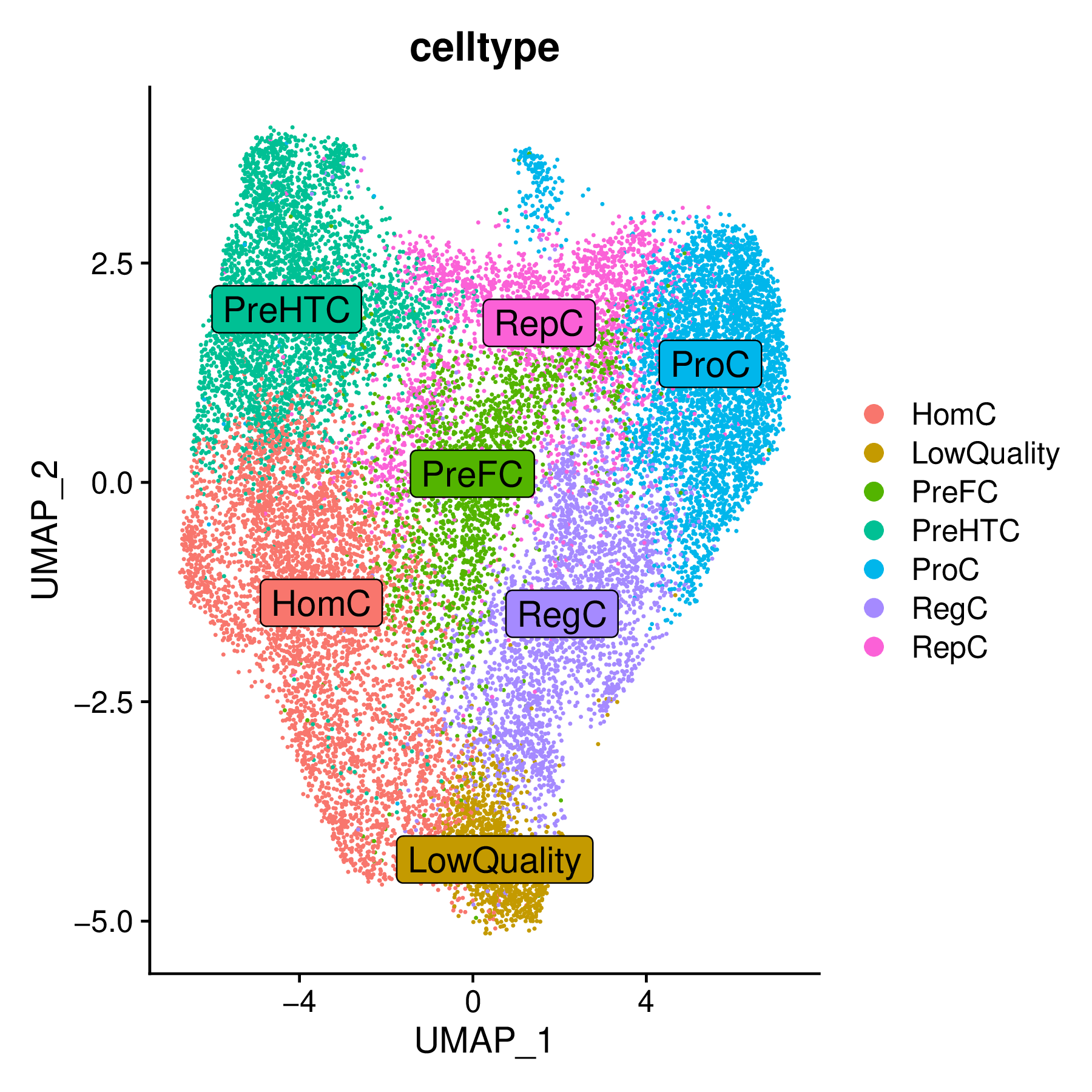
细胞降维图
这是标准的 UMAP/t-SNE 降维图,展示了参与分析的各个细胞亚群的分布情况。
- 解读要点:
- 细胞身份:图中每个点代表一个细胞,不同颜色对应不同的细胞类型或聚类。这是后续所有速度分析和轨迹可视化的基础。
- 空间关系:细胞在图上的远近关系反映了它们在转录组水平上的相似性。通常,具有分化关系的细胞类型在空间上会比较邻近。
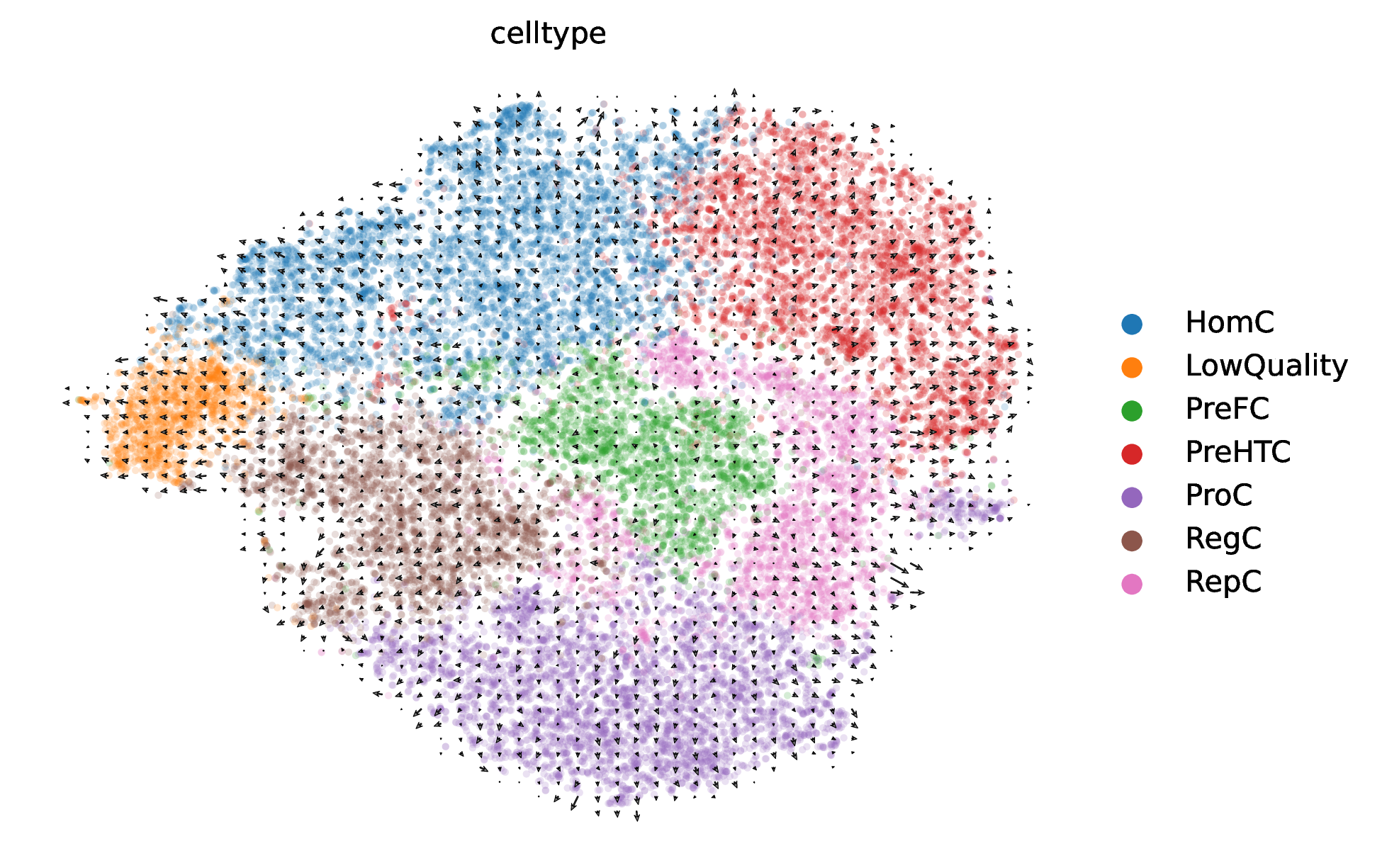
RNA 速度分化轨迹图
这是 scVelo 分析最核心的结果图。它在细胞降维图的基础上,叠加了一个矢量场(Vector Field),以箭头的形式直观地展示了细胞状态转变的方向和趋势。
- 解读要点:
- 箭头方向:箭头指示了 RNA 速度预测的细胞“未来”状态的走向。一个连续的箭头流(Streamline)代表了一条潜在的细胞分化或转变路径。
- 轨迹推断:通过观察箭头的流动,可以无监督地推断出细胞分化的起点(通常是箭头流出的区域)、中间过渡态和终点(箭头汇入的区域)。例如,如果箭头从细胞类型 A 指向细胞类型 B,则表明存在从 A 到 B 的分化轨迹。
- 循环过程:如果箭头形成一个闭环,则可能表示存在细胞周期等循环性的生物学过程。
潜伏时间(Latent Time)图
潜伏时间是 scVelo 动态模型推断出的一个重要的时间度量,它代表了每个细胞在其自身动态变化轨迹中所处的位置,可以理解为细胞的“内部生物钟”。

- 解读要点:
- 颜色梯度:图中颜色的深浅(通常从蓝到黄)代表了潜伏时间的早晚。颜色越接近 0(或越冷),表示细胞越处于分化过程的早期或起始状态;颜色越接近 1(或越暖),表示细胞越接近分化末端。
- 验证轨迹:潜伏时间的分布趋势应该与前面速度流图推断的轨迹方向一致。例如,沿着一条从 A 到 B 的分化路径,细胞的潜伏时间应该逐渐增加。这为推断的轨迹提供了有力的佐证。
Top 驱动基因热图
为了找出驱动细胞状态转变的关键基因,scVelo 会根据基因在动态模型中的似然性(Likelihood)进行排序。似然性越高的基因,其表达动态与模型拟合得越好,越有可能是驱动轨迹变化的关键调控因子。该热图展示了似然性排名靠前的基因(如 Top 300)的表达模式。
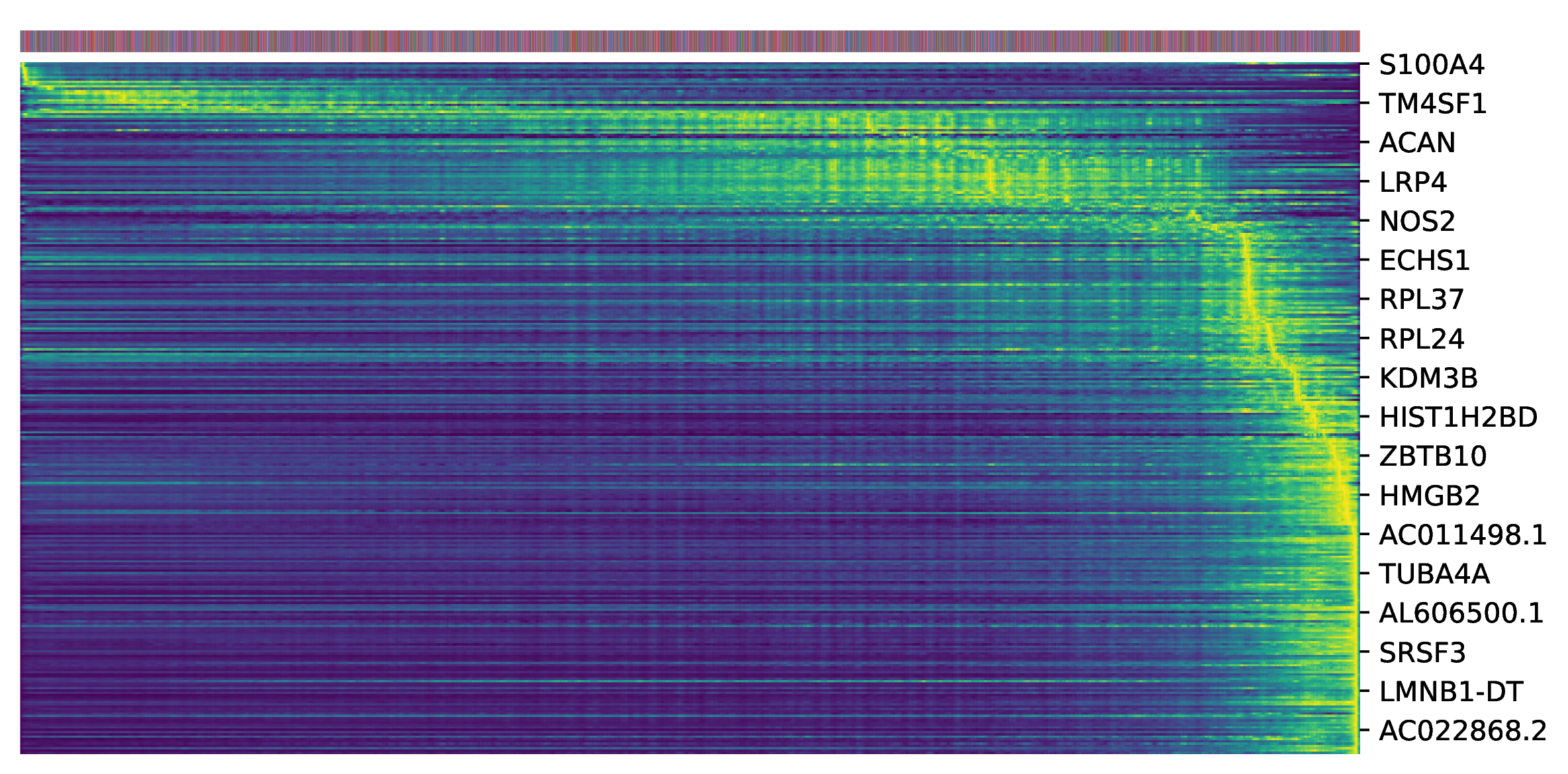
- 解读要点:
- 横坐标:细胞按照其“潜伏时间”从早到晚进行排序。
- 纵坐标:似然性排名靠前的基因。
- 表达模式:通过观察基因在热图上的表达模式,可以识别出在分化早期、中期或晚期特异性高表达的基因簇。这些基因很可能在细胞命运决定的相应阶段扮演着关键角色,是后续进行功能验证的重点候选对象。
结果文件清单
| 文件名 | 内容说明 |
|---|---|
scvelo_spliced_unspliced_prop.png | Spliced/Unspliced RNA 比例图。 |
scvelo_*_embedding_grid|stream.png | RNA 速度矢量场图 (*代表降维方法)。 |
scvelo_*_latentTime.png | 细胞潜伏时间图。 |
scvelo_latentTime_topGenes.png | Top 驱动基因热图。 |
scvelo_latentTime_data.csv | 核心数据:每个细胞的潜伏时间值。 |
top_gene_300.csv | 核心基因:Top 300 驱动基因列表。 |
应用案例
scVelo 作为一种强大的轨迹推断工具,已在众多研究领域得到广泛应用。以下是两个典型的应用案例。
案例一:解析小鼠海马体细胞谱系
在一项关于小鼠海马体发育的研究中,研究者利用 scVelo 来验证和解析细胞分化的方向。

- 背景:通过对细胞进行聚类和标记基因(Marker)分析,研究者初步鉴定出了神经干细胞(Neuroblast)、中间神经元(Granule)、CA 区神经元以及少突胶质前体细胞(OPC)等多个细胞亚群。
- scVelo 应用:通过运行 scVelo,分析报告生成了 RNA 速度轨迹图(c 图)。图中的箭头清晰地显示,细胞流从 Neuroblast 群体分别流向 Granule 和 CA 群体,同时另一条轨迹显示了从 preOPC 向 OPC 的转化过程。
- 结论:scVelo 的结果不仅与基于标记基因的细胞身份鉴定结果(a 图)高度一致,还为这些离散的细胞亚群之间建立了动态的、有方向性的连接,从而验证并深化了对海马体细胞谱系分化的理解。
案例二:预测组织发育轨迹
scVelo 能够基于转录本的剪接动态来预测组织或器官在发育过程中的整体轨迹。
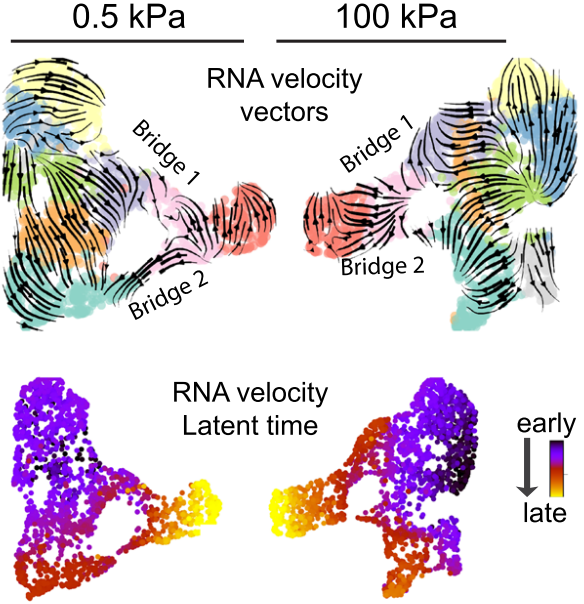
- 背景:在复杂的组织发育过程中,多种细胞类型协同分化,形成功能性结构。理解这一过程的动态和方向性是发育生物学的核心问题。
- scVelo 应用:研究者利用 scVelo 分析了发育中组织的数据。上图以箭头流的形式直观展示了主要的细胞分化路径,而下图则通过推断的潜伏时间(Latent Time)从另一个维度描绘了同样的发育进程,颜色由冷至暖代表了从发育早期到晚期的连续变化。
- 结论:两种可视化方式相互印证,共同揭示了组织发育的连续轨迹,并为研究特定时间点的调控事件提供了“伪时间”轴。
注意事项
为了确保 scVelo 分析结果的准确性和可靠性,请遵循以下建议:
WARNING
数据质量是关键
- Loom 文件准备:确保用于分析的
loom文件是使用标准流程(如velocyto)生成的,其中必须包含正确的spliced和unsplicedreads 计数。错误的输入将导致不可靠的速度估计。
1. 细胞类型选择
- 生物学相关性:在平台参数中选择的“细胞类型”应具有潜在的生物学连续性。将毫不相关的细胞类型(如 T 细胞和上皮细胞)放在一起进行分析,可能会产生无意义的轨迹。
- 细胞数量:每个细胞亚群应包含足够数量的细胞,以确保速度估计的稳健性。过于稀少的细胞群可能会导致速度估计不准确。
2. 结果的生物学验证
- 不要孤立解读:scVelo 提供的轨迹是基于转录动态的计算预测。务必将其与您已知的生物学知识、标记基因表达模式以及其他拟时序分析工具(如 Monocle、PAGA)的结果进行相互验证。
- 关注驱动基因:报告中提供的 Top 驱动基因列表是寻找核心调控因子的宝贵线索。请重点关注这些基因,并通过文献查询、功能富集分析或实验验证来确认它们在细胞命运决定中的作用。
3. 理解模型的局限性
- 转录后调控:scVelo 模型主要基于转录和剪接动力学,无法捕捉到翻译后修饰、蛋白降解等转录后调控事件对细胞状态的影响。
- 复杂动力学:对于存在多个速率、复杂调控回路或转录爆发(Transcriptional Bursting)非常剧烈的基因,模型的拟合可能会有偏差。
4. 可视化的误导性
- 降维失真:所有在二维空间(如 UMAP)中的可视化都存在一定程度的失真。细胞在图上的距离并不完全等同于它们真实的转录组差异。因此,解读轨迹时应更关注箭头的局部方向性,而非其全局的长度或弯曲度。
常见问题解答(FAQ)
Q1: 为什么我的分析结果中没有看到明显的箭头或轨迹?
A1: 可能的原因有多种:
- 生物学过程:您所分析的细胞群体可能本身就处于一个相对稳态的系统中,没有剧烈的、有方向性的状态转变。例如,一群功能成熟且稳定的细胞。
- 数据质量:
unsplicedreads 的比例过低或数据噪音过大,可能导致无法准确估计 RNA 速度。 - 参数选择:选择的细胞类型之间确实没有直接的分化关系。
- 基因过滤:用于速度计算的基因数量过少,无法提供足够强的方向性信号。
Q2: scVelo 推断的“潜伏时间”是真实的细胞分化时间吗?
A2: 不是。潜伏时间(Latent Time)是一个相对的、排序性质的度量,它反映了细胞在转录动态轨迹中所处的位置,可以理解为“伪时间”或“生物钟”。它与真实的物理时间(小时、天)并不直接等同,但其变化趋势与真实时间正相关。例如,潜伏时间为 0.8 的细胞确实比 0.2 的细胞更接近分化终点。
Q3: scVelo 和 Monocle 这两种拟时序分析工具有什么区别?我应该如何选择?
A3: 这是一个很好的问题,它们各有侧重:
- Monocle (2/3):主要基于细胞间的转录相似性来构建轨迹。它通过“学习”一条穿过细胞群的路径来对细胞进行排序。它擅长描绘一个已经发生的分化过程的“地图”,但无法内在地确定起点和终点,通常需要用户根据先验知识指定。
- scVelo:基于RNA 剪接动力学来推断每个细胞未来的状态方向。它的优势在于能够无监督地、有方向性地预测轨迹,直接告诉你细胞“将要去向哪里”。
- 如何选择:
- 如果您想预测细胞分化的方向、寻找命运决定点、研究循环过程,或者您的系统中起点和终点不明确,scVelo 是首选。
- 如果您更关心鉴定不同分化阶段(State)、寻找沿轨迹差异表达的基因,并且对分化的大致方向有先验知识,Monocle 是非常强大的工具。
- 最佳实践:将两者结合使用。例如,用 scVelo 确定轨迹的大方向和关键驱动基因,再用 Monocle 对这条轨迹进行更精细的阶段划分和差异基因分析。
Q4: 报告中的 Top 驱动基因是如何计算出来的?
A4: 这些基因是基于它们在 scVelo动态模型中的**似然性(Likelihood)**进行排序的。一个基因的似然性值越高,意味着它的spliced/unspliced表达模式与模型所描述的动态转录过程(上调、下调、稳态)拟合得越好。因此,这些高似然性的基因被认为是细胞状态转变过程中,其转录调控最为清晰和关键的基因,即“驱动基因”。
参考资料
[1] LA MANNO G, SOLDATOV R, ZEIRA A, et al. RNA velocity of single cells[J]. Nature, 2018, 560(7719): 494-498.
[2] BERGEN V, LANGE M, PEIDLI S, et al. Generalizing RNA velocity to transient cell states through dynamical modeling[J]. Nature Biotechnology, 2020, 38(12): 1408-1414.
[3] LI T, et al. On the mathematics of RNA velocity I: theoretical analysis[EB/OL]. bioRxiv, 2020.
[4] QIU X, ZHANG Y, YANG D, et al. Mapping vector field of single cells[EB/OL]. bioRxiv, 2021.
[5] SUNADOME K, et al. Directionality of developing skeletal muscles is set by mechanical forces[J]. Nature Communications, 2023, 14(1): 3060.
