全序列-可变剪接分析
介绍
NOTE
可变剪接(alternative splicing)又称作选择性剪接,是指在真核生物的基因转录过程中,前体RNA经过不同的剪接形式,产生多个不同的转录本。在这个剪接过程中,不同的外显子和内含子可以选择性地被剪除或保留。
IMPORTANT
寻因研发的全序列测序技术(scFAST-seq),是基于随机引物来捕获RNA分子,在单细胞的转录组水平上实现了全长覆盖,从而为可变剪接分析创造了机会和可能。这项转录组测序技术是基于droplet-based原理的高通量测序技术,因此,我们更建议在细胞群水平或样本水平上进行可变剪接分析。
rMATS分析软件
一、软件介绍
我们使用了rMATS来分析可变剪接,rMATS(eplicate Multivariate Analysis of Transcript Splicing)是一个基于RNA-seq数据来分析差异性可变剪接事件的软件。rMATS可以识别不同类型的可变剪接事件,包含 SE,A5SS,A3SS,MXE和RI五种类型。
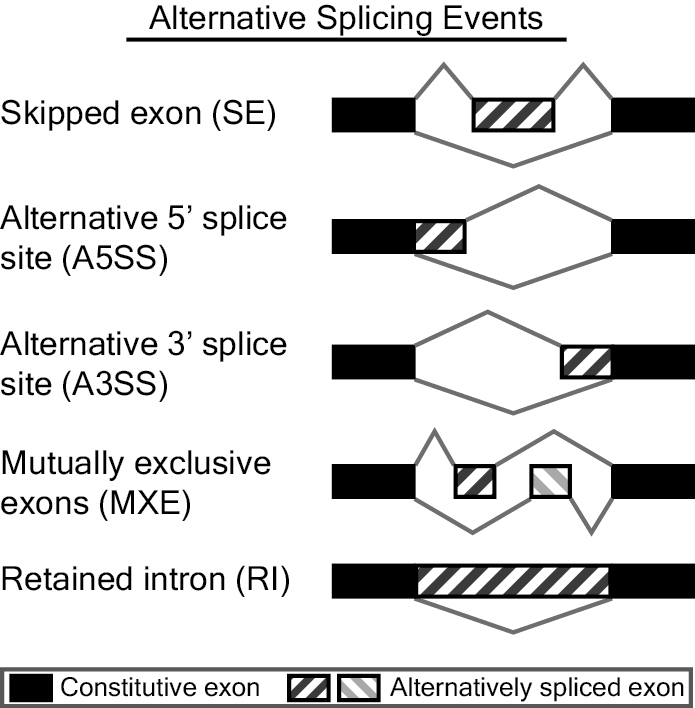
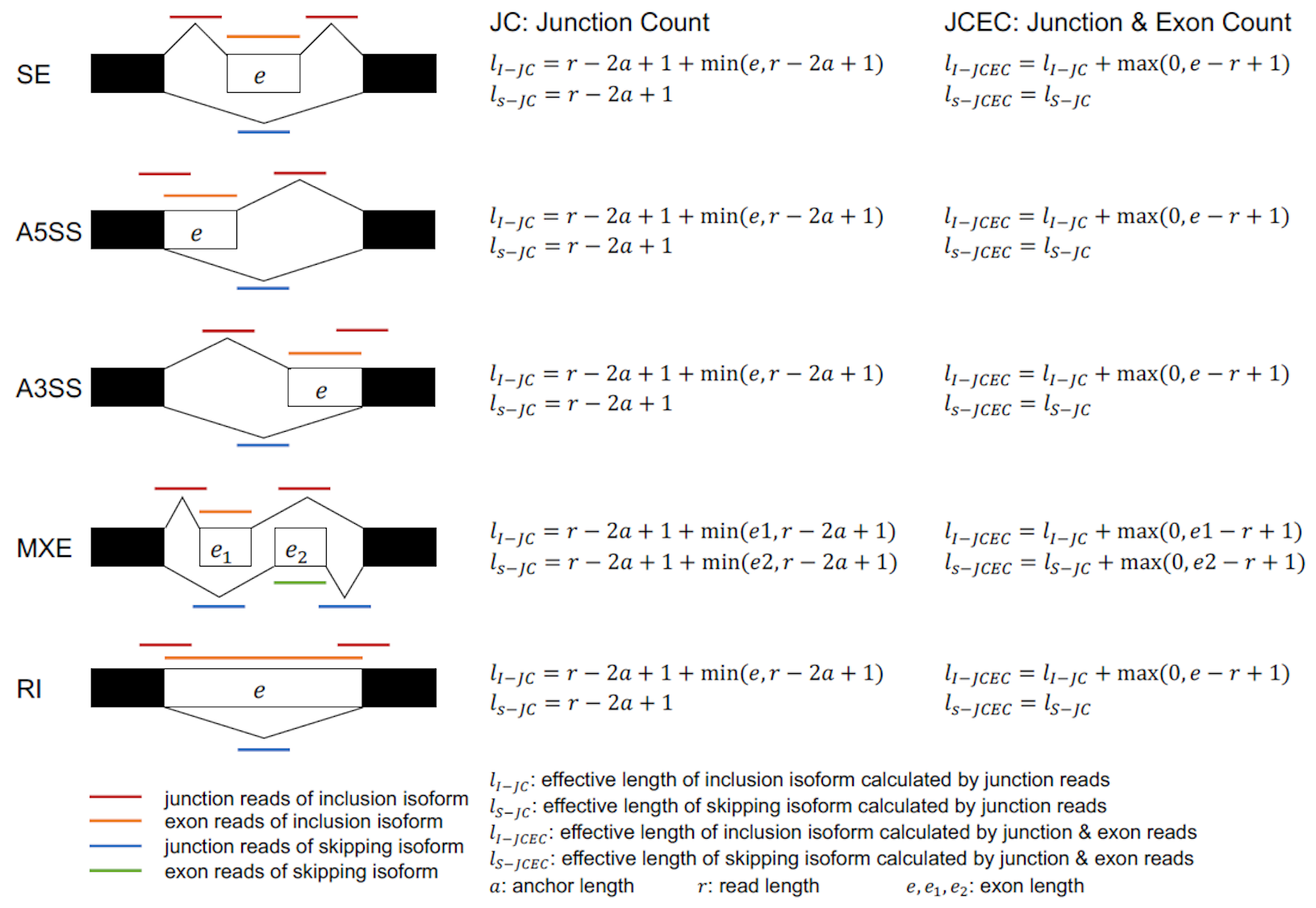
对识别到的可变剪接事件,rMATS分别按照两种不同的计算方式来统计支持reads数。(JC,Junction Counts;JCEC,Junction Counts and Exon Counts)
二、软件安装
1)conda安装rMATS:
TIP
推荐使用conda来安装rMATS软件,首先创建一个新的conda环境,您也可以选择激活已有环境:
conda create -n rMATS_env
conda activate rMATS_env安装:
conda install bioconda::rmats运行成功后,rMATS就安装好了。
2)配置环境:
IMPORTANT
运行后续代码,需要配置一些必要的工具,按照以下示例分别安装python包和R包:
# install python packages
python -m pip install pysam click
# install R
conda install r-base
# install R packages
Rscript -e 'install.packages(c("argparse", "Seurat"))''至此,分析环境已经配置完成了!
示例数据和脚本下载
在这里为您提供了相应的示例数据和分析脚本,来帮助您理解后续的分析流程。该数据均来源于寻因官网PBMC数据。
wget https://seekgene-public.oss-cn-beijing.aliyuncs.com/software/FAST/rMATS_demo.tar.gz
tar -zxvf rMATS_demo.tar.gzNOTE
示例数据包含3个文件:
PBMC.rds是基于SingleCellExperiment格式的单细胞数据PBMC_chr1.bam是从PBMC样本测序数据与GRCh38基因组的比对结果中抽取了1号染色体上的比对数据作为示例genes.gtf是基于GRCh38版本的基因组注释文件
三个脚本中,getbarcode.R 是用于读取rds的细胞barcode信息和分组信息;rmats_run.py首先根据指定的分组信息对bam文件进行拆分,然后使用rMATS来识别可变剪接事件,并在指定分组之间计算差异表达水平。run.sh是直接调用的运行脚本。
运行示例
CAUTION
运行以下命令前,请确保已激活环境:
conda activate rMATS_env运行以下命令,完成可变剪接分析:
# create output path
mkdir -p /path/result
# run
python /path/rmats_run.py \
--samplename demo \
--rds /path/demodata/PBMC.rds \
--outdir /path/result \
--bam /path/demodata/PBMC_chr1.bam \
--gtf /path/demodata/genes.gtf \
--cellanno seurat_clusters上述参数含义为:
| 参数名称 | 参数含义 |
|---|---|
| samplename | 样本名称 |
| rds | 基于SingleCellExperiment格式的数据,Seurat对象 |
| outdir | 输出目录 |
| bam | 比对结果文件 |
| gtf | 基因组注释文件 |
| cellanno | 指定的metadata名,需存在于rds里。注:cellanno列不能包含"/" |
TIP
小编用示例数据运行,在4core16GB的环境下,运行了28分钟左右。
结果解读
运行完成后得到了哪些结果?又该如何理解呢?接下来,为您简单介绍结果内容。
首先,查看结果目录,可以看到输出结果按照指定的cluster分为了不同的文件夹,以cluster0为例:
tree -L 1 result/cluster0/
result/cluster0/
├── A3SS.MATS.JCEC.txt
├── A5SS.MATS.JCEC.txt
├── demo.b1.txt
├── demo.b2.txt
├── demo_cluster0_A3SS.MATS.JC.txt
├── demo_cluster0_A5SS.MATS.JC.txt
├── demo_cluster0_MXE.MATS.JC.txt
├── demo_cluster0_RI.MATS.JC.txt
├── demo_cluster0_SE.MATS.JC.txt
├── fromGTF.A3SS.txt
├── fromGTF.A5SS.txt
├── fromGTF.MXE.txt
├── fromGTF.novelJunction.A3SS.txt
├── fromGTF.novelJunction.A5SS.txt
├── fromGTF.novelJunction.MXE.txt
├── fromGTF.novelJunction.RI.txt
├── fromGTF.novelJunction.SE.txt
├── fromGTF.novelSpliceSite.A3SS.txt
├── fromGTF.novelSpliceSite.A5SS.txt
├── fromGTF.novelSpliceSite.MXE.txt
├── fromGTF.novelSpliceSite.RI.txt
├── fromGTF.novelSpliceSite.SE.txt
├── fromGTF.RI.txt
├── fromGTF.SE.txt
├── JCEC.raw.input.A3SS.txt
├── JCEC.raw.input.A5SS.txt
├── JCEC.raw.input.MXE.txt
├── JCEC.raw.input.RI.txt
├── JCEC.raw.input.SE.txt
├── JC.raw.input.A3SS.txt
├── JC.raw.input.A5SS.txt
├── JC.raw.input.MXE.txt
├── JC.raw.input.RI.txt
├── JC.raw.input.SE.txt
├── MXE.MATS.JCEC.txt
├── RI.MATS.JCEC.txt
├── SE.MATS.JCEC.txt
├── summary.txt
└── tmp
1 directory, 38 files上述txt文件是rMATS对cluster0相对其余cluster进行的差异可变剪切分析结果,各个文件介绍可以参考rMATS官方说明: rmats-turbo/README.md at v4.3.0 · Xinglab/rmats-turbo (github.com)
在此,为您介绍主要的输出结果内容:
一、主要结果 demo_cluster0_*.MATS.JC.txt
该结果分为 SE,A5SS,A3SS,MXE和RI五个文件,分别对应5种可变剪接类型。
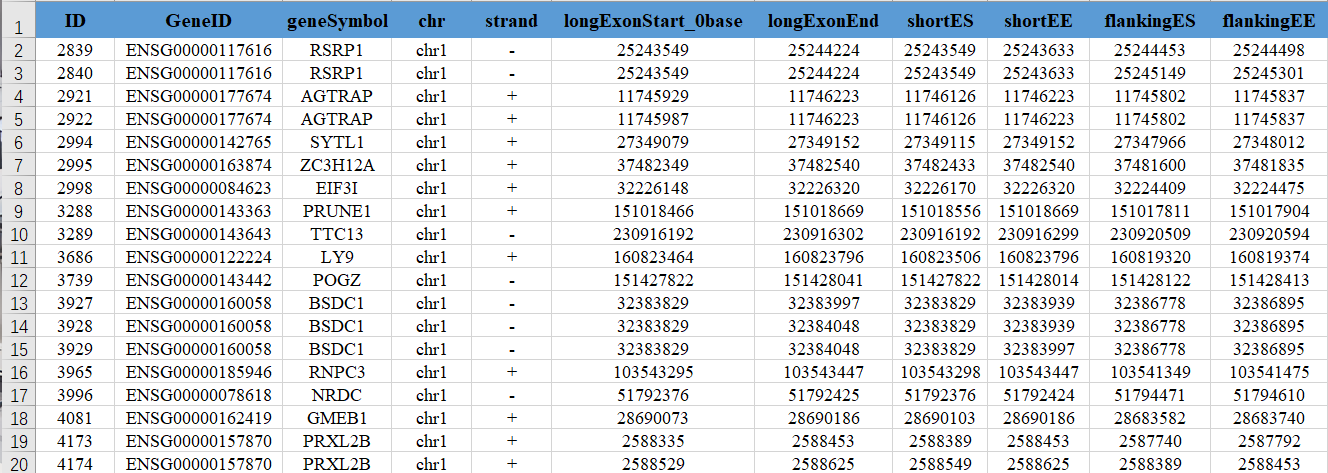
内容包含了各个剪接事件的起始结束、比对reads的详细信息,并就cluster0和其他所有cluster之间计算了各剪接事件的差异水平。各列描述为:
(1)ID:rMATS event id
(2)GenelD: 可变剪接事件所在基因编号
(3)geneSymbol: 可变剪接事件所在基因名称
(4)chr: 可变剪接事件所在染色体
(5)strand: 可变剪接事件所在染色体链的方向
(6)ExonStart_0base: 可变剪接事件跳跃外显子的起始位置,以0开始计数
(7)ExonEnd: 可变剪接事件跳跃外显子的终止位置
(8)upstreamES: 可变剪接事件跳跃外显子的上游exon起始位置
(9)upstreamEE: 可变剪接事件跳跃外显子的上游exon终止位置
(10)downstreamES: 可变剪接事件跳跃外显子的下游exon起始位置
(11)downstreamEE: 可变剪接事件跳跃外显子的下游exon终止位置
(12)ID:rMATS event id
(13)IJC_SAMPLE_1: 样本一在inclusion junction(IJC)下的count数,重复样本的结果以逗号分隔
(14)SJC_SAMPLE_1: 样本一在skipping junction(SJC)下的count数,重复样本的结果以逗号分隔
(15)IJC_SAMPLE_2: 样本二在inclusion junction(IJC)下的count数,重复样本的结果以逗号分隔
(16)SJC_SAMPLE_2: 样本二在skipping junction(SJC)下的count数,重复样本的结果以逗号分隔
(17)IncFormLen: 可变剪接事件Exon Inclusion Isoform的有效长度
(18)SkipFormLen: 可变剪接事件Exon Skipping Isoform的有效长度
(19)PValue: 两组样本间可变剪接事件表达差异显著性p值
(20)FDR: 可变剪接事件表达差异显著性FDR值
(21)IncLevel1: 处理组可变剪接事件Exon Inclusion Isoform在两个Isoform总表达量的比值
(22)IncLevel2: 对照组可变剪接事件Exon Inclusion Isoform在两个Isoform总表达量的比值
(23)IncLevelDifference: IncLevel1与IncLevel2的差值
二、主要结果 summary.txt

该文件是对cluster0的差异可变剪接事件的汇总,统计了 FDR<0.05 的可变剪接事件数量。
其中 SignificantEventsJC/JCEC 是 SigEventsJC/JCECSample1HigherInclusion 和 SigEventsJC/JCECSample2HigherInclusion 的加和。JC和JCEC是指由两种不同计算方式各自得到的结果。
TIP
更多结果的查询和了解可以移步rMATS官网https://github.com/Xinglab/rmats-turbo/blob/v4.3.0/README.md
参考文献:
Shen S., Park JW., Lu ZX., Lin L., Henry MD., Wu YN., Zhou Q., Xing Y. rMATS: Robust and Flexible Detection of Differential Alternative Splicing from Replicate RNA-Seq Data. PNAS, 111(51):E5593-601. doi: 10.1073/pnas.1419161111
