全序列的突变分析-新流程
导语:
NOTE
基于单细胞表达数据,我们能够深入探索每个细胞的基因表达模式,而结合了突变数据,则使得我们能够探索表达差异背后的可能影响因素。突变不仅有助于理解疾病是如何在细胞层面演变和扩散,也能够体现细胞在发育和功能上的多样性,是为研究生物体内部多样性和适应性提供了新的视角。
IMPORTANT
寻因的SeekOne全序列产品打破常规的3'或5'端转录组测序的局限,利用随机引物进行全转录组的随机捕获,得到了更丰富的转录组信息,因此,利用全序列数据可以进行单细胞转录组数据的突变分析。同样的,来源于全序列文库的panel富集数据也可以用来进行突变分析。
在本文档中,我们将详细描述对应的突变分析流程。该流程是参考了Souporcell (https://www.nature.com/articles/s41592-020-0820-1)而提出的。Souporcell是由 Heaton H.等人提出的基于基因型对细胞聚类的工具。需要注意的是,突变分析工具和流程众多,您可按照数据特征、工具特性自行选择。
NOTE
(突变:指DNA的变化,根据是否引起氨基酸变化可分为非同义突变和同义突变,根据突变类型可分为snp和indel,分别对应碱基替换、插入和缺失。)
part1:分析流程
基于全序列数据或panel数据,提出如下pipeline:
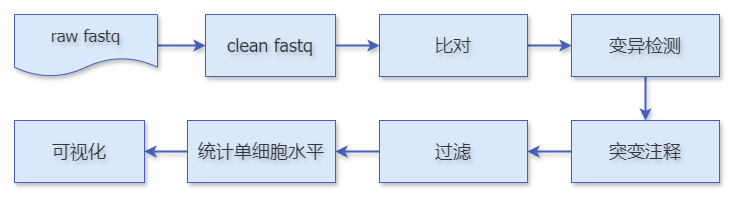
IMPORTANT
该流程首先通过seeksoultools软件识别测序reads中的标签信息,获得去除引物后的clean fastq数据,然后进行后续分析。在这里,使用了STAR软件进行比对,freebayes进行突变检测和vep进行突变注释。在得到所有突变后,考虑到测序错误等引起的假阳性,对突变结果进行过滤,最后通过vartrix来生成突变和覆盖矩阵,即得到了在单细胞水平上的突变信息。此外,我们还添加了差异分析和可视化的展示,以便于后续分析。
part2:运行环境与数据
1)获取数据:
2.1.1 原始数据
NOTE
以寻因官网人类细胞系数据为例,该数据是1:1:1混合了细胞系A549(KRAS G12S), HCC827(EGFR 19del) 和K562(BCR-ABL1 fusion)的全序列数据,因此可以作为示例对突变分析流程进行验证。
(以下代码均在终端命令行执行)
# fastq
wget https://seekgene-public.oss-cn-beijing.aliyuncs.com/software/data/demodata/panel.raw.tar.gz
# Decompress
tar -zxvf panel.raw.tar.gz2.1.2 获取分析脚本和rds数据
wget https://seekgene-public.oss-cn-beijing.aliyuncs.com/software/FAST/20250610_mut_script.zip
unzip 20250610_mut_script.zipTIP
该文件夹里包含了后续突变分析所需要的脚本和rds,这里的rds数据是细胞系数据经过全序列质控分析后得到的rds。
2.1.3 基因组数据
NOTE
人和小鼠的参考基因组可以在下方获取,其他物种可以参考http://seeksoul.seekgene.com/zh/v1.2.2/reference.html#ref-label来构建。这里只需要下载人类基因组数据。
# human
wget -c -O GRCh38.tar.gz "https://seekgene-public.oss-cn-beijing.aliyuncs.com/software/data/reference/GRCh38.tar.gz"
# decompress
tar -zxvf GRCh38.tar.gz# mouse
wget -c -O mm10.tar.gz "https://seekgene-public.oss-cn-beijing.aliyuncs.com/software/data/reference/mm10_ensemble_102.tar.gz"
tar -zxvf mm10.tar.gz2.1.4 vep注释数据库
IMPORTANT
vep(Variant Effect Predictor)是对突变进行注释的分析软件,可以在ENSEMBL(http://useast.ensembl.org/info/docs/tools/vep/script/vep_cache.html#cache)上下载,此处下载物种为人,版本为111的数据库:
wget https://ftp.ensembl.org/pub/release-111/variation/indexed_vep_cache/homo_sapiens_merged_vep_111_GRCh38.tar.gz
tar -zxf homo_sapiens_merged_vep_111_GRCh38.tar.gz2)环境配置:
2.2.1 seeksoultools的安装
# seeksoultools
wget -c -O seeksoultools_fast_mut_dev.tar.gz "https://seekgene-public.oss-cn-beijing.aliyuncs.com/software/FAST/Fusion.v1/seeksoultools_fast_mut_dev.tar.gz"
# Decompress
mkdir seeksoultools
tar -zxvf seeksoultools_fast_mut_dev.tar.gz -C seeksoultools
source seeksoultools/bin/activate
seeksoultools/bin/conda-unpack
# export to PATH
export PATH="`pwd`/seeksoultools/bin:$PATH"2.2.2 其他软件的安装:
TIP
推荐使用conda/micromamba安装,集成在一个环境中,您可以直接从yaml文件安装,也可以分步单独安装:
yaml文件安装:
micromamba env create -f mut_script/env.yaml
micromamba activate test_mut_pip单独安装:
创建环境并安装所需软件:
micromamba create -n test_mut_pip
micromamba activate test_mut_pip
micromamba install bioconda::freebayes bioconda::bcftools bioconda::ensembl-vep bioconda::vartrix安装R/python所需包:
# R
micromamba install r-base
Rscript -e 'install.packages(c("Seurat", "tidyverse", "argparse", "patchwork", "Matrix", "future.apply", "stringr"), repos="https://mirrors.tuna.tsinghua.edu.cn/CRAN/")'
# python
pip install click json loguru collections pandas pysam BioNOTE
版本:
# seeksoultools 1.2.0
# freebayes 1.3.6
# bcftools 1.21
# vep 111.0
# vartrix 1.1.22
# #####################
# python 3.7.12
# click 8.1.7
# json 2.0.9
# loguru 0.7.2
# pandas 2.2.2
# pysam 0.22.1
# biopython 1.81
# #####################
# R 4.4.1
# Seurat 5.1.0
# tidyverse 2.0.0
# argparse 2.2.3
# patchwork 1.3.0
# Matrix 1.6-5
# future.apply 1.11.2
# stringr 1.5.1part3:运行示例和结果说明
输入:
star_index="/path/GRCh38/star"
fq1="/path/panel_R1_001.fastq.gz"
fq2="/path/panel_R2_001.fastq.gz"
sam="demo"
vepcache="/path/indexed_vep_cache/homo_sapiens_merged/111_GRCh38/"
rdsfile="/path/mut_script/demo.rds"
out="/path/output"
mkdir -p $out
cd $outNOTE
(注:您可按需求来完成测序数据质控过程)
step1: 提取标签信息
IMPORTANT
利用seeksoultools分析软件,识别barcode和UMI序列,并去除引物和接头序列,得到clean的fastq数据:
seeksoultools mut step1 \
--fq1 $fq1 \
--fq2 $fq2 \
--samplename $sam \
--outdir $out输出结果中sam.cutTSO_1.fq.gz和sam.cutTSO_2.fq.gz用于后续分析。
step2: 比对
接下来,使用STAR进行比对,并处理bam文件:
STAR \
--runThreadN 13 \
--limitOutSJcollapsed 5000000 \
--readMapNumber -1 \
--genomeDir $star_index \
--readFilesCommand zcat \
--outFileNamePrefix $out/${sam}.star. \
--outSAMtype BAM Unsorted \
--readFilesIn ${out}/step1/${sam}.cutTSO_1.fq.gz ${out}/step1/${sam}.cutTSO_2.fq.gz
samtools sort -@ 12 -O BAM -o $out/${sam}.star.sort.out.bam $out/${sam}.star.Aligned.out.bam
samtools index $out/${sam}.star.sort.out.bam
seeksoultools utils addtag --inbam $out/${sam}.star.sort.out.bam --outbam $out/${sam}.star.tagged_sorted.bam
samtools index $out/${sam}.star.tagged_sorted.bam输出结果说明:
| file | description |
|---|---|
| starAligned.out.bam | STAR输出的bam |
| sort.star.bam | STAR输出bam按照染色体位置排序后的bam |
| *.tagged_sorted.bam | tagged表示为bam增加标签,tag中的"CB" "UB"分别表示barcode和UMI序列 |
| *.bam.bai | 索引文件 |
step3: 突变检测、注释、过滤
使用mut.py脚本来完成突变的检测、注释、初步过滤等步骤:
python mut_script/mut.py \
--bam1 $out/${sam}.star.tagged_sorted.bam \
--rds $rdsfile \
--outdir $out \
--cachedata $vepcache \
--samplename $sam \
--species human \
--refpre $star_index/../fasta/genomestep4: 统计单细胞水平
在上一步中通过vartrix生成了突变矩阵,在这里保留属于细胞的突变信息,并生成相应的突变信息表格:
Rscript mut_script/tomatrix.R \
--refmatrix $out/matrix/ref_matrix.mtx \
--altmatrix $out/matrix/alt_matrix.mtx \
--annofile $out/${sam}.filter.vcf.txt \
--barcode $out/${sam}barcodelist.xls \
--variants $out/${sam}.filter.vcf.txt \
--outdir $out \
--sam $sam输出结果说明:
| file | description |
|---|---|
| sample.snp_indel.alt_UMI.matrix | 突变*细胞的矩阵,矩阵的值表示细胞发生该突变的UMI数 |
| sample.snp_indel.all_UMI.matrix | 突变*细胞的矩阵,矩阵的值表示细胞覆盖该突变位点的UMI数 |
sam.anno.cluster.xls
NOTE
存储突变的详细注释信息,不同列的说明可以参考http://asia.ensembl.org/info/docs/tools/vep/vep_formats.html#output
sam.simple.anno.cluster.txt
存储突变的基本信息,各列说明:
| column | description |
|---|---|
| sample | 样本名称 |
| Chr | 染色体信息 |
| pos | 突变所在位置 |
| Ref | 参考基因组碱基 |
| Allele | 突变后的碱基 |
| Consequence | 突变类型 |
| SYMBOL | 突变所在基因名称 |
| HGVSc | 突变导致的氨基酸改变 |
| HGVSp | 突变引起的蛋白改变 |
| Existing_variation | CLIN_SIG, SOMATIC, PHENO数据库中该位点的信息 |
| alt_clusters | 突变所在的cluster信息 |
| coverage_clusters | 覆盖该位点的cluster信息 |
step5: 差异分析和可视化
TIP
为了更好的理解突变在细胞群上的特征,可使用Fisher精确检验来计算突变在细胞群上是否有显著性富集,并借助umap图来展示突变在不同维度的表现:
Rscript mut_script/snv_module.R \
--rna_mat $rdsfile \
--name $sam \
--outdir $out/mutation_umap \
--snv_cover_mat $out/${sam}.snp_indel.all_UMI.matrix \
--snv_mut_mat $out/${sam}.snp_indel.alt_UMI.matrix结果文件中,demo_snv_markers.xls即为fisher检验结果,各列说明如下:
| column | description |
|---|---|
| SNV | 突变 |
| p_val | fisher检验p值 |
| ident*_ref | 覆盖该突变位点但未发生突变的细胞数 |
| ident*_mut | 发生该突变的细胞数 |
| ident*_cover | ref+mut |
| cluster | 目标细胞群 |
| p_val_adj | 校正后p值 |
(ident1 表示 目标细胞群,ident2 表示 非目标细胞群)
以 KRAS G12S 突变为例:
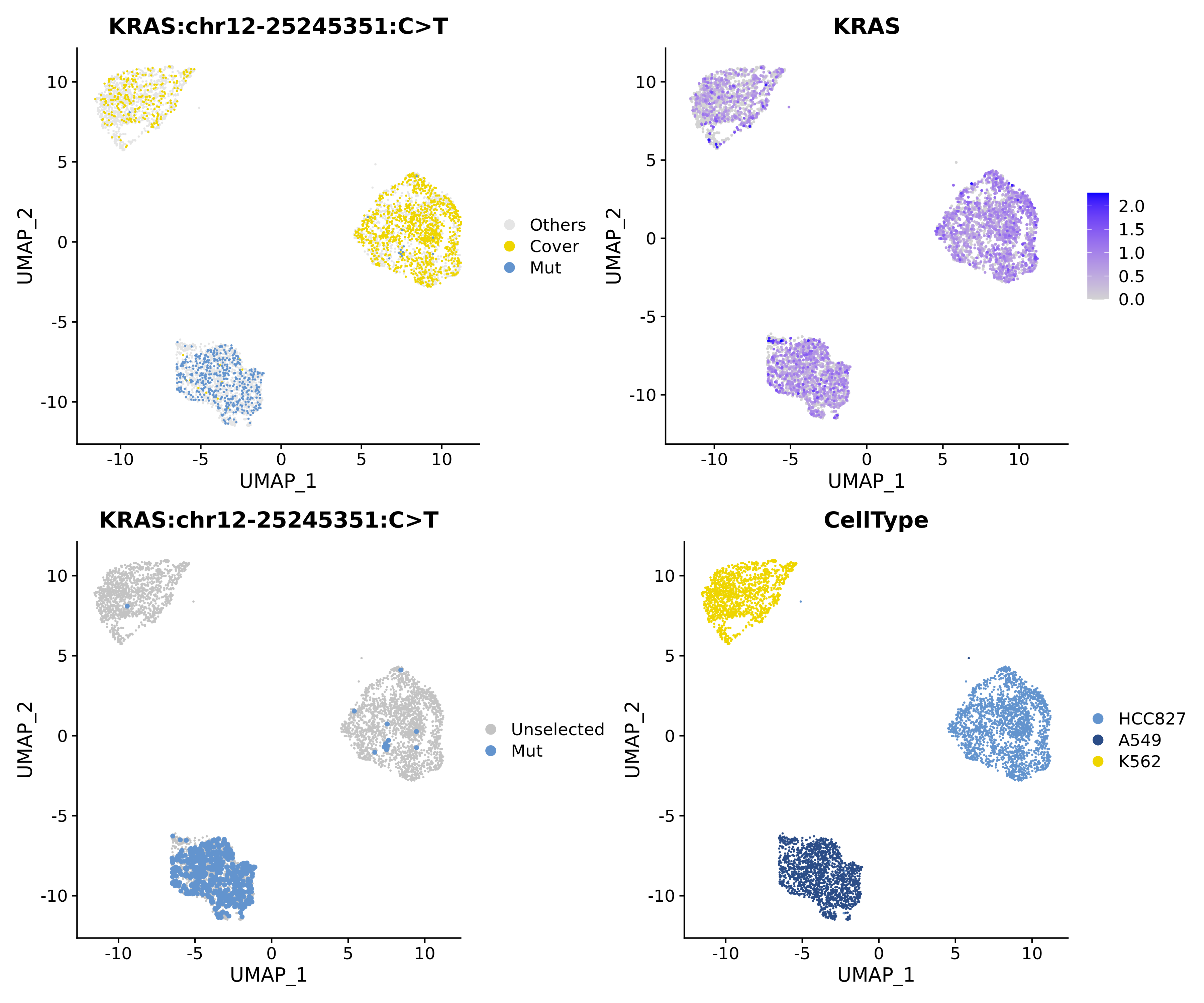
NOTE
图中每个点代表一个细胞
左上图:黄色cover表示覆盖该突变位点但未发生突变的细胞,蓝色代表发生该突变的细胞;
左下图:蓝色代表发生该突变的细胞;
右上图:该突变所在基因的表达水平,蓝色的深浅表示在RNA层面上表达量的高低。借助该图,可以了解该突变检出和基因表达与否的关联;
右下图:各细胞系。IMPORTANT
可以看到,KRAS G12S突变显著富集在细胞系A549(KRAS G12S)中。因此,可以验证突变检出的准确性。
结束语
在本文中,我们一起探讨了单细胞上的突变分析,并展示了一套分析流程作为示例供您参考,该流程通过整合和优化各种生物信息学工具和数据库,可以较好地检测到突变,完成在单细胞数据上的联合分析,能够有效的帮助您获得单细胞转录组在突变维度上的特征,并识别出潜在的功能性变异及其可能的影响。
